Synthetic Par polarity는 unpolarized mammalian cells에서 cytoskeleton asymmetry를 유도합니다.
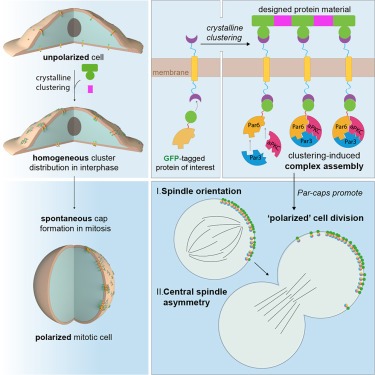
Abstract
Polarized cells은 polarized cytoskeleton에 의존하여 기능합니다. 그러나 cortical polarity cues가 어떻게 cytoskeleton polarization을 유도하는지는 여전히 밝혀지지 않았습니다.
여기서 우리는 최근에 확립된 2D protein arrays를 활용하여 다양한 세포 유형에서 mitosis 중에 관심 있는 거의 모든 단백질의 cortical polarity를 ectopically하게 engineering했습니다. 이를 통해 polarity signaling을 직접 조작하고 cytoskeleton polarization에 충분한 cortical cues를 식별할 수 있습니다. 이 분석법을 사용하여 asymmetric cell division 중 cytoskeleton polarity의 key regulator인 Par complex pathway의 logic을 해부했습니다. 우리는 모든 Par complex subunit의 cortical clustering이 complex assembly를 촉발하기에 충분하며 complex assembly에 대한 primary kinetic barrier는 Par6 autoinhibition의 완화라는 것을 보여주었습니다. 또한 cortical Par complex polarity를 유도하면 unpolarized mammalian cells에서 asymmetric cell division의 두 가지 특징, 즉 Par3를 통해 발생하는 spindle orientation과 aPKC activity에 따른 central spindle asymmetry가 유도된다는 사실을 발견했습니다.
Figure
designed protein arrays를 사용하여 외부로부터 cell polarity 유도
- 설계된 protein arrays를 활용하여 polarity가 없는 NIH/3T3 fibroblasts에서 유도 가능한 cortical polarity를 재구성하고자 함.
Figure 1. protein design을 이용한 cortex의 Artificial symmetry breaking
(A) 실험 원리: GBP-TM-GBP와 GFP-fused target을 안정적으로 co-expressing하는 3T3 세포를 B(c)GFP로 배양한 다음 A(d)로 배양하여 빠른 clustering을 유도.
(B) 왼쪽: (A)방식으로 처리된 세포를 SDCM으로 이미지화.
오른쪽: 세포에서 시간 경과에 따른 세포당 클러스터의 mean ± SEM 수.
(C) 왼쪽: protein clusters가 interphase인 cell cortex에 균일하게 분포되어 있음.
오른쪽: (B)방식으로 처리된 세포를 클러스터링한 후 10분 후에 SDCM으로 이미지화
(D) Array assembly는 GFP-LGN 및 GBP-TM-GBP를 안정적으로 발현하는 3T3 세포의 표면에서 triggered된 다음, 세포를 SDCM 이미징 전에 nocodazole로 12시간 이상 mitosis를 멈춤.
(E) GFP-aPKC 및 GBP-TM-GBP를 안정적으로 발현하는 3T3 세포의 표면에 Arrays를 조립하고 oblique plane light sheet microscopy로 세포를 이미징한 후 trypsin으로 cell rounding을 유도.
[Figure 1A] A(d)와 B(c)-GFP의 두 가지 구성 요소로 구성되며, 여기서 B(c)-GFP는 external anti-GFP nanobody를 통해 transmembrane segment (TM)에 결합하고, A(d)는 B(c) component를 hexagonal array로 클러스터링.
[Figure 1B,C] 이 방법을 사용한 클러스터링은 빠르고(∼20초), 효율적이며(diffraction-limited spot당 ∼80개의 표적), interphase의 cell surface에서 동시에 균일하게 일어남.
[Figure 1D] 긴 incubation periods 동안 mitosis 중에 세포가 모이면 이러한 클러스터가 합쳐져 cortical caps을 형성한다는 사실을 발견.
[Figure 1E] trypsin을 사용하여 세포를 인위적으로 둥글게 만들면 interphase에서 cap formation이 일어나며, 이 현상은 몇 분 내에 발생. 이는 mitosis보다는 spherical geometry와 관련이 있는 것으로 보임.
- cap formation과 spherical shape 사이의 상관관계는 우리의 polymer가 세포 주기 단계에 관계없이 모든 원형 세포의 표면에서 cap을 유도할 수 있음을 시사.
Par complex subunit의 클러스터링은 slow complex assembly를 유도.
- protein array를 활용하여 Par complex assembly 경로를 조사.
Figure 2. 클러스터링에 의해 유도된 core Par complex의 Sequential assembly.
(A) 실험 원리: GFP-fused Par6A 또는 그 mutants를 3T3 세포에 클러스터링하고, 이후 Par complex assembly를 모니터링.
(B) immunofluorescence로 모니터링한 Par6A 클러스터링에 따른 endogenous aPKC 및 Par3의 Recruitment.
(C) 시간에 따른 표시된 GFP-Par6A 구조와 endogenous aPKC 클러스터 간의 colocalization과 GFP-Par6A, endogenous aPKC 및 endogenous Par3 간의 triple colocalization의 mean ± SEM 백분율.
(D) 위와 같이 모니터링한 endogenous aPKC 및 Par3의 GFP-Par6AΔaPKC 클러스터로의 Recruitment.
[Figure 2A-C] 기준 조건(클러스터링 후 2분)에서는 Par6A, aPKC, Par3 사이의 triple colocalization이 낮았지만, 인위적으로 Par6A를 클러스터링하면 60분 동안 Par complex assembly를 유도하기에 충분. 이는 Par complex assembly가 느린 과정이라는 것을 보여줌.
[Figure 2A-C] Par 생물학에서 일반적으로 보고된 특징인 클러스터링이 그 자체로 core Par complex의 assembly를 주도하며, core subunit의 클러스터링이 유사한 kinetics로 Par complex assembly를 유도한다는 것을 입증.
[Figure 2D] aPKC에 결합할 수 없는 established Par6 point mutant인 Clustered Par6ΔaPKC26는 aPKC 또는 Par3를 recruit할 수 없음.
- 이러한 결과는 Par6-aPKC 상호 작용이 Par3 recruit와 full Par complex의 assembly에 필요하다는 것을 보여줌.
Relieving Par6 autoinhibition은 Par complex assembly의 rate-limiting step.
- 포유류의 Par complex assembly에 aPKC의 kinase activity가 필수 불가결
- slow assembly rate가 Par6 autoinhibition에 의한 것이라는 대안 가설을 제시
- Par6 autoinhibition이 slow assembly kinetics의 근본적인 이유라면, autoinhibition에 bias된 Par6 mutant는 Par complex assembly를 더욱 느리게 할 것이라고 추론
Figure 3. Par complex assembly의 rate-limiting step인 Relieving Par6 autoinhibition.
(A) 이 연구에 사용된 Par6A fragments.
(B) GFP-Par6A 또는 Par6A mutant clusters와 aPKC(왼쪽) 또는 Par3(오른쪽) 간의 mean ±SEM colocalization 비율.
(C) Par6ANter는 Par6ACter와 binds.
(D) GFP-fused Par6A 또는 Jo-Par6-In을 3T3 세포에 클러스터링하고 endogenous aPKC 및 Par3의 시간에 따른 recruitment.
(E) clustered subunit의 function으로써 core Par complex의 assembly에 선호되는 경로.
[Figure 3A,B] Par6A Nter(aPKC-binding site보유)는 aPKC를 recruit할 수 있었지만 Par3는 recrui할 수 없었고, clustering Par6A Cter(Par3-binding site보유)는 Par3를 recruit할 수 있었지만 aPKC는 recruit할 수 없었음.
[Figure 3B] Par 6 Nter에 의한 aPKC recruitment는 Par6 full-length보다 더 빨랐는데, 이는 aPKC recruitment가 느린 것이 Par6 autoinhibition을 완화할 필요가 있기 때문일 것으로 예상되는 결과임.
[Figure 3B] Par6 Cter는 Par3를 recruit할 수 있는 반면, full-length의 Par6 ΔaPKC mutant는 recruit할 수 없었음
- 이는 full-length의 Par6가 실제로 autoinhibited state에 존재하며, 이는 aPKC 결합에 의해 완화될 수 있음을 시사.
[Figure 3C] Par6의 two halves사이의 interaction은 mitochondria-targeted Par6 Nter가 cytosolic GFP-Par6 Cter를 mitochondria또는 vice versa로 강력하게 recruit하는 relocalization 실험을 통해 확인.
[Figure 3D] Par complex assembly은 Par6에 비해 Jo-Par6-In에서 두 배 더 오래 걸렸음. Jo-Par6-In은 WT에 비해 상대적으로 normal kinetics로 aPKC를 recruit하여, aPKC와 Par3 사이의 직접적인 상호 작용만으로는 Par3를 Par6에 recruit하기에 충분하지 않음을 다시 확인.
[Figure 3E] Par complex assembly에 대한 패러다임을 제안
(1) core subunits의 클러스터링은 complex assembly를 촉발할 수 있음.
(2) assembly의 rate-limiting step은 autoinhibited Par6의 opening임.
(3) aPKC의 kinase activity는 초기 Par complex assembly에 영향을 주지 않음.
(4) Par6가 있을 때 aPKC와 Par3 사이의 direct binding은 Par complex assembly에 필요하거나 충분하지 않음.
Par complex의 asymmetric cortex는 spindle orientation을 유도하기에 충분.
- Par complex caps이 포유류에서 asymmetric cell division의 주요 과정을 주도하기에 충분한지 조사
- spindle orientation 결정에 필요한 것으로 알려진 다른 단백질의 asymmetric cap이 포유류 세포에서도 충분한지 조사.
Figure 4. An asymmetric Par complex cortex는 mitotic spindle의 방향을 결정하기에 충분.
(A-G) 왼쪽: Jupiter -iRFP670, GBP-TM-GBP를 안정적으로 공동 발현하는 3T3 세포에 어레이를 조립하고 GFP-fusions을 표시. 세포를 12시간 이상 mitosis에 정지시킨 다음 nocodazole block을 방출하자마자 SDCM으로 이미지화.
오른쪽: 표시된 조건에서 division plane과 cap 사이의 각도.
(H) cap size의 function으로서 division plane과 cap 사이의 각도.
(I) 단순화된 spindle orientation pathway.
(J) Par6A cap을 갖는 Dividing cells들은 LGN이 없어진 경우 (또는 control siRNA로 처리된 경우) 위와 같이 처리되고 이미징되어, cap과 division plane 간의 각도를 결정하기 위해 사용.
(K) (J)에서와 같이 처리한 세포의 division Orientation.
[Figure 4A,H] GFP의 asymmetric cap이 division orientation에 영향을 미치지 않았으며, division angle은 cap의 위치나 크기와 상관관계가 없음.
- 이는 synthetic cap이 그 자체로 division orientation 에 영향을 미치지 않는다는 것을 보여줍니다
[Figure 4B-D] cap에 어떤 Par complex subunit을 표적으로 삼으면 oriented division이 견고해짐.
[Figure 4D,E] aPKCdead cap은 spindle orientation을 유도하는 데 WT와 마찬가지로 강력했으며 이는 aPKC의 kinase activity가 spindle orientation에 불필요함을 나타냄.
[Figure 4B-E] cap에 perpendicular spindle 들의 preferential orientation으로 인해, Par complex caps는 asymmetric cell division의 또 다른 특징, 즉 단 하나의 daughter cell에 의한 polar cap의 preferential inheritance를 유도하기에 충분.
- 이러한 결과는 Par complex의 asymmetric cortex가 unpolarized mammalian cells에서 spindle orientation을 유도하기에 충분하다는 것을 입증.
[Figure 4I] Inscutable (mInsc)과 LGN(Gpsm2라고도 함)은 Par3의 downstream에 작용하여 dynein을 통해 astral microtubules을 당겨 mitotic spindle의 방향을 잡는 것으로 잘 알려져 있음.
[Figure 4F,G] mInsc와 LGN의 cap이 spindle을 견고하게 배향하여 polar cap의 asymmetric inheritance를 유도한다는 것을 발견.
[Figure 4J,K] LGN depletion이 spindle orientation을 유도하는 Par6 cap의 능력을 크게 손상시킴에 따라 Par complex-induced spindle orientation이 실제로 LGN을 통해 발생.
[Figure 4H] cap에 Par complex components 또는 mInsc/LGN을 targeting했을 때 cap size와 spindle orientation의 견고성 사이에는 뚜렷한 상관관계가 발견되지 않음.
- 이는 cap의 크기보다는 cap의 분자 구성이 spindle orientation을 결정하는 요소임을 시사.
aPKC와 Par3의 asymmetric cortex는 central spindle asymmetry를 유도하기에 충분.
- cap이 asymmetric cell division의 다른 측면을 재구성하기에 충분한 지 조사
- Par-controlled phenomenon이 포유류에서 conserved되는지, 그리고 division assay에서 이를 재구성할 수 있는지 궁금
Figure 5. Asymmetric cortical aPKC가 central spindle symmetry breaking을 제어.
(A) anterior pIIb cell (core Par complex cap을 inheriting 하지 않은 세포)의 마커로 mRFP-Pon을 발현하는 SOP 세포를 고정하고 acetylated tubulin에 대한 immunostaining.
(B,C) GBP-TM-GBP를 안정적으로 발현하는 3T3 세포에 어레이를 조립하고 mitotic shake-off 12시간 전에 construct를 표시한 다음 acetyl-tubulin으로 염색.
(D-F) 표시된 샘플에서 central spindle을 통한 Acetyl-tubulin intensity pseudo-linescan (mean ± SEM)
(G) cap에 targeted된 단백질의 function으로 central spindle의 anterior side (no cap) 에서의 microtubule density의 Normalized enrichment를 측정.
(H) 요약: synthetic Par-complex cap은 central spindle symmetry breaking을 촉진하기에 충분하며, Par-cap side의 microtubule densities는 낮고 cap 반대쪽의 microtubule densities는 높음.
[Figure 5A] Drosophila sensory organ precursors (SOP)의 asymmetric division에서 central spindle로 알려진 anaphase-specific spindle도 symmetry breaking이 일어나면서 central spindle의 앞쪽이 뒤쪽보다 더 많은 microtubules을 포함
[Figure 5A] 이 asymmetric central spindle은 Notch와 그 ligand Delta를 포함하는 signaling endosomes이 하나의 daughter cell로만 polarized trafficking하도록 조율하여 asymmetric cell fate determination에 기여.
[Figure 5B,E] unpolarized 3T3 cells에서 aPKC의 asymmetric cap을 재구성한 결과, central spindle은 SOP에서와 같이 asymmetric하게 되었음. 중요한 것은 species사이에서도 orientation이 conserved 되었다는 점임.
[Figure 5A,B,D,E,F,G,H] aPKC를 포함하는 Par complex cap을 물려받은 쪽이 더 lower microtubule density를 나타냄.
[Figure 5G] central spindle asymmetry는 다른 core Par complex component Par3의 polar targeting에서도 발생했지만, Par6에서는 발생하지 않았음.
[Figure 5G] GFP-Par6 cap이 없는 쪽에서 microtubule enrichment에 대한 약간의 bias가 관찰되었지만, 이는 GFP 대조군과 비교했을 때 유의하지 않았음. 중요한 것은 mitotic cells를 선택해 mitotic shake-off를 사용하거나 nocodazole을 사용해 mitosis를 멈추게 했을 때 유사한 asymmetry levels이 관찰되었음.
- 이는 metaphase에 소요되는 시간이 central spindle symmetry breaking process와 관련이 없음을 시사.
- aPKC와 Par3의 asymmetric cap을 재구성하는 것만으로도 unpolarized mammalian cells에서 central spindle symmetry breaking을 유도하기에 충분하다는 것을 알 수 있음.
[Figure 5C,F,G] mInsc와 LGN의 polar targeting은 확립된 polarity axis를 따라 spindle을 강력하게 oriented했지만(Figure 4F,G), asymmetric central spindle의 형성을 촉진하지는 않았음.
- 이러한 결과를 종합하면 central spindle asymmetry와 spindle orientation은 untangled Par complex activity의 두 가지 discrete outputs이라는 것을 알 수 있음.
[Figure 5G] central spindle asymmetry의 key effector가 aPKC임을 가리키며, 이는 central spindle symmetry breaking을 유도하는 데 가장 강력한 aPKC cap과 일치.
Asymmetric cortical aPKC activity는 central spindle asymmetry를 유도.
- aPKC가 프로세스를 시작하는 Par complex subunit아라는 가설 제시.
Figure 6. Asymmetric aPKC kinase activity는 central spindle symmetry breaking을 촉진.
(A) GBP-TM-GBP 및 GFP-aPKC를 안정적으로 발현하는 3T3 세포 또는 그 kinase dead variant를 mitotic shake-off 12시간 전에 array formation을 위해 처리한 후 acetyl-tubulin으로 염색.
(B) (A)처럼 처리한 세포의 central spindle을 통한 Acetyl-tubulin intensity pseudo-linescan (mean ± SEM).
(C) (A)처럼 처리된 세포에서 GFP-aPKC cap 반대쪽의 microtubule density의 Normalized enrichment를 측정.
(D) GBP-TM-GBP를 안정적으로 발현하고 GFP 융합을 나타내는 3T3 세포를 (A)처럼 처리한 후 cap 반대쪽의 microtubule density의 Normalized enrichment를 측정.
(E) 요약: Par3는 recruit하지만 aPKC는 recruit하지 않는 Par6A Cter cap은 aPKC가 없기 때문에 asymmetric central spindle을 형성하지 않습니다. 반대로, Par6A Nter cap은 Par6A full-length와 달리 central spindle asymmetry를 유도하며, 이는 aPKC의 kinase activity를 억제하는 Par6 Cter의 기능과 일치.
[Figure 6A-C] aPKC dead는 Figure 4F에서 Par complex assembly와 spindle orientation을 유도하는 데는 성공했지만 중앙 스핀들 대칭 파괴를 유도하는 데는 실패.
- 이는 central spindle symmetry breaking이 aPKC의 kinase activity의 asymmetric cortex에 의해 제어된다는 것을 입증.
- [Figure 6D,E] Par3는 recruit하지만 aPKC는 recruit하지 않는 Par6 Cter 클러스터는 asymmetric central spindles의 형성을 촉진하지 않았으며, 이는 aPKC를 효율적으로 recruit하는 full-length Par6와 유사.
[Figure 6D,E] aPKC는 recruit하지만 Par3는 recruit하지 않고, aPKC-inhibitory C-terminal domain이 결여된 Par6 Nter 클러스터링은 aPKC 클러스터링과 유사하게 asymmetric central spindles formation을 유도.
[Figure 6D] Par complex assembly에 영향을 미치지 않는 aPKC의 kinase-hyperactive mutant를 클러스터링한 결과, wild-type aPKC에 비해 central spindle asymmetry level이 훨씬 더 높았음.
[Figure 6E] central spindle asymmetry는 aPKC의 kinase activity에 의존하며, 이는 Par6의 C-terminal domain에 의해 억제될 수 있음.
Central spindle symmetry breaking은 CAMSAP3, Kif2A, Kif2B 및 Kif2C에 의해 제어
- aPKC의 central spindle asymmetry downstream의 분자 메커니즘을 규명하기 시작
- central spindle symmetry breaking에서 Camsap과 Kinesins-13의 최종적인 역할을 조사
Figure 7. central spindle symmetry breaking을 촉진하는 Camsap3, Kif2A, Kif2B 및 Kif2C
(A,B) acetyl-tubulin and Camsap2(A) 또는 Kif2A(B)에 대해 immunostained된 synthetic GFP-aPKC cap을 사용하여 3T3 세포를 분할.
(C,D) (A) 및 (B)에서와 같이 처리된 세포의 central spindle을 통한 Pseudo-linescan (mean ± SEM).
(E,F) GBP-TM-GBP 및 GFP-aPKC를 안정적으로 발현하는 3T3 세포를 mitotic shake-off 12시간 전에 array formation 전에 표시된 siRNA로 처리하고 acetyl-tubulin으로 염색. 그런 다음 aPKC cap 반대쪽의 microtubule density의 normalized enrichment를 측정.
(G) Figure 4, 5, 6,7 요약.
왼쪽: GFP-aPKC, GFP-Par6A, GFP-Par3, GFP-Insc 및 GFP-LGN의 cap은 de novo 설정된 polarity axis를 따라 spindle을 배향하기에 충분. Spindle orientation에는 LGN이 필요.
오른쪽: aPKC와 Par3 cap을 가진 세포가 cell division을 진행할 때 asymmetric central spindle을 형성하며, 이는 aPKC의 kinase activity에 따라 달라짐. Central spindle asymmetry에는 Camsap3, Kif2A, Kif2B 및 Kif2C가 필요하며, 따라서 aPKC 표적이 될 수 있음.
[Figure 7A,B] 먼저 aPKC cap에 의해 유도된 asymmetric central spindles에서 Camsap2와 Kif2A의 subcellular localization을 조사.
[Figure 7A,C] Camsap2는 microtubule densities와 일치하는 asymmetric distribution을 보였으며, cap이 없는 쪽의 밀도가 더 높고, aPKC cap이 있는 쪽의 밀도가 더 낮음.
[Figure 7B,D] 반면에 Kif2A도 asymmetric localization을 보였지만, 이 경우에는 cap이 없는 쪽의 밀도가 더 낮고 다른 쪽의 밀도가 더 높아 microtubule densities의 asymmetry와 반대되는 양상을 보임.
[Figure 7E] 중요한 것은 Camsap1 및 Camsap2 depletion은 central spindle asymmetry에 큰 영향을 미치지 않았지만, Camsap3 depletion은 symmetric central spindles formation을 초래.
[Figure 7F] 반면, 모든 Kinesins -13 homologs Kif2A, Kif2B 또는 Kif2C/MCAK의 depletion은 central spindle asymmetry를 막음.
- 이러한 데이터를 종합해 볼 때, Par complex에 의한 central spindle symmetry breaking의 molecular mechanism은 파리에서 포유류에 이르기까지 conserved되어 있으며, Camsap3와 Kif2A/Kif2B/MCAK는 각각 Patronin과 Klp10A의 ancestral functio을 물려받았음을 시사.
Disscussion
극성 경로의 입력/출력 로직에 대한 합성 해부
우리의 연구는 이전 연구와 비교하여 피질 극성을 재구성하는 직교 방법을 제공합니다 .11,12,13,14,15,17 구체적으로, 우리의 방법은 이전에 동일한 방법에서 동시에 발견되지 않은 5 가지 특성, 즉 (1) 유도 가능, (2) 빠름, (3) 높은 처리량, (4) 분열 중 단일 세포 수준에서 작동, (5) cap의 분자 구성 제어 가능을 결합합니다. 이러한 핵심 조합을 통해 cap에 조립된 극성 단백질의 하류 효과(즉, 경로 출력)를 해부할 수 있을 뿐만 아니라 피질에서 극성 신호 네트워크의 조립(즉, 입력)을 연구할 수도 있습니다. 우리는 이 새로운 방법을 활용하여 파 복합체의 분자 조립 경로를 체계적이고 정량적으로 해부할 수 있었고(그림 2와 3), 결과적으로 사전 조립된 파 복합체 cap이 세포 골격 대칭성 파괴에 미치는 영향을 연구하고 관련된 분자 메커니즘을 확인할 수 있었습니다(그림 4, 5, 6, 7).
파 복합체 조립을 위한 통합 프레임워크
수백만 개의 어레이에서 수집한 데이터를 통합하여 파 복합체 경로의 조립 순서를 새롭게 조명하고 이 프로세스에 대한 통합 패러다임을 제안할 수 있었습니다(그림 3G). 중요한 것은 우리의 결과가 이전 연구와 매우 일치한다는 점입니다. 특히 다음과 같은 사실을 확인했습니다: (1) Par6와 aPKC는 상호 작용할 수 있으며, 이 상호 작용에 필수적인 aPKCE85/R91과 Par6K19가 존재하고,26 (2) Par6와 Par3는 상호 작용할 수 있으며, 이 상호 작용에 필수적인 Par3G600,602와 Par6121-257이 존재하고,43 (3) Par 복합체 조립에는 Par3의 클러스터링/올리고머화가 필요하며,30 (4) 세포-세포 접합부에서 Par 복합체가 클러스터로 조립할 수 있으며,44 (5) Par3의 aPKC 인산화에도 불구하고 Par 복합체 조립이 발생할 수 있음이 밝혀졌습니다.31
우리의 연구는 이러한 이전 발견을 확장하고 Par 복합체 생물학에 대한 새로운 통찰력을 제공합니다. 특히 다음과 같은 사실을 발견했습니다: (1) 핵심 Par 복합체 구성 요소의 클러스터링은 삼자 복합체의 조립을 유도할 수 있고(그림 2, S5 및 S6), (2) Par 복합체 조립에는 시간이 걸리며, 이는 아마도 Par6 자가 억제 때문일 수 있으며(그림 3 및 S7), (3) Par 복합체 조립에 aPKC 키나제 활성은 필수적이며(그림 S5), (4) aPKC와 Par3가 직접 상호작용할 수 있다고 하더라도 Par3가 aPKC와 효과적으로 결합하기 위해서는 Par6가 필수적임을 확인했습니다(그림 2, S5 및 S6). 이 마지막 발견은 Par3 결합을 폐지하는 aPKC의 V606A 돌연변이가 파리의 내인성 Par3 cap에 recruit되지 않는 것으로 밝혀진 사실과 다소 상충되지만,31 이는 당사의 결과에 비추어 볼 때 이러한 세포에서 Par6가 제한적이기 때문으로 설명할 수 있습니다(자세한 내용은 STAR 방법의 “Par 복합체 조립 경로의 근거” 섹션 참조).
파 복합체 극성은 세포 골격 대칭성 파괴에 두 가지 개별적인 영향을 미칩니다.
이 연구의 주요 발견은 유사 분열 중에 Par 복합체의 비대칭 피질이 두 개의 개별적이고 독립적인 출력을 가지며, 정상적으로 분극되지 않은 세포의 “분극된” 세포 분열을 촉진한다는 것입니다(그림 7G). 한편으로, 모든 Par 복합체 서브유닛(Par3, Par6A 또는 aPKC)의 극성 cap과 다운스트림 이펙터인 mInsc 및 LGN을 유도하는 것만으로도 극성 축을 따라 스핀들 방향을 유도할 수 있습니다(그림 7G, 왼쪽 패널). 반면, mInsc 또는 LGN이 아닌 Par3 및 aPKC의 cap을 유도하면 중앙 스핀들 대칭 파괴가 유도됩니다(그림 7G, 오른쪽 패널). 중요한 점은 Par 매개 스핀들 오리엔테이션에는 LGN이 필요하지만(그림 4J), 중앙 스핀들 비대칭에는 필요하지 않다는 점입니다(그림 S9M). 반대로, 중앙 스핀들 대칭성 파괴는 스핀들 배향(그림 4E)에는 필요 없는 aPKC의 키나아제 활성에 의존합니다(그림 6). 즉, 스핀들 배향과 중심 스핀들 비대칭은 Par 복합체의 하류에서 발생하지만, 풀릴 수 있는 두 가지 다른 분자 캐스케이드를 통해 발생합니다. 스핀들 배향은 Par3/mInsc/LGN을 통해 발생하고, 중심 스핀들 비대칭은 aPKC의 키나제 활성을 통해 발생합니다. 또한, 플라이 SOP의 중심 스핀들 비대칭에는 플라이 Par3(바주카포)가 필요하다는 것을 발견했습니다(그림 S9A-S9C). SOP 세포는 mInsc가 부족하여 Par 복합체를 통해 스핀들을 배향하지 않기 때문에,45 스핀들 배향과 중심 스핀들 비대칭은 파리의 생체 내에서도 풀릴 수 있습니다. 이는 또한 일부 기능 상실 돌연변이가 파리5,10와 포유류 모두에서 스핀들 방향에 영향을 주지 않고 중심 스핀들 비대칭에 영향을 미치는 것으로 밝혀진 이유를 합리화합니다(그림 4 및 7). 또한 조립 결과에 비추어 볼 때, aPKC가 Par3에 결합하기 위해서는 Par6가 절대적으로 필요하기 때문에 이 두 가지 활동은 Par 복합체 내에서 Par6에 의해 연결된다는 점도 주목할 가치가 있습니다. 따라서 Par6는 Par 복합체의 두 출력 사이의 결합을 조절할 수 있습니다. 이는 Par6 C-말단 도메인이 aPKC의 키나아제 활성을 조절하여19 중앙 스핀들 비대칭성을 조정하는 능력과 일치합니다(그림 6).
우리의 데이터는 또한 중심 스핀들 대칭성 파괴를 설명할 수 있는 패러다임을 제시합니다. 실제로 세포질의 고도로 확산되는 특성으로 인해 세포 내에 미세소관 조절 인자의 구배가 존재할 수 있다고 상상하기는 어렵지만, 중심 스핀들 비대칭이 피질 aPKC 활성에 의해 제어된다는 우리의 발견(그림 6)은 활동 구배의 존재를 암시할 수 있습니다. 실제로, 중심 스핀들 비대칭을 담당하는 미세소관 조절 인자의 활성이 aPKC 매개 인산화에 의해 조절된다면, 비대칭적인 aPKC 피질은 세포질 포스파타제의 존재 하에서 인산화된 조절 인자의 정상 상태 구배를 초래할 수 있습니다. 따라서 확산에도 불구하고 활성 구배가 존재할 수 있습니다. 향후 연구의 흥미로운 질문은 피질과 사이토솔 사이의 정보 흐름을 제어하는 aPKC의 주요 표적을 식별하는 것입니다. 우리의 데이터에 따르면 미세소관 조절인자인 Camsap3, Kif2A, Kif2B, Kif2C/MCAK가 aPKC의 다운스트림 이펙터가 될 수 있습니다(그림 7). Camsap3는 미세소관이 해중합되지 않도록 보호하기 때문에,36,46 직접 또는 간접적으로 aPKC의 키나아제 활성에 의해 부정적으로 조절될 수 있으며, 결과적으로 Camsap3는 cap 반대쪽에서 더 활성화되어 미세소관 밀도가 더 높아질 수 있습니다(그림 7G, 오른쪽 패널). 반대로, 미세소관을 불안정하게 만드는 키네신인 Kif2A, Kif2B 및 Kif2C/MCAK는 aPKC에 의해 긍정적으로 조절되어 cap 쪽의 활성이 증가하여 중앙 스핀들의 이쪽에서 미세소관 밀도가 낮아질 수 있습니다(그림 7G, 오른쪽 패널).
결론적으로, 이 연구는 합성 생물학과 단백질 설계가 세포 생물학 문제를 직교적인 방식으로 해결할 수 있는 힘을 강조합니다. 특히 중심 스핀들 비대칭이 비대칭 분열의 보존된 특징임을 입증할 수 있었으며, 우리의 방법을 통해 다양한 종에서 내재적 세포 극성과 세포 골격 대칭성 파괴를 지배하는 분자 메커니즘을 더 자세히 설명할 수 있을 것으로 기대합니다. 특히, 파 복합체 cap의 재구성이 세포 운명 결정 인자의 비대칭 분리,1 비대칭 유전자 발현 또는 엔도솜,47 미토콘드리아,8 및/또는 리소좀의 극성 이동과 같은 섬유아세포에서 비대칭 세포 분열의 다른 측면을 유도하기에 충분한지 알아보는 것은 흥미로울 것입니다.7