RNA 매개 symmetry breaking이 단일 후각 수용체 선택을 가능하게 한다
Abstract
후각 수용체(Olfactory Receptor, OR) 선택은 모든 후각 뉴런이 약 2,000개 이상의 OR 대립유전자 중 하나를 안정적으로 transcription하는 transcriptional dominance를 위한 대립유전자 경쟁의 극단적인 예시다. OR 유전자 선택은 단일 OR에서 전사를 활성화하는 다중 염색체 enhancer hub에 의해 매개되고, 뒤따르는 OR translation-dependent feedback에 의해 안정화된다. 여기에서는 single cell genomics를 사용하여 다양한 enhancer 구성을 가진 많은 경쟁 hub가 형성되며, 그 중 하나만 euchromatic 특징과 transcription 능력을 유지한다는 것을 보여준다. 또한, 우리는 OR transcription이 enhancers를 모집하고 enhancer hub 활동을 국소적으로 강화하는 반면, OR RNA는 원거리에서 경쟁하는 OR의 transcription을 억제하여 transcriptional singularity로의 전환을 촉진한다는 증거를 제시한다. OR transcription은 동등한 enhancer hub 간의 대칭을 깨기에 충분하지만, OR translation은 우세한 hub에서 transcription을 안정화 했으며, 이는 transcriptional prevalence를 위해 OR 대립유전자에 의해 구현되는 순차적 non-coding 및 coding 메커니즘이 있을 수 있음을 나타낸다. 우리는 coding OR mRNA가 핵 구조에 영향을 미치고 자신의 transcription을 강화하며, 경쟁자의 transcription을 억제하는 non-coding 기능을 가지고 있으며, 이는 확률론적 세포 운명 결정에 일반화할 수 있는 함의를 가진다고 제안한다.
Figures
GI accessibility changes with neuronal differentiation and genomic compartmentalization
– 신경계 계통 세포에서 pseudotime에 따라 정렬했을 때 *GI의 accessibility는 감소하지만 OR 자체의 발현은 계속 증가함
– 이는 신경계 계통 세포 발달 과정에서 대부분의 GI 및 OR 관련 promoter 불활성화가 이뤄지며, 강력한 단일 OR 전사로 전환됨을 의미
Fig. 1. single nucleus ATAC와 single nucleus RNA-seq를 결합하면 점진적인 GI 비활성화가 밝혀짐.
(A) 한 마우스에서 양질의 검증된 6,497개 세포의 RNA 및 ATAC 데이터에서 weighted nearest neighbours analysis를 통해 구축한 마우스 MOE multiome의 UMAP. 신경계 계통에는 GBCs, INPs, iOSNs, mOSNs이 포함.
(B) 세포를 pseudotime에 따라 색으로 표시한 신경 계통의 UMAP projection.
(C) 알려진 마커를 사용한 pseudotime projection의 검증.
(D) 가장 가까운 pseudotime으로 반올림된 모든 세포의 평균 누적 GI accessibility dynamics. 초기 (GBC–INP3), 중간 (INP3–iOSN), 후기 (mOSN).
(E) pseudotime에 따른 개별 GI accessibility.
(F) pseudotime에 따른 누적 GI (검정색) 및 OR 프로모터 (주황색) accessibility
(G) 각 세포별 GI accessibility 와 가장 높게 표현된 OR의 발현 수준
(H) 각 세포별 GI accessibility 와 활성 GI당 접근성
(I) 각 세포별 GI accessibility 와 상위 mOSN cCREs accessibility
(J) 각 세포별 GI accessibility 와 mOSNs에서 LHX2/EBF1 cCREs의 누적 accessibility
(K) mOSNs에서 mOSN cCREs, GIs (3.64% ± 3.40%, n = 63), OR 프로모터의 접근 가능한 세포 비율을 비교하는 박스 플롯.
(L) 1,010 mOSNs에서 509,545개의 고유한 세포 쌍이 접근 가능한 GIs에 대해 테스트.
(M) 0에서 12개의 GIs를 공유하는 세포 쌍을 구성하는 모든 고유 세포 간의 OR의 평균 빈도. 점선 빨간색 선은 고유한 OR의 수가 고유한 세포의 수와 같아질 경우 예상되는 평균 OR 빈도를 나타냄.
(N) 0에서 12개의 GIs를 공유하는 세포 쌍을 구성하는 모든 고유 세포에 의해 표현된 각 OR의 빈도. 각 색은 서로 다른 OR을 의미.
[Fig 1A] 신경 계통의 세포인 globose basal cells (GBCs), immediate neuronal precursors (INPs), immature OSNs (iOSNs), mOSNs19 등을 식별
[Fig 1B, C] 여러 신경 계통의 세포들을 pseudotime에 따라 나열한 후 알려진 마커로 진화 단계를 나눔.
[Fig 1D, E] Pseudotime에 따른 GI accessibility의 변화를 식별. 중기 (pseudotime = 5)에 평균적인 accessibility가 최고로 증가한 후 지속적으로 감소.
[Fig 1G-K] OR promoter의 accessibility는 GI처럼 감소하지만 OR expression은 지속적으로 증가하며, GI가 아닌 다른 cis regulator의 경우 이러한 accessibility 감소가 없음.
[Fig 1I] 분화된 신경 계통 세포에서 GI 조합이 동일한 경우는 매우 드물며, 특정 OR의 발현을 유도하는 고유한 GI 조합을 가지게 됨.
[Fig 1M, N] GI accessibility의 변화는 특정 OR 종류와 관계 없음.
Dip-C shows distinct features between active and inactive GI hubs
– Dip-C 기법을 통해 active 및 inactive GI hub를 식별하고 OR gene과의 접촉 특이성을 분석함.
– Active GIH의 접촉 특이성이 inactive GIH 대비 높고, 선택된 OR gene이 불활성화된 OR gene에 비해 GI와의 접촉 특이성이 높음.
Fig. 2. Dip-C가 활성화 및 비활성화 GI hubs 사이의 차이점을 발굴함.
(A) Dip-C(Chromosome Conformation Capture 기법을 이용한 3D 구조 분석)는 Mor28iGFP 및 gg8-tTA>tetOP-2iGFP 마우스로부터 분리한 161개의 GFP 양성 핵에 대해 수행됨. 개별 핵의 3차원 모델은 haplotype-imputed contact maps를 사용하여 제작되었고, 염색체별로 색이 구분됨. 활성 OR allele(큰 구)과 GI(중간 크기 구)를 제외한 모든 유전자가 제거된 대표적인 Mor28iGFP 핵이 표시되었으며, 활성 Mor28 allele은 GI 클러스터 근처에 위치해 활성 hub를 표시함. 추가적인 GI 클러스터도 감지됨.
(B) 각 세포(행)에 대해 이진 배열이 생성되어 P2 allele(비활성 Cas, chr7)이 활성화될 때 2.5 p.r. 미만의 GI를 나타낸 그림. 활성 상태(상단, gg8-tTA>tetOP-2iGFP Dip-C) 및 비활성 상태(하단, OMPiGFP) 모두에 대해 적용
(C) P2+ 핵에서 GI의 공간적 관계에 대한 hierarchical clustering. 활성 GI hub는 녹색, 가장 비슷한 크기의 비활성 hub는 검정색으로 표시됨.
(D) 지정된 성숙의 지표 마커로 염색된, 지정된 시간점에서의 신경 세포의 대표 이미지.
(E-H) 활성 hub 내 GIs 사이, 비활성 hub 내 GIs 사이, 활성 OR과 GIs 사이, 그리고 비활성 hub 내에서 최대로 관여하는 비활성 OR과 GIs 사이에서 이루어진 염색체 간 접촉에 대한 Heatmap.
[Fig 2A] 각 신경세포의 핵을 3차원으로 modeling한 결과. 염색체 OR 구획 및 활성 Mor28 대립유전자와 연관된 GI hub를 보여줌.
[Fig 2B] 혼합된 mOSN 세포와 비교했을 때 순수 P2 + 및 M or28 + OSN에서 두 OR allele의 근거리에서 GI의 연관성이 확인됨.
[Fig 2C, D] GI hub를 활성화 여부에 따라 active 및 inactive로 정의해 식별.
[Fig 2E-H] active GIH에서 inactive 대비 더 높은 접촉 특이성을 가지며, 활성 OR과 경쟁할 수 있을만한 비활성 OR이라 해도 GI와의 접촉 특이성이 활성 OR에 비해 두 배 가까이 낮음.
Active and inactive hubs possess distinct biochemical properties and histone modification features
– Liquid Hi-C를 통해 염색체를 조각내가며 활성 hub 및 비활성 hub의 접촉 특이성 변화를 관찰함.
– 활성 GI hub의 접촉은 digestion이 진행될 때 접촉 특이성이 더욱 큰 폭으로 감소하며 H3K27ac에 대한 접촉 특이성이 강화되는 등 비활성 GI hub 와 구분되는 뚜렷한 생화학적 특징을 가짐.
Fig. 3. 활성화 및 비활성화 GI hubs가 서로 다른 chromatin 특성을 보유함
(A) gg8-tTA>tetOP-2iGFP GFP 양성 분류된 세포에 대해 0분(대조군), 5분, 30분, 60분 동안 Liquid Hi-C가 수행되었으며, 비활성 hub(inactive ORs to GIs, 하단)와 활성 hub(active OR to GIs, 상단)의 차별적 안정성을 측정한 결과. 모든 히트맵은 hub를 중심으로 한 1Mb 반경 내의 염색체 간 상호작용을 50kb 해상도로 나타냄. 히트맵은 각 시간대별로 생성된 세 개의 생물학적 반복 실험으로부터 병합된 데이터.
(B) CSS(접촉 특이성 점수)의 평균 변화율이 DpnII 사전 처리 과정에서 시간에 따라 정량화됨: 5분(active OR, − 28.9 ± 19.4%, inactive OR − 14.9 ± 1.06%), 30분(active OR, −52.5 ± 15.3%, inactive OR − 30.1% ± 1.97%), 60분(active OR, − 63.2 ± 10.6%, inactive OR − 38.0 ± 2.1%).
(C, D) gg8-tTA>tetOP-2iGFP GFP 양성 분류된 세포의 두 개의 생물학적 반복 실험에서 H3K27ac HiChIP이 수행되었으며, 그 결과가 병합되어 Hi-C와 비교하여 CSS의 차이가 활성 GI hub와 비활성 GI hub에서 50kb 해상도로 평가됨. 삽입된 히트맵은 hub를 중심으로 한 200kb 반경 내의 접촉을 확대하여 보여줌.
[Fig 3A] 활성 OR-G의 접촉은 집중도가 높아 heatmap 중앙에 점의 형태로 표시되지만, 비활성 OR-GI 접촉은 특정 지역에 집중되지 않음.
[Fig 3B] predigestion 5분 이내에 활성화되는 hub는 비활성 hub 대비 접촉 특이성이 두 배 감소한 후 60분에 유의한 차이까지 벌어짐.
[Fig 3C, D] 활성 hub의 접촉은 H3K27ac에 대해 강화되지만 다른 GI-OR 접촉의 특이성은 감소함.
OR transcription facilitates ‘symmetry breaking’ and singular OR gene choice
– 신경 세포 성숙에 따라 특정 OR 유전자의 전사만 강화되고 나머지가 감소하는 현상을 X chromosome의 silencing 메커니즘과 유사한 symmetry breaking에 의해 이뤄짐을 가설로 세움.
– 다유전성 OR 전사 중에도 P2 기반 유도는 특정 OR의 선택적 발현 증폭 및 다른 OR의 발현 감소로 이어졌으며, 이 때 GI hub는 해당 OR 특이적인 hub가 새롭게 활성화되고 나머지는 silencing됨.
Fig. 4. 단일 OR 유전자 선택을 위한 모델로서의 Symmetry breaking
(A) OSN 발달 중 GI 3D 재구성, chromatin 리모델링 및 OR 전사에 대한 모델.
(B, C) (좌측) gg8-tTA>tetOP-2iGFP (B) 및 OMP-tTA>tetOP-2iGFP (C) 마우스의 MOEs에서 P2 단백질(magenta)과 GFP(green)를 대상으로 한 immunofluorescence 분석 결과. 각 immunofluorescence 패널의 왼쪽에서, tTA 드라이버와 그 표현 단계가 주황색 세포로 표시. 각 패널 상단의 개략도는 원래 위치에 있는 변형된 P2 allele의 디자인을 나타냄. (중앙) RNA-seq MA 플롯이 OMP-tTA>tetO-GFP에 대해 DeSeq2 정규화된 OR 유전자 계수를 나타냄. 유의하게 변화된 OR들이 빨간색으로 표시. (우측) GFP 양성 세포에서 Hi-C를 사용하여 P2와 주변 1Mb 반경의 모든 trans GIs 사이의 contacts per billion(cpb)을 집계하여 측정. 점선 검정색 선은 OMPiGFP OSNs에서의 P2 to GI contacts (음성 대조군).
(D-F) OMP-tTA>tetOP-2iGFP (D), Mor28icre>tdTom fl/+ (E) 및 OMP-tTA>tetOP-2iGFP; Mor28icre>tdTom fl/+ (F) 마우스의 후각 상피도.
(G) GFP+, tdT+, GFP+tdT+ 세포에서 P2(상단) 및 Mor28 위치(하단)에 대한 ATAC-seq 결과. 이는 quadruple transgenic 마우스의 GFP+, tdT+, GFP+tdT+ 세포 또는 OMPiGFP 마우스의 GFP+ 세포에서 수행됨. 각 조건에 대해 ATAC-seq는 세 개의 별도의 생물학적 반복에서 수행된 결과를 병합.
(H) 세 가지 세포 유형 모두에서 세 개의 생물학적 반복에 대한 Mor28, P2 및 중간 비활성 OR의 RNA-seq reads(TPM).
(I-N) GFP+(I, L), tdT+(J, M), GFP+tdT+(K, N) 세포에서 P2(i–k) 또는 Mor28(l–n) 위치와 주변 1Mb 반경의 모든 trans GIs 사이의 집계 cpb를 측정하는 In situ Hi-C. 점선 검정색 선은 OMPiGFP OSNs에서의 P2(상단) 또는 Mor28(하단)과 GIs 사이의 contacts를 의미(음성 대조군). 각 조건에 대해 In situ Hi-C는 세 개의 별도의 생물학적 반복에서 수행된 결과를 병합.
[Fig 4A] OSN 전구 세포에서 여러 GI hubs가 다양한 OR 공동 발현을 유도하지만 mOSN 단계에서는 하나의 hub만 활성 상태로 남게 됨. 연구진은 이러한 메커니즘이 X 염색체 한 쌍 중 확률적으로 하나는
보호, 다른 하나는 silencing 시키는 symmetry breaking 원리를 제안함.
[Fig 4B] P2에 의해 유도된 다유전성 OR 전사 동안 gg8-tTA>tetO-P2 mouce의 mOSN에서 특정 OR이 우선적으로 선택되며, 이는 OR 유전자 간의 대칭을 깨고 특정 유전자 선택을 촉진하는
메커니즘이 존재함을 시사함. 더 강력한 P2 유도는 이전에
선택된 OR을 silencing 시키고 P2와 GI 간의 접촉을 강화함.
[Fig 4D-F] P2가 이전에 활성화된 GI hub를 하이재킹하는지, 단순히 silencing 시키는 것을 확인하기 위해 다른 OR 유전자를 선택했던
OSN을 추적할 수 있게 labeling한 후 P2 유도로 특정 OR을 모두 선택하도록 설계함.
[Fig 4G-N] 활성화되는 OR 종류
변화에 따라 기존 GI hub는 접촉이 사라지고 새로 유도된 OR과
그 근처의 다른 GI hub 사이에 새로운 접촉이 생김. 즉, 기존 활성화된 GI hub의 하이재킹이 아닌 새로운 hub의 활성화 및 기존 hub silencing에 의해 symmetry breaking이 발생함.
OR protein-independent symmetry breaking indicates potential non-coding OR RNA functions
– Translation으로 이어지지 않는 OR RNA 단독으로도 OR selection 기능을 수행할 수 있음.
– OR RNA가 단독으로 OR selection에 기여할 수 있음에도, OR protein은 이러한 selection이 안정적으로 수행되도록 하는 역할을 가짐.
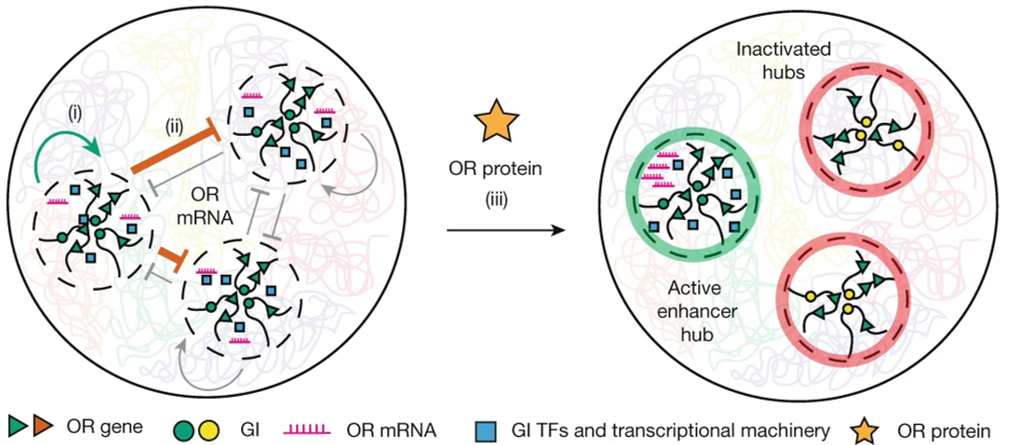
Fig. 5. OR RNA 생산은 singularity로의 전환을 촉진.
(A) (좌측) OMP-tTA>tetO-P2(nc)iGFP 마우스의 MOE에서 P2 단백질(magenta)과 GFP(green)을 대상으로 하는 immunofluorescence 분석 결과. mOSNs에서 P2 mRNA는 발현하지만 단백질은 발현하지 않음. (중앙) DEseq2로 정규화된 OR 유전자 계수를 OMPitTA>tetO-GFP로 정규화한 MA 플롯. 유의하게 변화된 OR 유전자는 빨간색으로 표시. (우측) GFP 양성 세포에서 in situ Hi-C는 P2와 주변 1Mb 반경의 모든 염색체간 GIs 사이의 집계된 cpb를 측정
(B) (좌측) gg8-tTA>tetO-P2(nc)iGFP 마우스의 MOE에서 P2 단백질(magenta)과 GFP(green)을 대상으로 하는 immunofluorescence 분석. (중앙) DEseq2로 정규화된 OR 유전자 계수를 gg8itTA>tetO-GFP로 정규화한 MA 플롯. 유의하게 변화된 OR들은 빨간색으로 표시. (우측) GFP 양성 세포에서 in situ Hi-C는 P2와 주변 1Mb 반경의 모든 염색체간 GIs 사이의 집계된 cpb를 측정.
(C) symmetry breaking 과정의 다른 단계에서 molecular dynamic 시뮬레이션 스냅샷
(D) 시간에 따른 가장 큰 cluster 내의 바인더의 비율을 모니터링함으로써 포착된 symmetry breaking의 dynamics. 가장 큰 cluster 내의 바인더의 비율은 하나의 큰 cluster 만이 평형에서 자체 조립되는 symmetry breaking 사건(EB > EBT(c))에 대해 관찰됨(빨간 곡선).
(E, F) 바인더가 생성 중인 OR mRNA에 친화성이 있는 경우, (E) RNA 농도 증가는 바인더-바인더 또는 바인더-hub 친화성을 증가시킬 것이며, (F) 이는 대칭 깨짐 과정을 가속화.
(G) 개략적 요약. OR RNA 합성(i)은 전사된 P2 위에 GI hub 형성을 편향시키고, 그것이 생성된 GI hub를 강화하며(ii) 경쟁 hub에서의 전사를 억제함. 이어서, P2 단백질 표현(iii)은 이 선택을 안정화시켜, tTA 없이도 지속적인 P2 표현을 가능하게 함. ctrl은 대조군, mut.은 변이.
[Fig 5A] P2와 유사한 non coding 유전자를 사용했을 때 단백질을 만들 수 없음에도 기존과 동일하게 특정 OR을 제외한 나머지 OR을 충분히 silencing할
수 있음. RNA 단계에서 symmetry breaking 메커니즘이
동작함을 시사함.
[Fig 5B] OR RNA가 전사 특이성에 중요한 역할을 함에도 non coding RNA만으로는 안정적인 OR 선택이 이뤄지지 않았으며, 이는 OR 단백질이 OR 선택 안정화에 기여함을 의미.
[Fig 5C] Fig. 5A에서 정상적인 신경 세포 발달에 따라 H3K27me3가 감소한 것과 유사한 변화가 EZH2i 처리 시 관찰됨.
*Fig 5D-F는 Discussion에 인용하여 설명
Disscussion
우리는 단일 지배적 cluster 내에서 GI hub 결합 인자들의 자기 유사성이 다수의 경쟁하는 allele들 중 하나의 OR allele을 선택하고 전사하는 위상 분리를 초래하는 물리 기반 대칭 깨짐 모델을 제안한다(Fig. 5e,h). symmetry breaking은 여러 hub에서 낮은 다유전자 OR 표현이 단일 hub에서 강력한 단일 OR 표현으로 귀결될 수 있는 방법을 설명할 수 있다. 이는 GI hub 결합 인자들이 단일 큰 cluster로 조직되기 전에 여러 작은 초점으로 집약되는 과정을 통해 이루어진다(Fig. 5c 및 Extended Data Fig. 10j,k). 이 과정은 아마도 초기 분화 단계에서 cis GI–OR 상호작용으로 시작되어, trans GI 모집과 경쟁하는 GI hub의 조립을 알리는 낮은 수준의 다유전자 OR 전사를 시작한다. 이 단계에서, 현지 RNA 합성의 자발적 변동이 하나의 hub에서 전사를 강화하고 다른 hub를 침묵시키는 위상 전환의 신호가 될 수 있다(Fig. 5c–f). 생성 중인 OR RNA의 스스로의 증강 및 다른 OR 억제 작용에 대한 한 가지 설명은 제한된 확산 가능한 transactivator의 효율적 모집에 기여한다는 것이다(Fig. 5e). 우리는 이 transactivator가 mOSNs에서 낮은 수준으로 전사되고 OR RNA의 시퀀스 및/또는 구조, LHX2, EBF1 또는 LDB1에 대한 친화성, 그리고 농도 의존적 위상 분리 특성을 나타낸다는 가설을 제안한다. 이런 맥락에서, OR RNA는 이 제한된 인자를 모집함으로써 현지 hub enhancer로 작용하고 다른 hub에서 그것을 격리함으로써 전역 hub repressor로 작용한다. 이러한 이중 기능으로, OR RNA 합성의 작은 변동이 hub 간의 대칭을 빠르게 깨뜨려 단일 OR 전사로의 전환을 가속화할 수 있다(Fig. 5e–g). symmetry breaking 모델의 매력은 OR 전사의 두 가지 모드만을 허용한다는 것이다. 여러 hub에서의 낮고 다유전자 전사(신경 계통 세포의 초기 발달 단계, INPs에서 iOSNs까지), 또는 하나에서의 단일하고 강력한 전사(mOSNs)이다. 즉, 지배적인 염색체간 GI hub에서의 높은 OR 전사 비율이 분화 동안 단일 OR 표현을 강제한다. 뉴런에서 염색체간 구획을 형성하는 유전자 목록이 계속 확장됨에 따라, 서로 배타적인 세포 운명 결정에서 다른 코딩 mRNA의 비코딩 역할을 조사하는 것이 흥미로울 것이다.
우리는 RNA 매개 symmetry breaking 과정을 선호하지만, 우리 데이터의 다른 설명을 무시할 수 없다. P2 DNA 및 GI hub 조립에 전사 인자 결합을 용이하게 할 수 있는 OR 위치의 전사 가능한 염색질 리모델링도 tTA 유도 시 편향된 P2 선택에 기여할 수 있다. 마찬가지로, tTA는 P2 프로모터에서 내인성 전사 인자와 협력하여 P2 위치로 GI hub 모집을 용이하게 할 수 있다. 그러나 두 시나리오 모두에서, P2 RNA 수준이 임계값에 도달할 때까지 경쟁하는 OR–GI hub 상호작용은 사라진다. 이는 OR mRNA가 symmetry breaking에서 직접적인 역할을 한다는 것을 지지한다. 우리는 또한 tTA 유도된 P2 전사가 다유전자 상태(INPs, iOSNs)에서 경쟁하는 내인성 OR들의 전사보다 강하다는 것을 인정하며, 이는 P2 선택을 인위적으로 편향시킬 수 있다. 그러나, tTA 주도의 P2 전사는 mOSNs에서 이미 선택된 OR의 전사만큼 높지 않지만, OR 선택 장치를 또한 탈취한다. 따라서, tTA 유도가 P2에 부여하는 전사적 이점이 MOE의 dorsoventral axis (등허리축)을 따라 다른 내인성 OR들이 가지는 이점을 모방할 가능성이 높으며, 이는 편향된, 위치에 따른 방식으로 대칭을 깨뜨린다.