병원체 protein의 모듈성을 통한 정교한 host phosphatase 모방
Abstract
병원체는 숙주 세포를 조작하기 위해 다양한 effector protein을 생산합니다. 그러나 effector 레퍼토리에서 기능적 다양성이 생성되는 방식은 잘 알려져 있지 않습니다. 식물 병원체인 Phytophthora의 많은 effector는 “(L)WY” 모티프의 탠덤 반복을 포함하며, 이는 구조적으로 보존되지만 시퀀스는 가변적입니다.
이 연구에서는 식물 host에서 Ser/Thr protein phosphatase 2A (PP2A) 핵심 효소를 효율적으로 recruit하는 여러 Phytophthora effector에서 특정 (L)WY-LWY 조합에 의해 형성된 기능 모듈을 발견했습니다. Effector-PP2A complex의 결정 구조는 (L)WY-LWY 모듈이 host PP2A 핵심 효소를 하이재킹하여 기능적 holoenzyme을 형성할 수 있음을 보여줍니다. N-term에서 PP2A 상호작용 모듈을 공유하는 동안, 이 effector는 다양한 C-term LWY 단위를 소유하고 host에서 별개의 phosphoprotein 세트를 조절합니다.
이 결과는 effector 레퍼토리에서 protein 모듈성에 의해 촉진되는 병원체와 다양화에 의한 분자 모방을 통한 필수 host phosphatase의 전유를 강조합니다.
Results
PSR2 hijacks PP2A core enzymes in plant hosts to promote disease
LWY 단위가 특정 host 분자와의 상호작용을 조절할 수 있는지 확인하기 위해 immunoprecipitation과 이어서 mass spectrometry (IP-MS)까지 사용하여 Arabidopsis thaliana에서 PSR2와 상호작용 하는 protein을 찾았습니다.
[Figure 1] Ser/Thr protein phosphatase 2A (PP2A) core 효소는 Phytophthora effector PSR2의 표적이 되는 감수성 인자입니다
(A) PSR2는 Arabidopsis에서 PP2A A subunit, C subunit과 연관되지만 B subunit과는 연관되지 않음을 보이는 IP-MS 결과.
(B) PSR2는 PP2A A subunit과 함께 co-immunoprecipitate 됨을 보이는 결과.
(C) PSR2 complex는 PP2A phosphatase 활성을 가짐을 보이는 결과.
(D-E) PSR2의 독성 활동에 PP2A A subunit RCN1 (D)와 PP2A C subunit PP2A-4 (E)가 필요함을 보이는 결과.
Figure 1A와 1B를 통해 IP-MS와 co-immunoprecipitation 결과 PSR2가 Arabidopsis에서 PP2A와 연관되어 있음을 확인했습니다.
Figure 1C는 형질전환 된 식물 조직에서 풍부한 PSR2 protein complex가 PP2A 특이 억제제인 okadaic acid (OA)에 의해 완전히 억제될 수 있는 phosphatase 활성을 가지고 있다는 점을 보입니다.
Figure 1D에서 PP2A A subunit 돌연변이인 rcn1에서 PSR2를 발현시켰고 이 독성 활성이 거의 사라졌음을 관찰했습니다.
Figure 1E는 rcn1과 유사하게 PSR2의 독성 활성도 PP2A C subunit 돌연변이인 pp2a-4 배경에서 거의 사라졌으며, 이는 식물 host에서 PSR2의 세포 기능이 PP2A phosphatase 활성에 의존함을 의미합니다.
> 이러한 결과는 PSR2와 PP2A가 상호작용하며 PP2A의 phosphatase 기능을 통해 PSR2가 식물 내에서 기능함을 알 수 있습니다.
PSR2 forms a complex with PP2A A subunit through LWY2-LWY3
PSR2가 Arabidopsis 핵심 효소 중 PP2A A and/or C subunit와 직접 상호작용할 수 있는지 여부를 확인하기 위해 in vitro pull-down과 gel filtration assays를 수행했습니다.
[Figure 2] PSR2는 (L)WY2-LWY3에 의해 형성된 인터페이스를 통해 PP2A A subunit과 상호작용합니다
(A) PDF1-PSR2 binary complex의 형성을 보이는 gel filtration chromatography 결과.
(B) 분해능이 2.3Å인 binary PSR2 (59–670 aa)-PDF1 (1–390 aa) complex의 결정 구조.
(C) PSR2-PDF1 상호작용 인터페이스는 LWY3 (회색)의 세 번째 α 나선 (α3)을 중심으로 하고 양쪽에서 L1과 L2에 의해 강화됨을 보이는 결과.
(D–F) α3 (D), L1 (E), L2 (F)의 12개 residue가 PDF1 (회색)과 직접 상호작용함을 보이는 결과.
(G) LWY3의 α3에서 REQ triad (R256, E260, Q263)가 PDF1 상호작용에 필수적임을 확인한 In vitro pull-down 분석 결과.
(H-I) REQ triad가 식물에서 기능성 PSR2-PP2A holoenzyme을 형성하는 데 필요함을 보이는 결과.
Figure 2A에서는 PSR2가 PDF1과 안정적인 binary complex를 형성함을 보여 주었습니다.
Figure 2B는 2.3Å에서 PSR2(59–670 aa)-PDF1(1–390 aa) complex의 결정 구조를 풀었습니다.
Figre 2C와 2D에서는 PSR2가 코어 역할을 하는 LWY3의 세 번째 α 나선 (이하 α3)과 광범위한 상호작용 인터페이스를 형성한다는 것과 α3는 PDF1 표면의 작은 홈에 삽입되어 PDF1과 다중 수소 결합 및 π-π 상호작용을 형성함을 보입니다.
Figure 2E와 2F를 보면 2C에서 표현한 LWY3 (이하 L1, L2)의 양쪽 연결은 L1의 K299, Y303, E307, D310 (2E) L2의 K211, K215, K219, Q221 (2F)와 같이 charged residue가 풍부합니다. 이런 residue들은 PDF1과 염다리 결합 (salt bridge bond) 또는 수소 결합을 형성하여 complex를 더욱 안정화시킵니다.
Figure 2G와 2H에서는 상호작용 인터페이스의 중심으로서 이 triad의 역할과 일치하게, PSR2 돌연변이 R256A/E260A/Q263A가 in vitro (2G)와 식물 (2H)에서 PDF1 상호작용이 거의 사라졌습니다.
Figure 2I는 돌연변이가 N. benthamiana에서 발현될 때 PSR2 R256A/E260A/Q263A protein complex에서 phosphatase 활성이 더 이상 검출되지 않음을 보입니다.
> 이러한 결과는 “REQ” triad는 PSR2-PDF1 complex 형성에서 중심적인 역할을 하며, 중요한 것은 특정 기능을 할당할 수 있음을 보였습니다. 또한, PSR2의 (L)WY 단위가 기능적 모듈 역할을 할 수 있음을 보여줍니다.
PSR2 binds tighter to the PP2A core enzyme than an Arabidopsis B subunit
PSR2가 PP2A core enzyme을 recruit 할 때 endogenous B subunit과 유사하게 결합하는지 여부와 효율적으로 경쟁할 수 있는지 여부를 조사하기 위해 PDF1과 PSR2 또는 Arabidopsis PP2A B subunit ATB′γ의 결합 친화도를 비교했습니다.
[Figure 3] PSR2는 PP2A B subunit의 분자 모방체로서 기능적 holoenzyme을 형성하기 위해 host core enzyme를 효율적으로 모집할 수 있습니다
(A) PDF1의 PSR2 결합 부위 (빨간색)가 인간 PP2A A subunit PPP2R1A에 보존되어 endogenous B subunit에 결합함을 보이는 결과.
(B) PDF1 (회색)과 PPP2R1A (청록색)의 보존된 residue가 각각 PSR2, 인간 PP2A B subunit과 상호작용함을 보이는 결과.
(C) Arabidopsis PP2A B subunit ATB’γ를 갖는 PDF1보다 PSR2와 PDF1의 더 높은 결합 친화성을 나타내는 등온 적정 열량계 (Isothermal titration calorimetry : ITC) 측정 결과.
(D) PSR2가 In vitro에서 미리 형성된 PDF1-ATB’γ-Cα holoenzyme로부터 ATB’γ를 효율적으로 대체함을 보이는 gel filtration chromatography 결과.
(E) ATB’γ가 미리 형성된 PDF1-PSR2-Cα holoenzyme에서 PSR2를 효율적으로 대체할 수 없음을 보이는 gel filtration chromatography 결과.
Figure 3A를 보면 PSR2는 알려진 모든 인간 PP2A B subunit과 구조적으로 구별되지만 A subunit의 N-term 부분에 있는 유사한 영역에 결합합니다.
Figure 3B는 실제로 PDF1와 PPP2R1A에 보존된 여러 residue가 각각 PSR2 및 인간 B subunit과의 상호작용에 관여함을 보입니다.
Figure 3C에서는 ITC를 사용하여 PSR2-PDF1 결합에 대한 Kd는 0.35 ± 0.08 μM로 추정되었습니다. 이에 비해 ATB’γ-PDF1 결합에 대한 Kd는 약 3.33 ± 0.49 μM로 PSR2-PDF1보다 약 10배 더 높았습니다.
Figure 3D를 보면 미리 형성된 PDF1-Cα-ATB’γ complex에 PSR2를 추가할 때 PSR2는 ATB’γ의 대체를 촉진하고 PDF1-Cα core enzyme으로 자체 holoenzyme을 형성했습니다.
반면, Figure 3E에서는 ATB’γ는 미리 형성된 PDF1-Cα-PSR2 holoenzyme으로부터 PSR2를 효율적으로 분리할 수 없었습니다.
> 이러한 결과는 PSR2가 B subunit의 분자 모방체로서 PP2A 핵심 효소를 경쟁적으로 recruit 할 수 있음을 보여줍니다.
The PP2A-interacting module is adopted in multiple LWY effectors & Effector-PP2A holoenzymes are diverse
LWY effector의 모듈식 구조를 고려할 때 PSR2 (L)WY2-LWY3 모듈을 포함하는 모든 effector도 PP2A core enzyme를 모집할 수 있다고 추론했고 이를 테스트하기 위해 P. sojae와 P. infestans에서 예측된 134개의 WY1-(LWY)n effector에 대해 구조와 서열을 기반으로 유사성 검색을 진행했습니다.
추가적으로 PP2A 상호작용 effector의 기능에서 잠재적 다양성을 조사하기 위해 PP2A recruit effector에서 C-term vs N-term LWY 단위의 진화 궤적을 분석했습니다.
[Figure 4] 12개의 effector는 N-term에 PSR2와 같은 PP2A 상호작용 모듈을 채택했지만 다양한 C-term LWY 유닛을 가지고 있습니다
(A) PP2A 상호작용 모듈을 보유할 것으로 예상되는 12개의 LWY effector.
(B) C-term LWY 단위는 PP2A 관련 effector에서 더 높은 수준의 다양성을 나타냄을 보이는 결과.
Figure 4A에서는 N. benthamiana에서 개별적으로 발현되는 effector를 확인했고 effector protein complex를 풍부하게 만들었습니다. 여기에서 PP2A A subunit과의 상호작용과 phosphatase 활성 보유를 조사한 결과 P. infestans의 PSR2 ortholog를 포함하여 15개의 후보 effector 중 12개가 planta에서 기능성 PP2A holoenzyme을 형성했습니다.
식물 PP2A core enzyme와 결합할 수 있는 13개의 LWY effector에서 최소 5개의 WY/LWY 단위를 포함하고 예측된 PP2A 상호작용 모듈이 항상 N-term에 위치한다는 것을 관찰했습니다.
Figure 4B에서 진행한 구조 및 서열 유사성에 대한 pairwise analysis 결과 N-term의 PP2A 상호작용 단위와 비교하여 C-term LWY 단위에서 전반적으로 더 높은 다양성을 나타냈습니다.
> 이러한 결과는 PSR2와 유사한 PP2A 상호작용 모듈의 소유와 Phytophthora effector에 의한 host PP2A core enzyme을 가로채는 능력 사이에는 강한 상관관계가 있고 PP2A core enzyme과의 연관성에 대해 LWY의 유사한 메커니즘이 effector에 의해 사용됨을 나타냅니다.
PSR2 and PITG_15142 regulate the phosphorylation of different host proteins
PP2A 상호작용 effector의 기능적 보존과 다양성을 더 자세히 조사하기 위해 다양한 종에서 구조를 분석하였고 liquid chromatography-mass spectrometry (LC-MS)를 사용하여 라벨 없는 정량적 phosphoprotein 분석을 수행했습니다.
[Figure 5] PSR2 및 PITG_15142는 별개의 host protein 세트의 인산화에 영향을 미칩니다
(A) 해상도가 3.1Å인 P. infestans effector PITG_15142의 결정 구조.
(B) PITG_15142 (81–221 aa, WY1의 경우 녹색, LWY2의 경우 노란색)와 apo 상태 PSR2 (170–310 aa, 회색)에서 PP2A 상호작용 모듈의 구조 중첩.
(C) REQ triad가 필요한 PDF1과 PITG_15142의 직접적인 상호작용을 보여주는 In vitro pull-down.
(D) N. benthamiana에서 PITG_15142의 독성 활동에 필요한 REQ triad.
(E) PSR2, PITG_15142에 존재하는 다양한 C-term 단위.
(F) PSR2가 Arabidopsis의 phosphoproteome에 미치는 영향을 보이는 volcano plot.
(G) rcn1-6 돌연변이에서 PSR2를 발현하는 식물에서 PSR2 감소된 phosphopeptide의 변화를 보여주는 volcano plot.
(H) PSR2 또는 PITG_15142를 발현하는 Arabidopsis에서 인산화 수준이 감소된 phosphoprotein의 gene ontology (GO) 분석.
(I) PSR2 또는 PITG_15142를 발현하는 Arabidopsis에서 감소된 phosphopeptide의 벤 다이어그램.
Figure 5A에서는 P. infestans effector PITG_15142의 결정 구조를 3.1Å에서 결정했습니다.
Figure 5B를 보면 PITG_15142는 N-term (L)WY 단위에 예측된 PP2A 상호작용 모듈이 있는 WY1-(LWY)4 구조를 가지고 있고 실제로 PITG_15142의 WY1-LWY2는 PSR2의 (L)WY2-LWY3과 상당한 구조적 유사성을 가지며 RMSD (root-mean-square deviation) 점수는 1.38Å입니다. PDF1과의 직접적인 상호작용을 매개하는 PSR2의 7개 residue는 PITG_15142에 보존됩니다. 이러한 보존된 residue에는 REQ triad에 해당하는 R167, E171, Q174가 포함됩니다.
Figure 5C는 PSR2와 유사하게, REQ triad의 돌연변이는 in vitro에서 PITG_15142와 PDF1의 상호작용을 감소시켰습니다.
Figure 5D에서는 PITG_15142 R167A/E171A/Q174A 돌연변이가 N. benthamiana에서 P. capsici 감염을 촉진할 수 없었으며, 이는 REQ triad가 식물 세포에서 PITG_15142의 기능에 필수적임을 나타냅니다.
Figure 5E에서는 PP2A 상호작용의 유사성에도 불구하고 PSR2와 PITG_15142 구조를 비교한 결과 C-term 단위에서 차이가 드러났습니다. PSR2(LWY5-LWY7)와 PITG_15142(LWY3-LWY5)를 비교한 결과 RMSD 값이 3.43Å인 것으로 나타났습니다.
Figure 5F에서 확인한 결과 peptide의 인산화 상태에 영향을 미쳤습니다. PSR2-5에서, 250개의 peptide는 인산화가 감소된 것을 보였고 126개는 인산화가 증가된 것을 보였습니다.
Figure 5G를 보면 PSR2-5에서 인산화가 감소한 250개의 phosphopeptide 중 204개는 PSR2가 rcn1-6 돌연변이 상태에서 발현될 때 더 이상 변화하지 않았습니다.
Figure 5H는 PSR2에 의해 인산화가 감소된 protein의 gene ontology (GO) 분석을 보였고 이는 nucleobase-containing compound의 생합성 과정에서 농축을 의미합니다. 하지만 PITG_15142에 의해 인산화가 감소된 peptide는 주로 곰팡이나 건조에 대한 반응에 관여합니다.
Figure 5I를 봤을 때 PSR2-발현 식물과 PITG_15142-발현 식물 모두에서 하나의 peptide만이 인산화가 감소되었으며, 이는 두 effector가 별개의 host protein 세트의 인산화를 조절한다는 개념과 일치합니다.
> 이러한 결과는 PITG_15142와 PP2A 상호작용 모듈을 소유한 또 다른 LWY effector가 PDF1과 상호작용하고 PSR2에서 사용하는 동일한 메커니즘을 통해 effector-PP2A holoenzyme을 형성함을 검증했습니다. 또한 기질 결합의 잠재적인 다양화를 보여주었고 추가적으로 PSR2 매개 host protein의 탈인산화가 Arabidopsis PP2A core enzyme에 의존한다는 것을 입증했습니다. PP2A 상호작용 모듈을 포함하는 effector가 host core enzyme와 holoenzyme을 형성하지만 C-term LWY 단위를 기반으로 다른 phosphoprotein을 조절한다는 것을 확인했습니다. 이러한 effector는 기능적 다양성을 나타내며 LWY 탠덤 반복을 기반으로 하는 모듈식 구조에 의해 가능합니다.
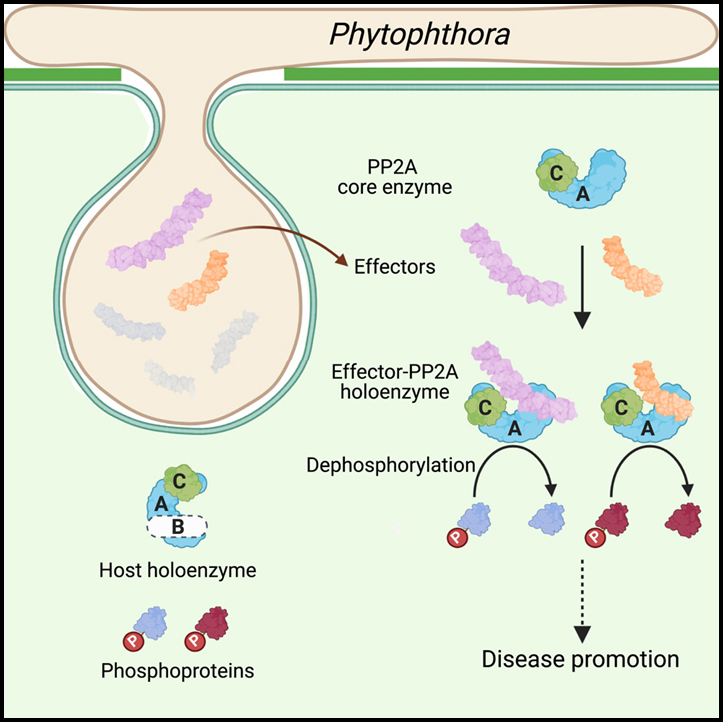
[Figure 6] Effector 진화가 LWY 반복 단위에 기반한 protein 모듈화에 의해 어떻게 촉진될 수 있는지를 보여주는 모델
Disscussion
주요한 protein phosphatase인 PP2A는 다양한 세포 과정을 조절하며 그 기능 장애는 많은 질병과 관련이 있습니다. 인간 PP2A는 발암성 바이러스에 의한 독성 표적이며, 그 중 일부는 핵심 효소에 결합할 수도 있습니다. 그러나 이러한 바이러스 protein은 대부분 B subunit의 기능을 모방하기보다는 PP2A 효소 활성을 억제합니다. 식물에서 PP2A는 면역 기능을 조절하고 세균성 병원균 Pseudomonas syringae의 Type III-secreted effector AvrE와 Erwinia amylovora의 homolog DspA/E의 독성 기능과 연관되는 것으로 보고되었습니다. AvrE는 독성 활동에 기여하는 여러 PP2A B subunit과 상호작용합니다. 또한, oomycete 병원체 Hyaloperonospora arabidopsidis에 의해 생성된 effector HaRxL23은 AvrE의 독성 활성을 부분적으로 보완할 수 있으며, 이는 HaRxL23이 host PP2A를 조작할 수도 있음을 나타냅니다. Phytophthora에서 여러 PP2A B 모방 effector를 식별한 결과 PP2A가 독립적인 메커니즘을 통해 계 전체의 병원균에 의해 광범위하게 표적이 되는 주요 인자라는 것이 입증되었습니다. 이것은 Pseudomonas syringae와 Phytophthora capsici 모두에 대한 PP2A 핵심 효소 돌연변이의 저항성을 가진 표현형과 일치합니다. Effector-PP2A complex의 상세한 구조적 특성 분석은 정확한 엔지니어링을 통해 질병 저항성을 향상시킬 수 있는 새로운 기회를 제공합니다.
가속화된 effector 진화는 병원체의 host 적응에 필수적입니다. 동일한 기능 모듈의 채택을 통해 여러 Phytophthora effector에 의한 주요 효소 complex의 정교한 host 모방은 protein 모듈성이 예측 가능한 방식으로 독성 표적의 확장을 촉진할 수 있는 방법을 보여줍니다. 구조적으로 보존되지만 기능적으로 가변적인 (L)WY 단위는 기능 모듈의 저장소 역할을 할 수 있습니다. 탠덤 반복으로 이러한 모듈의 셔플링은 host 조작의 새로운 활동이 발생할 수 있는 effector 레퍼토리의 진화 가능성을 향상시킬 수 있습니다. 박테리아 병원체에서, promoter와 N-term translocation 신호에 걸친 DNA 서열의 “reassortment”는 새로운 type III-secreted effector가 생성될 수 있는 메커니즘으로 제안 되었습니다. 따라서 modular protein/gene 구조에 기반한 기능적 단위의 셔플링은 병원성 진화에서 공통된 주제입니다.