학습된 surface fingerprints와 단백질 상호작용의 새로운 설계
Abstract
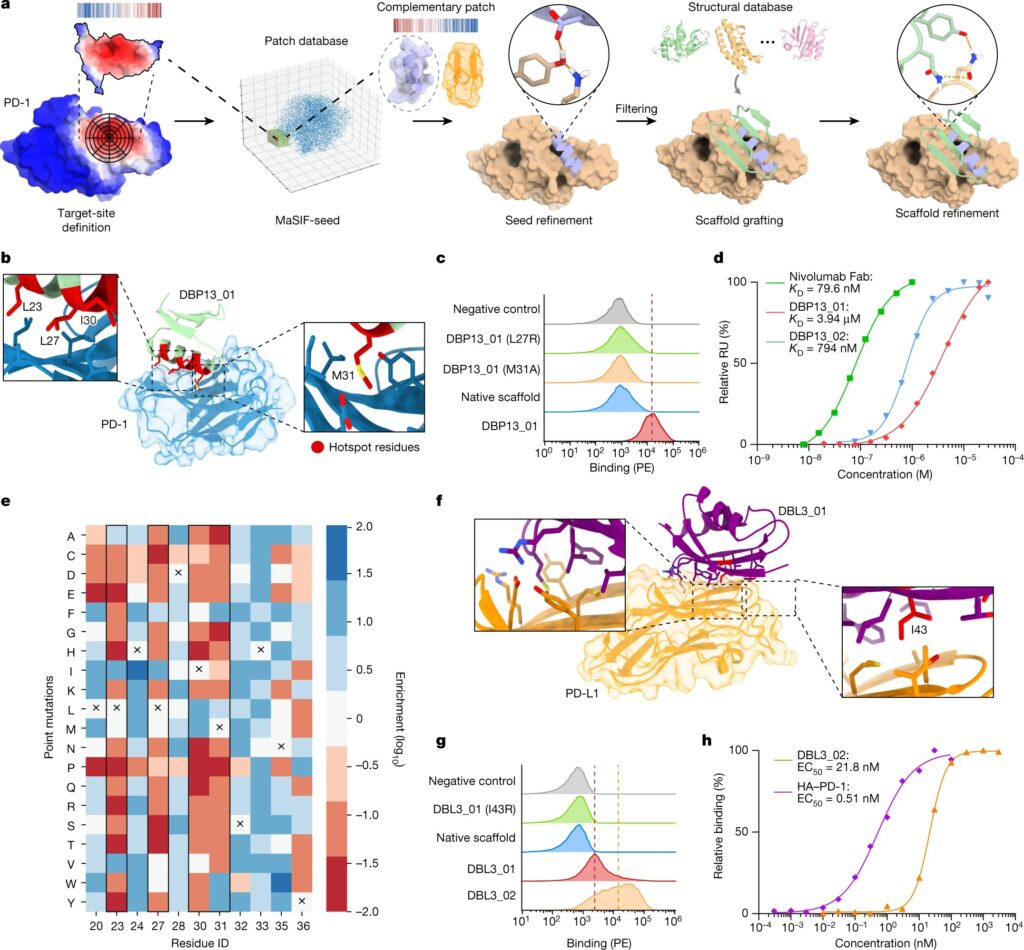
단백질 간의 물리적 상호작용은 생명체를 지배하는 대부분의 생물학적 과정에 필수적입니다. 그러나 genomic, proteomic 및 structural 데이터가 증가함에도 불구하고 이러한 상호작용의 분자적 결정 요인을 이해하기는 어려웠습니다. 이러한 지식 격차는 cellular protein–protein interaction 네트워크에 대한 포괄적인 이해와 합성 생물학 및 번역 응용 분야에 중요한 단백질 결합체의 신규 설계에 큰 장애물이 되어 왔습니다. 여기서는 단백질 표면에서 작동하는 기하학적 딥러닝 프레임워크를 사용하여 protein–protein interaction을 유도하는 데 중요한 기하학적 및 화학적 특징을 설명하는 지문을 생성합니다. 우리는 이러한 지문이 새로운 단백질 상호 작용의 계산 설계에서 새로운 패러다임을 나타내는 분자 인식의 핵심 측면을 포착한다는 가설을 세웠습니다. 원칙을 증명하기 위해, 우리는 네 가지 단백질 표적과 결합하는 몇 가지 새로운 단백질 결합체를 컴퓨터로 설계했습니다: SARS-CoV-2 spike, PD-1, PD-L1 및 CTLA-4. 몇몇 디자인은 실험적으로 최적화한 반면, 다른 디자인은 순수하게 in silico로 생성되어 구조 및 돌연변이 특성화를 통해 nanomolar affinity에 도달하여 매우 정확한 예측을 보여주었습니다. 전반적으로 surface-centric approach는 molecular recognition의 물리적, 화학적 결정 요인을 포착하여 단백질 상호 작용과 더 넓게는 기능을 갖춘 인공 단백질의 새로운 설계를 위한 접근 방식을 가능하게 합니다.