bacterial cell 내부의 atomic detail에서 translation dynamics 시각화
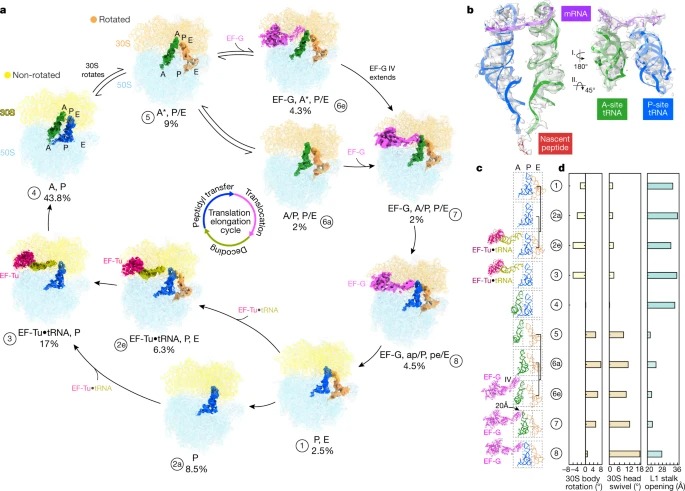
Translation은 protein synthesis의 기본 과정이며, 모든 살아있는 세포에서 리보솜에 의해 촉매된다. 여기서 우리는 Mycoplasma pneumoniae 내부의 Translation의 구조역학을 시각화하기 위해 cryo-electron tomography과 sub-tomogram analysis을 사용한다. functional state를 자세히 해석하기 위해 먼저 모든 translating 리보솜의 high-resolution in-cell average map을 얻고 M에 대한 원자 모델을 구축한다. 리보솜 단백질의 뚜렷한 확장을 드러내는 pneumoniae 리보솜 그런 다음 분류는 형태와 구성이 다른 13개의 리보솜 상태를 해결한다. 이것들은 이전에 in vitro에서 해결된 주요 상태를 요약하고, active translation 중에 중간생성물을 반영한다. 이러한 상태를 기반으로, 우리는 네이티브 세포 내부의 translation elongation을 애니메이션화하고 항생제가 어떻게 세포 translation 환경을 재구성하는지 보여준다. translation elongation 동안 리보솜은 종종 정의된 3차원 배열로 조립되어 polysome을 형성한다. translating 리보솜의 intracellular organization을 매핑함으로써, 우리는 polysome으로의 그들의 연관성이 리보솜 단백질 L9에 의해 매개되는 국소 조정 메커니즘을 포함한다는 것을 보여준다. 우리는 polysome 내에서 L9의 확장된 구성이 translation fidelity를 용이하게 하기 위해 충돌을 완화한다고 제안한다.
따라서 우리의 연구는 세포 내부의 atomic detail에서 molecular process를 시각화 할 수 있는 가능성을 보여준다.