포유류에 걸친 spermatogenesis의 분자적 진화
Abstract
Testis는 spermatogenesis를 통해 gametes를 생성하고 포유류의 형태학적, 분자적 수준에서 빠르게 진화한다. 그러나 포유류에 걸친 개별 spermatogenic 세포 유형의 분자 진화는 거의 특성화되지 않은 채로 남아 있다. 여기서 우리는 세 가지 주요 포유류 계통(eutherian, marsupial and monotreme)과 조류(the evolutionary outgroup)를 포함하고 7종의 영장류를 포함하는 11종의 testis에 대한 single-nucleus transcriptome data의 진화 분석을 보고한다. 우리는 testis의 빠른 진화가 late spermatogenic stage에서 유전자 발현 변화, 아미노산 치환 및 새로운 유전자의 accelerated fixation rate에 의해 주도되었다는 것을 발견했으며, 이는 아마도 reduced pleiotropic constraint, haploid 선택 및 transcriptionally permissive chromatin에 의해 촉진되었을 것이다. 우리는 종에 걸친 개별 유전자의 시간적 발현 변화와 조상 spermatogenic 과정을 제어하는 보존된 발현 프로그램을 식별한다. spermatogonia (spermatogenesis에 연료를 공급하는 생식 세포)와 Sertoli 세포(체세포 지지체)에서 주로 발현되는 유전자는 진화 과정에서 X 염색체에 축적되는데, 아마도 남성에게 유익한 선택적 힘 때문일 것이다. 추가 연구에서는 X- 및 Y-보유 정자 사이의 전사체 차이를 확인했으며 meiotic sex-chromosome inactivation (MSCI)가 monotremes에서도 발생하므로 포유류 성염색체 시스템에 공통적이라는 사실을 밝혀냈다. 따라서 MSCI의 기초가 되는 unsynapsed chromatin의 meiotic silencing의 메커니즘은 조상 포유류의 특징이다. 우리의 연구는 spermatogenesis와 관련된 선택적 힘의 분자 진화를 조명하고 포유류 전반에 걸친 testis의 생물학을 조사하기 위한 자원을 제공한다.
Figure
Fig. 1. 포유류 10마리와 조류 1마리에 대한 snRNA 프로파일링
a, snRNA-seq 데이터 세트의 표본 추출과 획일화한 manifold approximation and projection (UMAP).
b, cell-type pseudo-bulk의 Principal component (PC) 분석
c, 전체 testis에 대한 pseudo-bulk transcriptome에 기반한 유전자 발현 계통 발생.
Fig. 2. 유전자 발현 발산과 진화력
a, 양막류와 영장류의 testicular 세포 유형 중 발현 트리의 Total branch length.
b, 100개의 부트스트랩 복제(점)에서 인간과 다른 종 사이의 Spearman의 상관관계.
c, 인간에서 발현된 유전자의 평균 pLI 값 (pLI 점수는 기능 상실 돌연변이에 대한 유전자의 허용오차를 반영한다; 낮은 값은 허용오차가 낮다는 것을 의미한다.)
d, 마우스에서 녹아웃되었을 때 치명적인 표현형을 초래하는 발현된 유전자의 백분율(4,742개의 녹아웃 중).
e, 마카크(원숭이 종)에서 발현된 유전자의 over synonymous (dS) 뉴클레오타이드 치환에 대한 nonsynonymous (dN)의 평균 정규화된 비율.
f, 침팬지에서 양성 선택 하에 발현된 유전자의 비율(양성 선택을 위해 테스트된 유전자 11,170개 중)
g, 마우스에서 발현된 유전자의 평균 계통발생 연령.
h, 고릴라의 단백질 코딩 유전자(위) 또는 유전자 간 요소(아래)에 매핑되는 UMI의 백분율.
i, 인간의 주어진 세포 유형에서 우세한 발현을 가진 모든 유전자에 대해 표시된 번역 효율 값(참조 자료).
j, 주머니쥐에서 tissue-specificity value (참조 데이터)의 평균.
k, 마우스에서 불임과 관련된 발현 유전자의 백분율(3,552번의 녹아웃 중).
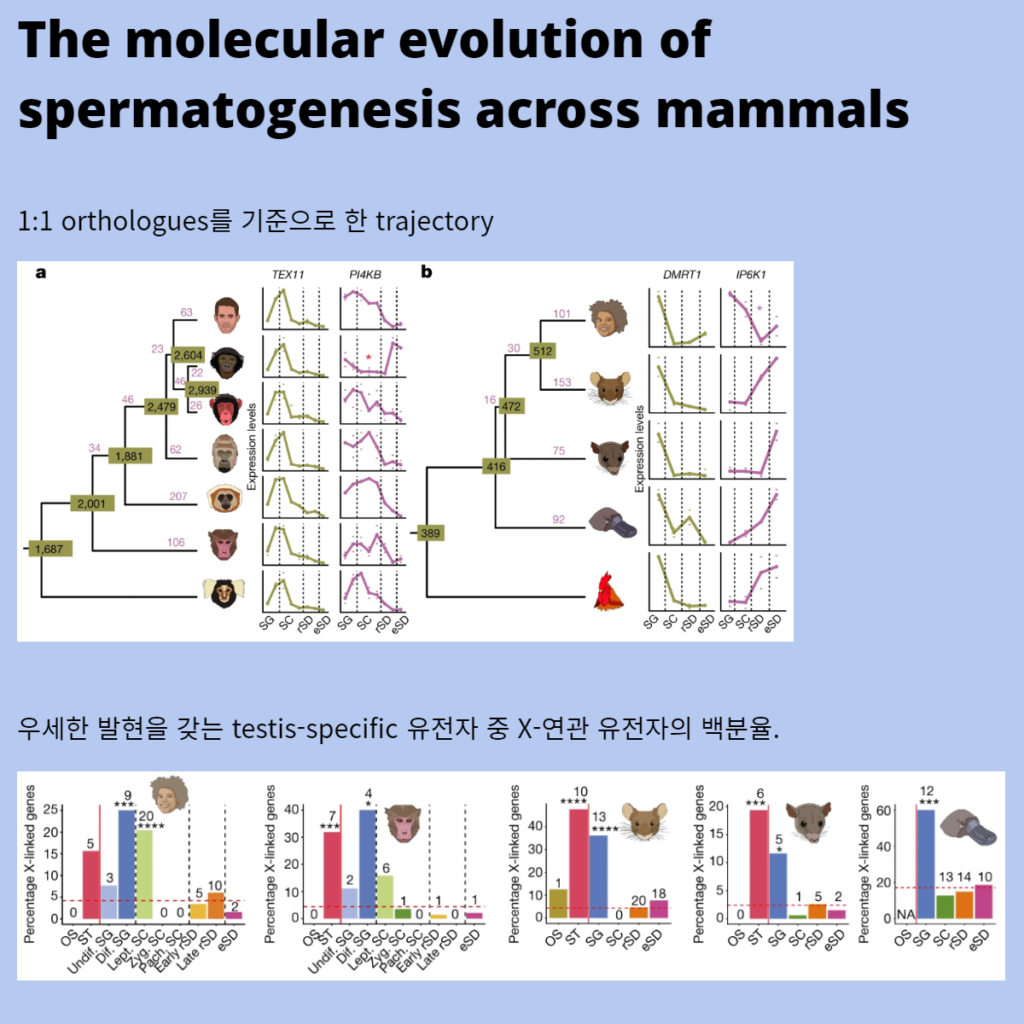
Fig. 3. spermatogenesis에 따른 유전자 발현 궤적의 진화
a, 4,459개의 1:1 orthologues를 기준으로 한 영장류의 궤적(인간, 보노보, 침팬지, 고릴라, 긴팔원숭이, 마카크, 마모셋; 위에서 아래로).
b, 2,927개의 1:1 orthologues를 기준으로 한 양막 궤적(인간, 쥐, 주머니쥐, 오리너구리, 닭; 위에서 아래로).
c, 인간, 보노보, 침팬지 및 고릴라(위에서 아래로)에 대한 spermatogenesis를 따라 snRNA-seq 데이터에서 RUBCNL 표현 궤적.
d, 인간, 침팬지, 오랑우탄 테스트에서 RUBCNL 발현 검출은 smISH using RNAScope에 의해 수행되었다.
왼쪽, 헤마톡실린으로 counterstain된 seminiferous tubule 단면과 SG, SC 및 spermatids (SD)가 포함된 영역의 클로즈업.
오른쪽, 섹션당 10개의 관에서 RUBCNL 발현 수준의 정량화.
Fig. 4. 포유류의 성염색체 진화
a, 인간, 쥐, 주머니쥐, 오리너구리(위에서 아래로)를 위한 계통수.
b, 인간, 마카크, 마우스, 주머니쥐 및 오리너구리에 대해 주어진 세포 유형에서 우세한 발현을 갖는 testis-specific 유전자 중 X-연관 유전자의 백분율.
Disscussion
우리의 분석은 이전에 관찰된 testis의 빠른 진화가 late spermatogenesis, 특히 rSD에서 molecular innovation의 accelerated rate에 의해 주도된다는 것을 발견했다. 우리의 연구 결과는 late spermatogenesis (아마도 정자 경쟁에 의해 주도됨)에서 진화하는 동안 조절 변화, 아미노산 변화 대체 및 새로운 유전자의 accelerated fixation이 reduced pleiotropic constraint, transcriptionally permissive chromatin environment 및 cytoplasmic bridge를 통한 transcript 교환을 통해 효과적으로 diploid 되지 않는 spermatid 유전자에 대한 잠재적인 haploid selection에 의해 촉진된 시나리오를 제안한다. testis의 급속한 진화를 설명하는 late spermatogenic stage와 일치하여, early spermatogenic cell type 및 somatic cell는 분자 수준에서 천천히 진화하는 기관인 뇌의 세포 유형과 유사한 제약 및 혁신 패턴을 보인다(확장 데이터 그림 3a,b,d). 우리는 testis(확장 데이터 그림 1f)에 대해 발음되는 포유류 간의 세포 유형 풍부도의 차이가 아마도 이 기관의 빠른 유전자 발현 차이에 기여했다는 것에 주목한다.
개별 유전자에 대한 우리의 종 간 비교는 종 간의 시간적 발현 차이를 드러냈으며, 이는 아마도 reduced pleiotropic constraint에 의해 촉진되었을 것이다. 따라서 우리의 결과는 종별 spermatogenesis 표현형의 진화에 대한 기여를 실험적으로 면밀히 조사할 수 있는 광범위한 후보 목록을 제공한다. 우리는 또한 개별 포유류 계통과 포유류 전체에 조상을 둔 spermatogenic 과정의 기초가 되는 보존된 표현 프로그램을 발견했다.
추가 분석은 spermatogenesis에서 성염색체 유전자의 역할을 밝혀냈다. 우리는 SG와 Sertoli 세포에서 주로 발현되는 유전자가 진화하는 동안 포유류와 그들의 두 성염색체 시스템에 걸쳐 X 염색체에 독립적으로 축적된다는 것을 발견했다. 이는 X염색체가 강력한 남성 관련 선택적 힘에 의해 형성되어 active transcription이 가능한 testis 세포 유형에서 기능적 역할을 하는 X- linked 유전자가 출현했음을 시사한다. 실제로, 여기서 발견된 SG 및 Sertoli 세포 enrichment 외에도, 이전 연구는 이러한 선택적 압력이 meiosis 이후 발현을 촉진하는 X에 유전자의 반복적인 복제로 이어진다는 것을 보여주었다.
X와 Y를 가진 정자를 분리하는 우리의 능력과 새로운 오리너구리 게놈 어셈블리의 가용성은 MSCI가 포유류의 성염색체 시스템의 일반적인 특징이라는 것을 밝혀냈고, MSUC가 공통 포유류 조상에 이미 존재했음을 나타낸다. 이전 연구에서는 조류에서 MSCI에 대한 증거를 찾지 못했는데, 이는 MSUC가 약 3억 1천만 년 전 파충류 계통에서 분리된 후 공통 포유류 조상에서 발생했는지 아니면 이 메커니즘이 조류 계통에서 사라져서 진화 초기에 발생했는지에 대한 의문을 제기한다. 후자의 시나리오는 무척추동물 종에서 MSCI의 관찰과 일치할 것이다.
우리의 데이터와 결과는 우리가 데이터 탐색을 용이하게 하기 위해 개발한 동반 온라인 리소스(https://apps.kaessmannlab.org/SpermEvol/))와 함께 포유류 전반에 걸친 testis 및 관련 불임 장애의 생물학을 조사하기 위한 광범위한 리소스를 제공한다. 향후 연구는 snRNA-seq 데이터의 한계를 극복하기 위해 우리의 snRNA-seq 데이터를 보완하는 것을 추구해야 한다. scRNA-seq 데이터는 cytoplasm 고유의 transcriptome pattern을 추론하는 데 유용할 것이고, testis의 확연한 isoform 다양성을 평가하기 위해서는 single-cell full-length transcript data가 필요하며, spermatogenesis 진화에 대한 post-transcriptional changes의 기여를 이해하기 위해서는 ingle-cell translatome data가 필요하다.