방향족 chitin biosynthesis의 Structural basis.
Abstract
자연에서 가장 풍부한 aminopolysaccharide인 chitin은 N-acetylglucosamine(GlcNAc) unit으로 구성된 extracellular polymer이다. chitin 생합성의 주요 반응은 UDP-GlcNAc에서 성장 중인 chitin 사슬로, GlcNAc를 전달하는 membrane-integrated glycosyltransferase인 chitin synthase에 의해 촉매된다. 그러나 이 과정의 정확한 메커니즘은 아직 설명되지 않았다.
여기서 우리는 파괴적인 콩뿌리 부패 병원성 Phytophthora sojae (PsChs1)에서 chitin 합성효소의 5가지 cryo-electron microscopy 구조를 보고합니다. 이들은 효소의 apo, GlcNAc -bound, nascent chitin oligomer-bound, UDP-bound (post-synthesis)및 chitin synthase inhibitor nikkomycin Z-bound states of the enzyme을 나타내며, chitin 생합성의 여러 단계에 대한 자세한 보기를 제공한다. 이 구조는 substrate-binding site, 촉매 중심 및 생성물 중합체가 배출될 수 있는 polymer-translocating channel의 입구를 갖는 chitin synthesis reaction chamber를 드러낸다. 이 배열은 UDP-GlcNAc binding 및 polymer elongation에서 생성물 release까지의 chitin 생합성에서 연속적인 주요 사건을 반영한다. 우리는 chitin-translocating channel 내에서 swinging loop를 확인했는데, 이는 product polymer를 세포막의 extracellular로 배출하기 위해 translocating channel로 유도하면서 substrate가 이탈하는 것을 방지하는 ‘gate lock’ 역할을 한다.
이 연구는 chitin 생합성의 방향성 다단계 메커니즘을 밝히고 chitin 합성을 억제하기 위한 구조적 근거를 제공한다.
Figure
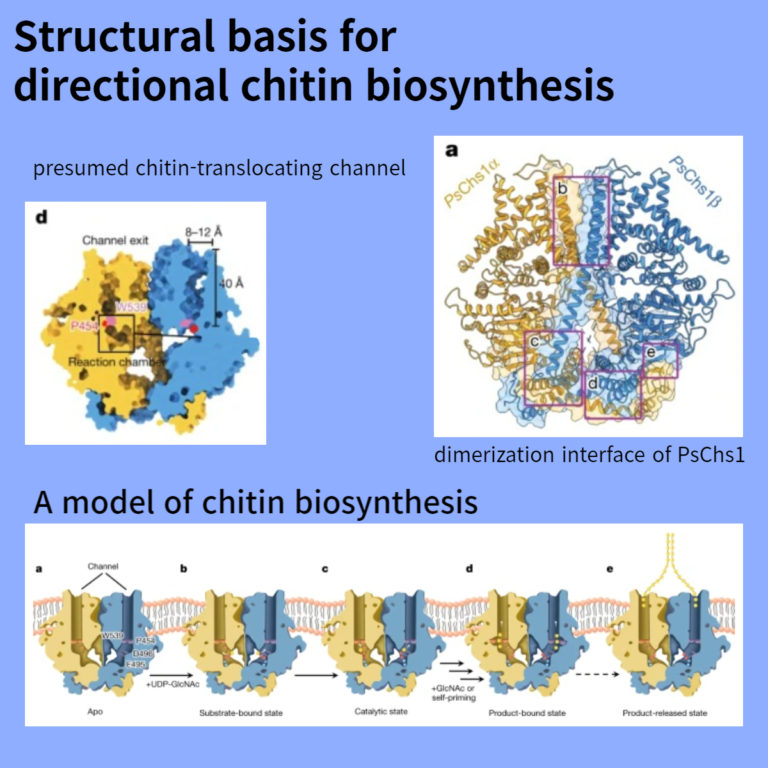
Fig. 1: The apo PsChs1 structure.
(a,b) 막의 extracellular side (top view), 막의 within the plane (side view), 또는 cytoplasmic side (bottom view)에서 바라본 PsChs1 dimer의 구조.
(c) PsChs1 protomer의 도메인 아키텍처 및 ribbon 표현.
(d) 추정된 chitin-translocating channel의 Sliced-surface view.
(e) PsChs1의 reaction chamber (왼쪽)와 conserved motifs의 reaction chamber (오른쪽)
Fig. 2: PsChs1 dimer안의 Interface interactions.
(a) ribbon 다이어그램으로 표시된 PsChs1의 dimerization 인터페이스의 전체도.
(b–e) 인터페이스 상호작용에 관련된 네 가지 영역 표시.
(b) PsChs1α TM2 with PsChs1β TM5
(c) PsChs1α GT domain, MIT subdomain and SP subdomain with PsChs1β TM5
(d) PsChs1α SP subdomain with PsChs1β MIT subdomain
(e) PsChs1α SP subdomain with PsChs1β LG subdomain
Fig. 3: PsChs1 and ligands 사이의 Interactions.
(a) UDP-GlcNAc-binding site의 Sliced-surface view (오른쪽) 및 UDP-GlcNAc와 PsChs1 사이의 상세한 상호 작용(왼쪽).
(b) apo-PsChs1 (light grey), UDP-GlcNAc-bound PsChs1 (yellow) and UDP/(GlcNAc)3-bound PsChs1 (pink) 구조의 중첩은 swinging loop and catalytic residues의 conformational changes를 보였다.
(c) PsChs1 in the UDP/(GlcNAc)3-bound state의 Sliced-surface view (middle), UDP/(GlcNAc)3의 밀도 (왼쪽), PsChs1와의 detailed interactions (오른쪽)
(d) UDP-binding site의 sliced-surface view (오른쪽), UDP와 PsChs1의 detailed interactions (왼쪽)
(e) UDP/(GlcNAc)3-bound PsChs1 (pink) 와 UDP-bound PsChs1 (cyan) 구조의 중첩.
Fig. 4. NikZ에 의한 of PsChs1의 Inhibition.
(a) NikZ의 화학적 구조와 EM 밀도.
(b) NikZ-binding site의 sliced-surface view (왼쪽), NikZ and PsChs1의 detailed interactions (오른쪽).
Discussion
PsChs1은 N-terminally intertwined dimer로 조립된 두 개의 거울상 protomers로 구성된 독특한 4차 구조를 나타내며, chitin 생합성의 parallel and directional mode를 시사한다. dimeric and oligomeric CHS 복합체의 형성은 이전에 in vitro and in vivo에서 보고되었으며 본 연구의 PsChs1의 구조 데이터와 최근 발표된 Candida albicans의 CaChs2와 일치한다. Dimeric PsChs1과 CaChs2는 모두 α-chitin을 생성한다. PsChs1과 CaChs2 또는 다른 CHS 사이의 시퀀스 정렬은 dimeric interface residues가 매우 가변적이며(Extended Data Fig. 1a) PsChs1 dimerization에 중요한 N-말단 SP 및 MIT 하위 도메인이 Oomycete CHS10에만 존재함을 나타낸다. 이러한 관찰은 우리 구조에 표시된 dimerization 메커니즘이 oomycetes과 일부 균류의 CHS에 고유할 수 있으며, 다른 분류군과 다른 올리고머화 전략이 진화했을 수 있음을 시사한다. 그럼에도 불구하고, 단일 촉매 단위의 well-ordered assembly는 coalescence전 nascent sugar chains 및 chitin 나노 섬유의 형성과 관련이 있기 때문에, CHS 올리고머화는 chitin 섬유의 형성에 중요한 것으로 보인다.
우리는 chitin 생합성이 free GlcNAc(Extended Data Fig. 2d, f)가 없을 때 시작된다는 것을 알아챘는데, 이는 생합성에 free GlcNAc가 필요하지 않으며 free GlcNAc가 없을 때 활성 부위에 결합하는 첫 번째 분자가 UDP-GlcNAc임을 시사했다. CHS35의 nonreducing end-chain elongation 메커니즘과 함께 기판 결합 포켓의 공간적 제약은 두 개의 UDP-GlcNAc 분자가 활성 부위에 동시에 결합하여 반응을 시작할 가능성을 배제한다. 비록 GlcNAc가 PsChs1(Extended Data Fig. 2e)에 대한 긍정적인 이펙터는 아니지만, chito-oligosaccharides 대신 free GlcNAc를 첨가하면 합성이 빨라질 수 있다(Extended Data Fig. 2f). 이러한 결과는 GlcNAc가 chitin 생합성의 초기 단계에 대한 수용체임을 시사한다. 마찬가지로, 효모 chitin 합성효소 및 셀룰로오스 합성효소에 대해 ‘ self-priming’ 메커니즘이 제안되었으며, 매우 최근에 히알루론산 synthase의 viral homologue에 대해 제안되었다.
우리의 구조 데이터와 이전에 발표된 셀룰로오스 합성 효소 구조는 homodimeric 또는 올리고머 효소의 각 프로모터가 single sugar chain을 independently and in parallel로 합성하고 translocate한다는 것을 보여주었다. 셀룰로오스 생합성에 대한 최근의 구조적 통찰은 당 단위체의 180° alternating 배열을 글리코사이드 결합을 중심으로 terminal sugar unit의 simple rotation에 의해 달성할 수 있으며, dual substrate-binding site의 필요성을 제거한다고 설명했다.
chitin polymer elongation은 SN2 displacement reaction을 통해 donor substrate로부터 acceptor substrate에 당분을 첨가하는 단계를 반복함으로써 달성되는 것이 분명하다. 길쭉한 chitin 고분자는 머리끝에서 translocating channel을 통해 transmembrane domain의 extracellular side로 배출되도록 되어 있다. 우리의 PsChs1 구조는 이 채널의 입구가 apo 및 substrate-bound state에서 swinging loop, 특히 residue Pro454에 의해 차단되지만 (GlcNAc)3 -bound state에서 열려 있음을 밝혔다. 따라서, swinging(VLPGA) loop는 nascent chitin이 세포막을 가로질러 직접 운반되는 것을 용이하게 하는 gate lock 역할을 할 수 있다(그림 3b, e). gate lock loop가 닫힌 상태에서 촉매 센터와 채널 입구 주변의 공간은 단일 유닛 GlcNAc 수용체만 배치할 수 있습니다. VLPGA 루프에서 gate lock을 열고 GlcNAc 유닛이 수용체 GlcNAc(UDP-GlcNAc에서 (exogenously 추가되거나 유도된)에 합류하여 GlcNAc dimer를 형성할 때 nascent disaccharide의 채널에 대한 액세스를 허용하기 위해 큰 구조 변화가 필요하다. 게이트 잠금 장치는 생합성이 완료되고 chitin 사슬이 배출될 때까지 chain elongation 동안 열린 상태로 유지되어야 한다.
5가지 PsChs1 구조와 생화학적 데이터를 종합하여 Fig. 5와 같이 chitin 생합성 모델을 제안한다. 또한 합성된 chitin 사슬은 pyranose ring의 소수성 표면을 통해 a축(pyranose ring에 수직)을 따라 평행하게 정렬되어 chitin 시트를 형성할 수 있으며, chitin 시트는 b축(pyranose ring에 평행)을 따라 반평행 방식으로 자가 조립되어 최종적으로 더 안정한 chitin allomorph인 α-chitin을 형성할 수 있다(Extended Data Fig. 9h). dimeric PsChs1은 단일 사슬의 parallel 배열에 의한 chitin 시트의 형성과 일치한다.
Fig. 5: A model of chitin biosynthesis.
(a) apo enzyme 내, chitin-translocating channel의 입구는 gate lock loop에 의해 차단된다.
(b) Chitin synthesis은 donor substrate UDP-GlcNAc가 reaction chamber로 uridine-binding tub에 상주하면서 시작된다.
(c,d) UDP-GlcNAc hydrolysis (self-priming mechanism) 또는 exogenously 첨가된 GlcNAc가 chitin biosynthesis를 개시하는 acceptor 역할을 하는 것이 제안된다. 이 프로세스에는 다음 단계가 포함되어야 한다. Glu495는 donor substrate에 결합하고 안정화시키며, catalytic residue Asp496은 donor substrate에 대한 nucleophilic attack을 보조한다. 2가 metal ion은 donor substrate의 diphosphate group에 결합하여 disaccharide formation을 완료하기 위해 UDP moiety 방출을 돕는다. disaccharide formation은 gate lock loop와 Pro454의 flipping에 conformational changes를 유도하여 입구를 열고 nascent sugar chain을 translocating channel로 접근할 수 있게 할 가능성이 있다.
(d) sugar unit의 첨가가 완료되면 catalytic Glu495와 leaving UDP의 metal ion-bound diphosphate은 반응 중심에서 떨어져 회전한다.
(e) 여러 번의 reaction rounds (대시 화살표) 이후, 효소는 post-synthesis state를 채택하고 chitooligosaccharide product를 배출한다: translocating channel은 gate lock에 의해 닫히고, substrate-binding site에 앉아 있는 leaving group UDP는 new donor substrate로 치환될 필요가 있다. 그리고 Glu495와 catalytic residue Asp496의 위치는 chitin biosynthesis의 새로운 사이클을 시작하기 위해 pre-synthesis state로 복원된다.