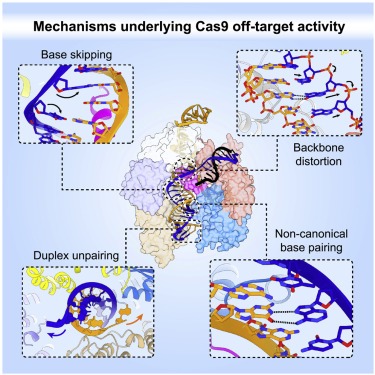
Cas9 off-target activity의 구조적 기반
Abstract
CRISPR-associated genome editor nuclease Cas9의 target DNA 특이성은 가이드 RNA에서 20-nucleotide segment에 대한 상보성에 의해 결정된다. 그러나 Cas9는 부분적으로 상보적인 off-target sequence를 결합하고 절단할 수 있으므로 임상 응용에 대한 안전성에 대한 우려를 제기한다. 여기서, 우리는 bona fide off-target substrate에 결합되는 Cas9의 결정학적 구조를 보고하여, off-target heteroduplex라는 가이드 내의 다양한 비표준 염기쌍 상호 작용에 의해 off-target 결합이 가능하다는 것을 밝힌다. 가이드 RNA에 대한 single-nucleotide deletion을 포함하는 Off-target substrate는 RNA bulge formation보다는 base skipping 또는 multiple noncanonical base pair에 의해 수용된다. 마지막으로, PAM- distal mismatch는 duplex unpairing을 초래하고 Cas9 REC 로브의 입체구조 변화를 유도하여 입체구조 활성화를 방해한다. 이러한 통찰력은 Cas9의 off-target에 대한 구조적 근거를 제공하고 가이드 RNA 및 off-target 예측 알고리즘의 개선된 합리적 설계에 기여한다.
Figure
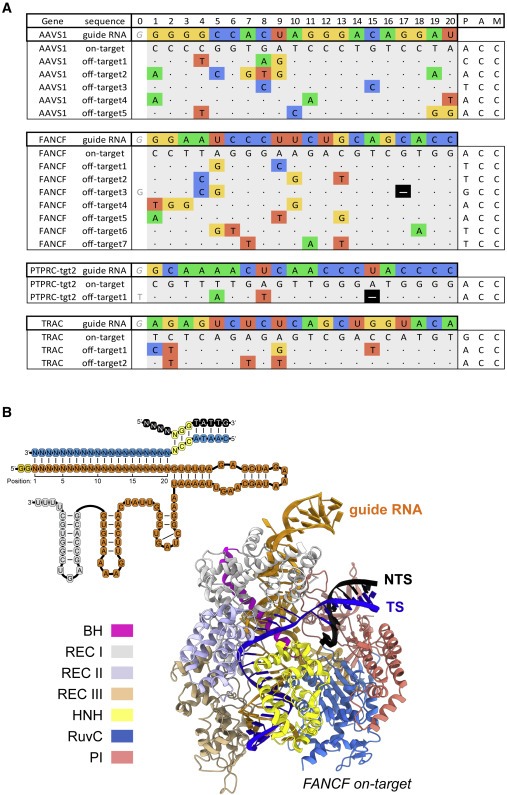
Figure 1. Cas9 off-target의 biochemical 및 structural analysis
(A) 생화학 및 구조 분석을 위해 선택된 Guide RNA 및 (오프) target DNA sequence.
(B) 위: 결정화에 사용되는 guide RNA (주황색), target DNA strand (파란색) 및 non-target DNA strand (검은색) 시퀀스의 개략적인 표현.
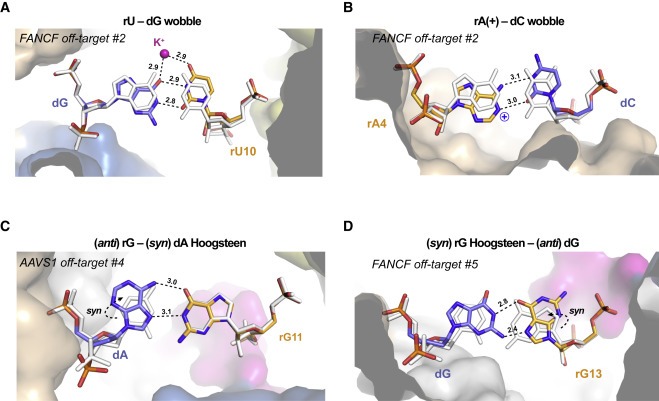
Figure 2. Cas9 off-target binding은 noncanonical base pairing에 의해 활성화된다.
(A-D) Close-up view
(A) FANCF off-target #2 complex의 duplex position 10에서 rU-dG wobble base pair
(B) FANCF off-target #2 complex의 duplex position 4에서 rA-dC wobble base pair
(C) AAVS1 off-target #4 complex의duplex position 11에서 rG-dA Hoogsteen base pair
(D) FANCF off-target #5 complex의 duplex position 13에서 rG-dG Hoogsteen base pair
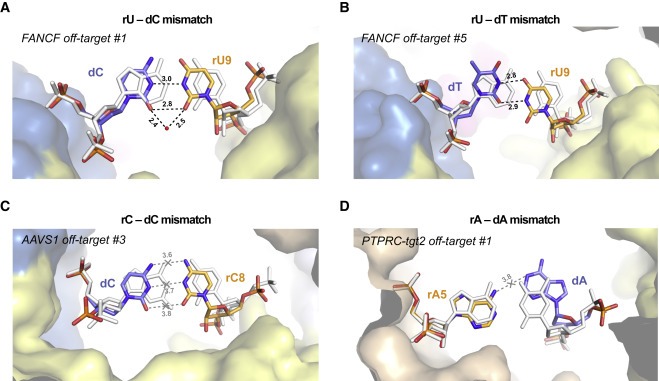
Figure 3. Duplex backbone distortion은 noncanonical base pair의 형성을 용이하게 한다.
(A-D) Close-up view
(A) FANCF off-target #1 complex의 duplex position 9에서 rU-dC base pair
(B) FANCF off-target #5 complex의 duplex position 9에서 rU-dT base pair
(C) AAVS1 off-target #3 complex의duplex position 8에서 rC-dC mismatch
(D) PTPRC-tgt2 off-target #1 complex의 duplex position 5에서 rA-dA mismatch
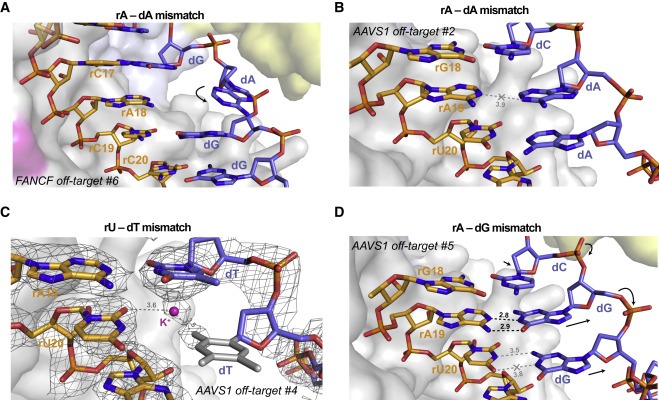
Figure 4. TS distortion은 guide:off-target heteroduplex의 seed region에서 mismatch accommodation을 용이하게 한다.
(A-C) Close up view
(A) FANCF off-target #6 complex의 position 18에서 rA-dA mismatch
(B) AAVS1 off-target #2 complex의 position 19에서 rA-dA mismatch
(C) AAVS1 off-target #5 complex의 position 19에서 rA-dG base pair와 position 20에서 the unpaired rU-dG mismatch
(D) AAVS1 off-target #4 complex의 PAM-proximal position 20에서 rU-dT mismatch의 Close-up view.
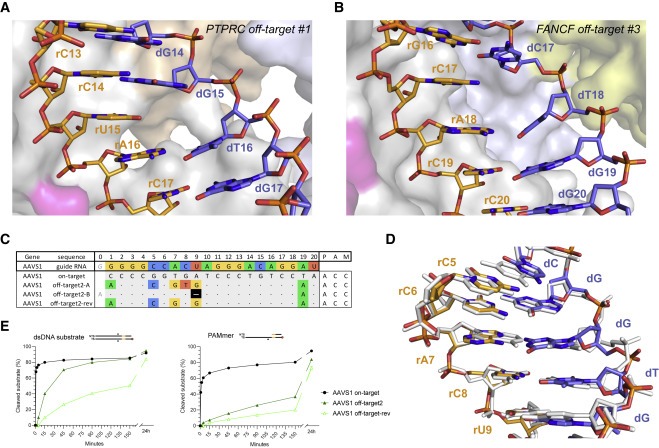
Figure 5. Off-targets with single-nucleotide deletion은 base skipping이나 multiple mismatch를 통해 수용된다.
(A) PTPRC-tgt2 off-target #1 complex의 duplex position 15에서 base skip의 Zoomed-in view
(B) FANCF off-target #3 complex의 duplex position 17에서 base skip의 Zoomed-in view
(C) AAVS1 off-target #2 complex (off-target2-A and off-target2-B)의 alternative base-pairing mode에 대한 개략적인 설명
(D) AAVS1 off-target #2 (colored)와 AAVS1 on-target (white) heteroduple의 Structural overlay
(E) AAVS1 on-target, off-target #2, and off-target #2-rev DNA substrate의 Cleavage kinetics.
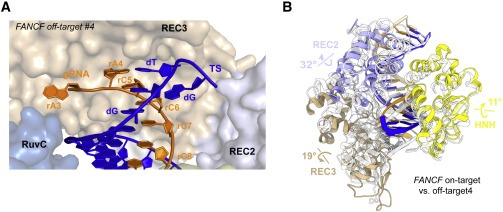
Figure 6. PAM-distal heteroduplex의 mismatch-induced unpairing시 Cas9 conformational rearrangement.
(A) FANCF off-target #4 heteroduplex의 the PAM-distal end에서 mismatched base unpairing에 대한 Close-up view.
(B) FANCF off-target #4 와 FANCF on-target complex structure의 Overlay.
Discussion
Cas9의 off-target activity는 이전의 게놈 편집, 생화학 및 생물물리학 연구(Boyle et al., 2017; Boyle et al., 2021; Doench et al., 2016; Jones et al., 2021; Lazzarotto et al., 2020; Zhang et al., 2020)에서 광범위하게 문서화되어 있다. genomic off-target sit의 계산 예측과 실험 검증을 위해 수많은 방법이 고안되었지만, 이는 선별 방법과 대상 순서에 따라 highly variable mismatch tolerance profile을 보고하였다. 따라서 이 현상에 대한 포괄적인 이해가 여전히 부족하다. 이 연구에서, 우리는 SITE-Seq assay를 사용하여 잘 연구된 12개의 gRNA의 off-target landscape를 조사했고, 개별 off-target substrate와 관련된 광범위한 cleavage activity를 관찰했다. off-target activity의 기초가 되는 분자 메커니즘을 밝히기 위해, 우리는 off-target recognition의 구조적 측면에 대한 근본적인 통찰력을 제공하는 16개의 off-target complex의 대표적인 세트의 원자 구조를 결정했다.
Limitations of the study
본 연구에서 제시된 구조적 데이터 세트는 반드시 범위가 제한된다. 모든 불일치 유형이 다루어지기는 하지만, Cas9의 intrinsic off-target activity를 뒷받침하는 mismatch tolerance에 대한 완전한 구조적 개요를 달성하기 위해서는 모든 위치에서 모든 mismatch를 포괄하기 위해 훨씬 더 많은 수의 off-target complex structure이 필요할 것이다. MD 시뮬레이션이 실험 데이터의 공백을 메우는 데 도움이 될 수 있지만, 우리의 연구 결과는 mismatch accommodation이 시퀀스 컨텍스트에 의존하는 것처럼 보여서 예측력을 제한한다는 것을 시사한다. single-nucleotide deletion을 포함하는 off-target substrate에 대한 통찰력을 제공하지만, 현재 주어진 heteroduplex 위치에서 어떤 메커니즘이 발생하는지 예측하는 것은 어렵다. 더욱이, 우리는 현재 insertion이 포함된 off-target substrate에 대한 구조 데이터가 부족하며, 이는 향후 연구에서 다루어질 것이다. 마지막으로, 모든 구조적으로 특징지어지는 off-target (~50%)이 in vitro에서 효율적으로 절단되는 것은 아니며, 이는 추가적인 요인이 진핵생물 게놈의 맥락에서 Cas9 off-targeting에 영향을 미친다는 것을 의미한다. 따라서 Cas9의 off-target activity를 정확하게 예측하기 위해 구조 및 계산 접근 방식을 결합한 추가 작업이 필요할 것이다.