human gut crassvirus의 structural atlas.
Abstract
CrAssphage 및 Crassvirales (이하 crassviruses)의 관련 바이러스들은 원래 metagenomic sequences의 cross-assembly에 의해 발견되었습니다. crassviruses는 인간 장에서 가장 풍부한 바이러스이며, 대부분의 individual gut viromes에서 발견되며, 일부 개인에서 바이러스 서열의 최대 95%를 차지합니다. Crassviruses는 human microbiome의 구성과 기능을 형성하는 데 중요한 역할을 할 가능성이 높지만, 대부분의 virally encoded proteins의 구조와 역할은 알려지지 않았으며 생물정보학적 분석을 통한 일반적인 예측만 가능합니다. 여기에서는 Bacteroides intestinalis virus ΦcrAss001의 cryo-electron microscopy reconstruction을 통해 대부분의 virion proteins의 기능적 할당에 대한 구조적 근거를 제시합니다. muzzle protein은 tail 끝에서 약 1 MDa 크기의 assembly를 형성하고 이전에 알려지지 않은 ‘crass fold’라고 명명 한 접힘을 나타내며, 이는 cargos ejection을 제어하는 게이트 키퍼 역할을 할 가능성이 높습니다. ΦcrAss001 virion은 약 103kb의 virus DNA를 포장하는 것 외에도 capsid와 특이하게도 tail 내에 바이러스로 인코딩된 cargo proteins을 위한 광범위한 저장 공간을 가지고 있습니다. cargo proteins 중 하나는 capsid와 tail 모두에 존재하며, 이는 단백질이 tail을 통해 extrusion되는 동안 단백질이 부분적으로 unfolding 되는 일반적인 protein ejection 메커니즘을 시사합니다. 이러한 발견은 고도로 풍부한 crassviruses의 조립 및 감염 메커니즘을 이해하는 데 구조적 기반을 제공합니다.
Figure
Fig. 1: ΦcrAss001 virion의 구조 및 functional assignments.
a,b, negative staining (a)과 cryogenic conditions (b)을 사용하여 기록한 virions의 Electron micrographs.
c, 외부에서 바라본(왼쪽) 및 z- clipped (오른쪽) virion reconstruction의 분자 표면을 gene product에 따라 색칠한 모습.
d, 위, ΦcrAss001 genome (accession: NC_049977.1) 영역 11643-68717의 open reading frames이 유전자 길이에 따라 크기가 조정된 c의 gene product (gp)에 해당하는 색으로 표시된 화살표로 표시됩니다. 아래쪽, gene conservation, 각 막대의 길이는 검출 가능한 homologue를 포함하는 Crassvirales 순서대로 각 crassvirus group의 비율을 나타내며, c에 해당하는 색으로 표시됩니다.
Fig. 2: ΦcrAss001 capsid proteins.
a, symmetry axes (회색 모양)가 표시된 icosahedral capsid의 asymmetric subunit의 Molecular surface, major capsid protein gp32 (노란색), capsid auxiliary protein gp36 (진한 파란색), head fibre proteins gp21 (HFT; 옅은 파란색, trimer) 및 gp29(HFD; 분홍색, dimer)가 있습니다.
b,c, 주요 capsid protein subunit (b)과 capsid auxiliary protein gp32 (c)의 Ribbon diagrams.
d, C3 capsid hexon의 Molecular surface.
e, HFT trimer의 Ribbon diagram.
f, the skewed hexon의 Molecular surface.
g, HFD dimer의 Ribbon diagram.
Fig. 3: The portal protein and the surrounding capsid vertex.
a, ribbons으로 표시된 portal protein (하늘색)과 C1 cargo protein (보라색)의 두 개의 반대되는 subunits.
b, channel axis를 따라 본 a에 표시된 단백질의 Dodecameric assembly.
c, C1 cargo protein subunits (보라색)과 interacting하는 portal protein wing region (하늘색).
d,e, major capsid protein gp32 (노란색), auxiliary capsid protein gp36 (진한 파란색) 및 portal protein gp20 (리본, 하늘색)으로 구성된 portal-containing capsid vertex의 Molecular surface, central channel을 따라 capsid 내부에서 본 모습(d) 및 portal vertex auxiliary protein gp57이 녹색, head fibre protein gp21이 연한 파란색으로 표시된 capsid 외부에서 본 모습(e).
f, 12개의 subunits이 중첩된 모습(왼쪽)과 central axis을 따라 본 oligomer (오른쪽)를 나타내는 portal protein의 리본 다이어그램으로, capsid-adaptable loops가 각각 하늘색과 빨간색으로 표시되어 있으며 각각 형태 A와 B에 해당합니다.
g, gp57(PVA) protein dimer가 녹색 리본으로 표시된 C5 reconstructed portal-containing capsid vertex의 Unit.
h, inter-subunit disulfide bonds가 있는 gp57 dimer가 노란색으로 표시되어 있습니다.
Fig. 4: The tail barrel and fibre docking hubs.
a, ribbon diagram으로 표시된 tail barrel의 두 개의 반대쪽 ring proteins chain, 위에서 아래로: R1(gp43)(연한 청록색), R2(gp40)(남색), R3(gp35)(중간 녹색), R4(gp34) 및 R5(gp34)(연한 녹색), THA(gp38)(주황색), THB(gp39)(분홍색) 및 gp22(짙은 회색)입니다.
b, 각 ring protein alongside의 Individual subunits과 dodecameric ring assemblies를 central axis를 따라 본 모습.
c, capsid end에서 central axis를 따라 본 12개의 tail hub assemblies로 형성된 칼라로, ribbon diagram에서 하나의 tail hub chain이 THA1 및 THA2는 주황색으로, THB는 분홍색으로 표시되어 있습니다.
d, c에서와 같이 collar가 90° 회전하고 확대되어 연동되는 tail hub가 표시됩니다.
e, tail hub proteins과 R1 및 R2 proteins 사이의 disulfide bonds (노란색, 공 모양, 막대 모양)을 확대한 모습.
Fig. 5: The muzzle assembly.
a, 위, ribbons (빨간색)으로 표시된 muzzle protein의 두 개의 반대편 subunits. 아래, cargo protein 이 보라색으로 표시된 central axis를 따라 본 muzzle의 hexamer.
b, 주황색은 ring-joining domain, 녹색은 β- propeller domain, 분홍색은 immunoglobulin-like domain 1(IG1), 청록색은 IG2, 하늘색과 노란색은 crass domain polypeptide segments인 muzzle protein의 single subunit (중앙 터널 축을 중심으로 90° 회전된 오른쪽 사슬).
c, b와 같이 도메인이 색칠된 muzzle protein의 Topology diagram.
d, 개별 도메인이 b,c와 같이 색칠된 muzzle protein의 모식도.
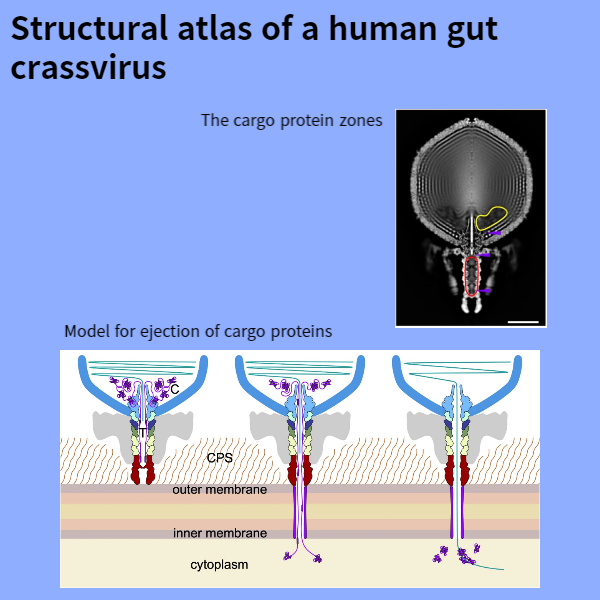
Fig. 6: The cargo protein zones.
a, rotationally-averaged virion map의 Z-clipped view.
b-d, C1(gp45)의 portal-bound domain (b), C1의 tail barrel-bound region (c) 및 muzzle-bound cargo protein fragment (d)의 구조가 해당 density maps (투명한 회색)과 함께 표시됩니다.
e, virion 내부의 cargo protein locations 모식도.
Disscussion
결론적으로, ΦcrAss001 virion의 reconstruction을 통해 crassviruses의 구조적 특징을 파악하고 comparative genomic analysis과 결합하여 virion assembly and infection에 관여하는 이전에 특성화되지 않은 대부분의 단백질에 기능을 부여할 수 있었습니다. 이러한 구조적 특징 중 새로운 crass fold를 포함하는 muzzle protein은 게이트키퍼 역할을 하며, 이는 아마도 바이러스와 숙주 세포 표면 사이의 접촉에 기여하는 것으로 추정됩니다. 널리 보존된 paralogous ring proteins로 형성된 확장된 tail barrel은 capsid cargo zone과 함께 cargo proteins 저장 용량이 커서 crassvirus infection 감염 초기 단계에 필요한 proteins이 virions 내부로 운반되어 host cell로 전달되도록 보장합니다. tail 부분에 cargo protein storage zone이 있는 것은 지금까지 바이러스에서 관찰된 적이 없는 독특한 특징이며, virus genome과 cargo proteins이 숙주에게 순서대로 방출되도록 compartmentalization되는 메커니즘을 시사합니다.