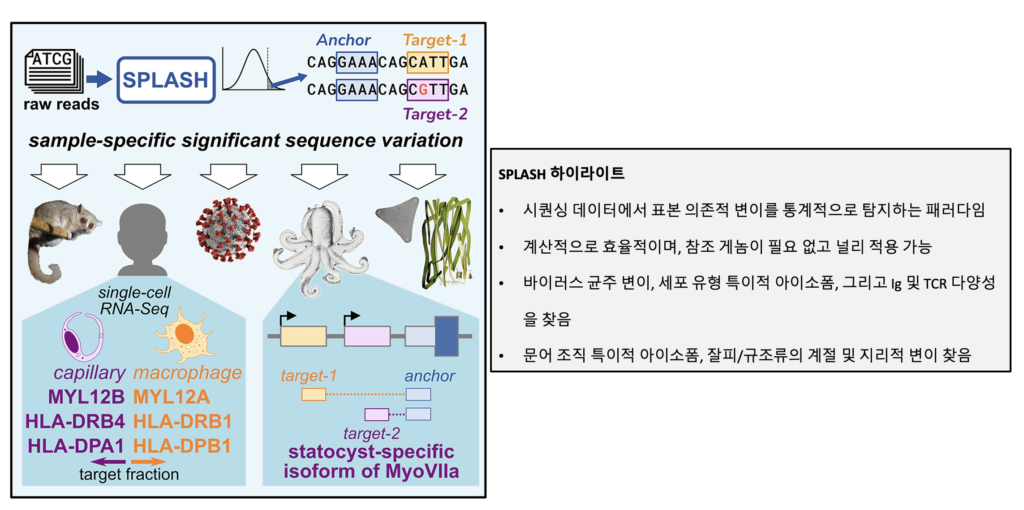
SPLASH: 생물학적 발견을 통합하는 통계적이고 참조 없는 유전적 알고리즘
Abstract
현재의 유전체 분석 워크플로우는 대체로 참조 서열과의 정렬(alignment)을 필요로 하며, 이는 발견을 제한합니다. 우리는 통합적인 패러다임인 SPLASH (Statistically Primary aLignment Agnostic Sequence Homing)를 소개합니다. 이는 원본(raw) 시퀀싱 데이터를 직접 분석하여, 통계적 검정을 사용해 조절 신호(signature of regulation)를 탐지합니다(표본 특이적 시퀀스 변이를 탐지). SPLASH는 다양한 유형의 변이를 탐지하며 대규모로 효율적으로 실행할 수 있습니다. SPLASH는 SARS-CoV-2에서 복잡한 변이 패턴을 식별하고, 단일 세포 수준에서 조절된 RNA 아이소폼을 발견하며, 적응성 면역 수용체(adaptive ummnye receptors)의 광범위한 시퀀스 다양성을 탐지하고, 기존 참조 게놈에서 기록되지 않은 생물체(non-model organisms)의 생물학적 특징을 밝혀냅니다: 기후 변화의 영향을 받는 해양 식물인 잘피의 지리적 및 계절적 변이와 규조류와의 연관성, 그리고 문어의 조직 특이적 전사체가 그 예시 입니다. SPLASH는 메타데이터나 참조 유전체 없이도 광범위한 발견을 가능하게 하는 유전체 분석의 통합적 접근법입니다.
Figure
SPLASH는 샘플에 따른 시퀀스 변이를 식별하기 위한 k-mer 기반, 통계 우선 접근 방식입니다
- SPLASH는 k-mer 기반, 통계 우선 방식으로, 참조 시퀀스에 의존하지 않고 샘플 종속 시퀀스 변이(variation)를 식별합니다.
- 앵커(anchor)와 타겟(target) k-mer 쌍을 분석해 다양한 시퀀스 변이를 파악하며, 각 앵커에 대한 p-value를 통해 샘플 간 변이를 평가합니다.
- 메타데이터 없이도 비감독 모드에서 작동하며, 각 앵커별 효과 크기(effect size)를 계산하여 샘플 그룹 간 명확한 변이를 감지합니다.
- 전통적인 정렬 방식에 비해 계산 부담을 크게 줄이며, 다양한 데이터셋과 매개변수에 강건하게 작동, 중요한 시퀀스 변이 패턴을 효과적으로 발견합니다.
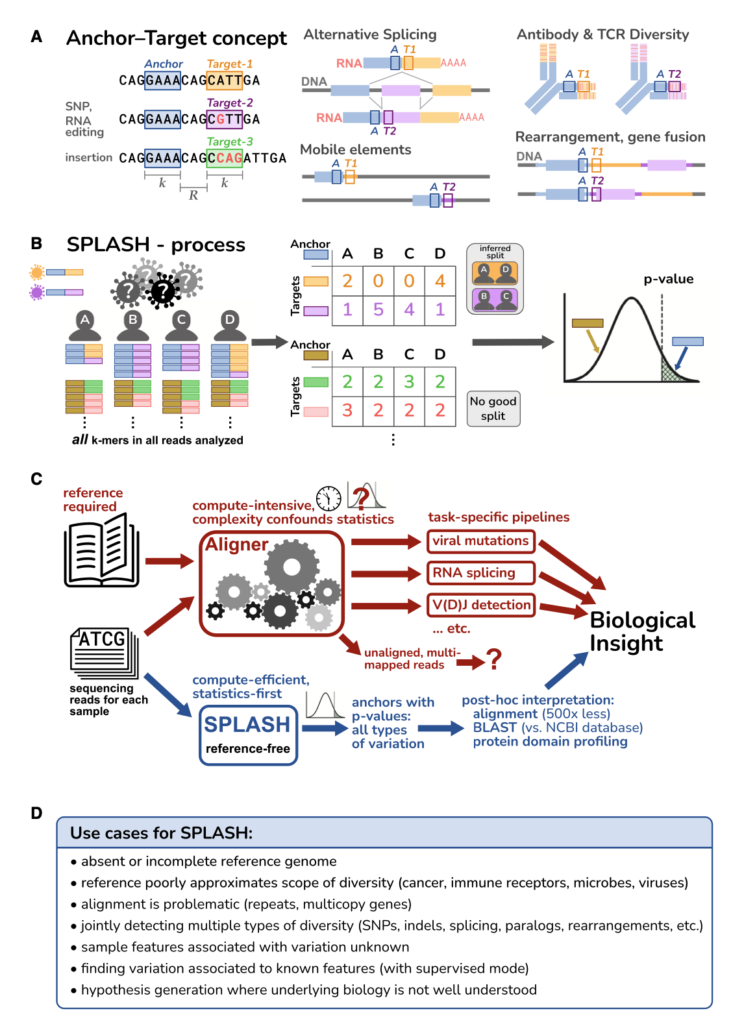
Figure 1. Overview of SPLASH
(A) Anchor- Target의 개념
(B) SPLASH의 동작 과정
(C) 기존 reference based method과의 비교
(D) SPLASH를 활용한 분석 예시
[Figure 1A] 앵커(anchor)는 reads에서 길이 k의 시퀀스(k-mer)이며, 타겟(target)은 고정된 길이 R 후에 따르는 k-mer입니다. 하나의 앵커는 다양한 타겟과 함께 나타나며, 이는 다양한 종류의 변이를 포착할 수 있습니다; 예시는 앵커를 파란 상자로, 타겟을 주황색 또는 보라색 상자로 도식적으로 나타냅니다.
[Figure 1B] SPLASH는 각 앵커에 대해 테이블을 만듭니다. 여기서 열은 샘플, 행은 타겟, 항목은 각각의 발생 횟수입니다. SPLASH는 샘플의 다양한 무작위 분할을 테스트하며, 각 샘플의 타겟 분포와 모든 샘플의 평균 타겟 분포 사이의 편차를 측정하는 통계치를 계산하여 가장 구별되는 분할을 찾습니다. 가장 좋은 분할을 식별한 후, SPLASH는 p-value bound를 보고합니다.
[Figure 1C] 정렬 기반 파이프라인은 참조 게놈의 필요성, 계산 집약도, 복잡성으로 인한 통계적 모델링의 어려움에 의해 제한됩니다. SPLASH는 엄격한 통계를 사용하여 원본 reads에서 직접 변이(variation)를 탐지하며, 계산 효율적이고 한 번에 많은 종류의 변이를 탐지합니다.
SPLASH는 SARS-CoV-2에서 변이주를 정의하고 기타 돌연변이를 de novo로 식별합니다.
- SPLASH는 참조 시퀀스나 샘플 메타데이터 없이도 SARS-CoV-2의 변이주를 정의하고 기타 중요한 변이(variation)를 직접 시퀀싱 읽기에서 효과적으로 식별하며, 샘플 간의 변이 차이를 높은 정밀도로 감지합니다.
- Delta와 Omicron 변이를 포함한 SARS-CoV-2의 주요 및 하위 계통을 구분하며, k-mer 쌍(앵커와 타겟)을 분석하여 spike protein과 기타 영역에서 변이주를 정의하는 변이를 성공적으로 매핑합니다.
- SPLASH의 결과는 참조 게놈 없이도 추가 분석될 수 있으며, consensus sequences를 아미노산으로 변환하고 이를 protein domain 데이터베이스와 매치하여 바이러스와 관련된 단백질의 중요한 변이 패턴을 식별합니다.
- 다양한 바이러스에 적용될 때 SPLASH는 바이러스 단백질 도메인의 변이와 면역 시스템과의 상호작용을 이해하는 데 도움이 될 수 있는 반복되는 테마를 드러냅니다. 이는 새로운 변이주나 신종 병원체에 대한 감시와 클러스터링에 유용할 수 있습니다.
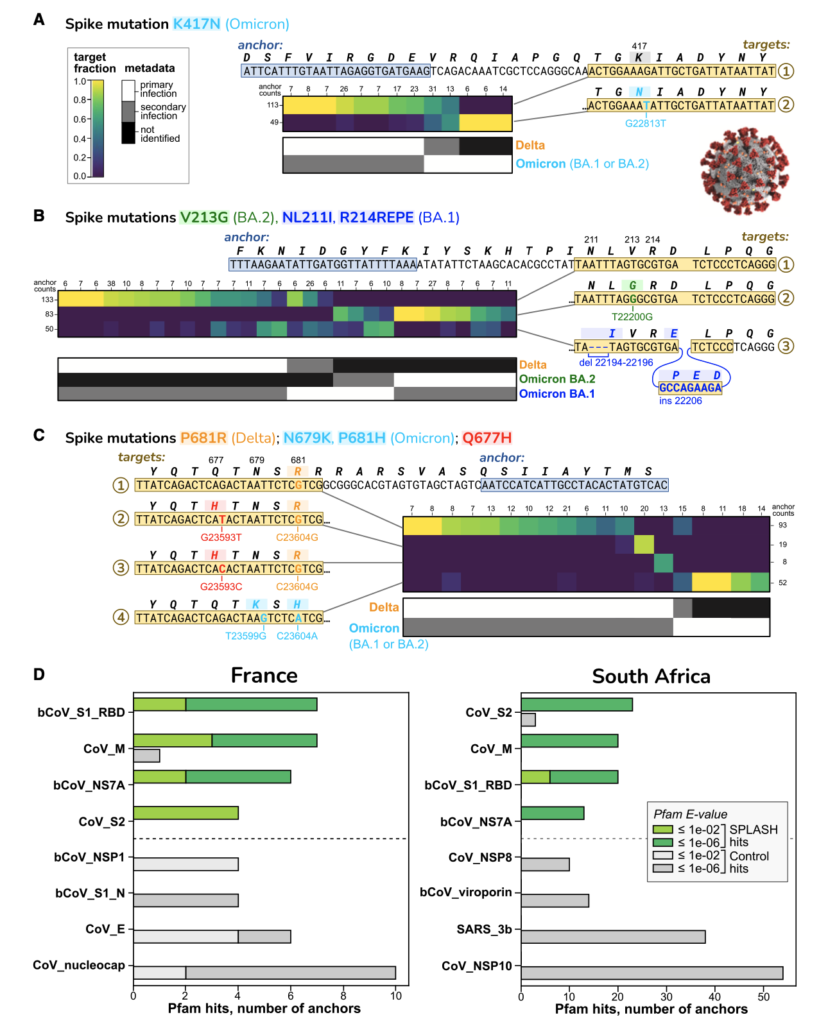
Figure 2. SPLASH는 SARS-CoV-2에서 변이주를 정의하는 변이(variations) 및 기타 변이들을 식별합니다.
(A)-(C) SARS-CoV-2 변이주를 구분하는 타겟 sets
(D) SPLASH 앵커에 대한 단백질 도메인의 분포는 통계적으로 대조군과 다릅니다.
[Figure 2A-2C] (A)–(C)에서는 SARS-CoV-2 변이주를 구분하는 타겟 세트가 보여지며, 모두 스파이크 단백질(S) 유전자에 있습니다. 각 히트맵에는 다른 샘플(환자)을 위한 열과 다른 타겟을 위한 행이 있으며, 색상은 해당 환자에서 관찰된 해당 타겟의 비율을 나타냅니다. 행과 열에 대한 요약 앵커 수가 제공됩니다. 또한, 원래 연구에서 각 환자에서 식별된 변이주(1차 및 2차)에 대한 범주형 메타데이터의 지도도 표시됩니다; 이 데이터는 SPLASH에 의해 사용되지 않았지만, 히트맵과 메타데이터 변이주 할당 사이에 명백한 일치가 있습니다.
[Figure 2A] 주요 변이주 수준에서 구분하기: 타겟 1(변이 없음)은 Delta와 일치; 타겟 2는 모든 Omicron(바로 BA.1 및 BA.2 하위 변이주)에서 발견되는 K417N을 포함; Delta와 Omicron에 동시 감염된 두 환자는 두 타겟 모두 보임 (p = 6.4E-07).
[Figure 2B] 하위 변이주 수준에서 구분하기: 타겟 1(변이 없음)은 Delta와 일치; 타겟 2는 V213G으로 BA.2 특유; 타겟 3은 삭제(NL211I)와 삽입(R214REPE)으로 BA.1 특유 (p = 1.0E-13).
[Figure 2C] 변이주와 관련 없는 변이 구분하기: 타겟 1은 P681R로 Delta 특유; 타겟 2와 3은 Q677H(서로 다른 변이에 의해)와 P681R을 인코딩; 타겟 4는 Omicron 특유인 N679K와 P681H을 가짐 (p = 4.9E-12).
[Figure 2D] SARS-CoV-2에서의 단백질 도메인 프로파일링. 상위 4개와 하위 4개 Pfam 단백질 도메인이 표시됩니다. S1 receptor-binding domain (RBD)과 S2 도메인은 두 데이터셋에서 SPLASH에 의해 높은 변이를 보여줍니다.
SPLASH는 단일 세포 RNA-seq에서 paralogs와 HLA의 조절된 발현을 식별합니다.
- SPLASH는 다양한 인간 세포에서 단일 세포 수준의 alternative splicing, 특정 paralogs 및 HLA 유전자의 조절된 발현을 효과적으로 식별하여 복잡한 유전적 변이를 감지하는 강력한 능력을 입증합니다.
- 특히 유사한 유전자 영역, 예를 들어 paralogs 간 또는 다형성이 높은 HLA 영역 내에서 세포 유형별 특정 발현 패턴을 구분하며, 참조 게놈의 한계에 의해 제시된 도전을 극복합니다.
- 이 방법은 T세포와 대식세포에서 HLA 유전자의 allele 특이적 발현 패턴을 밝혀내어, 다른 세포 유형에서 개별 allele이 어떻게 발현되는지에 있어 상당한 variation을 나타냅니다.
- SPLASH의 단일 세포 수준에서 복잡한 유전 및 전사적 조경을 식별할 수 있는 능력은 세포 특이적 및 질병 관련 유전 조절을 이해하는 데 있어 그 잠재력을 보여줍니다.
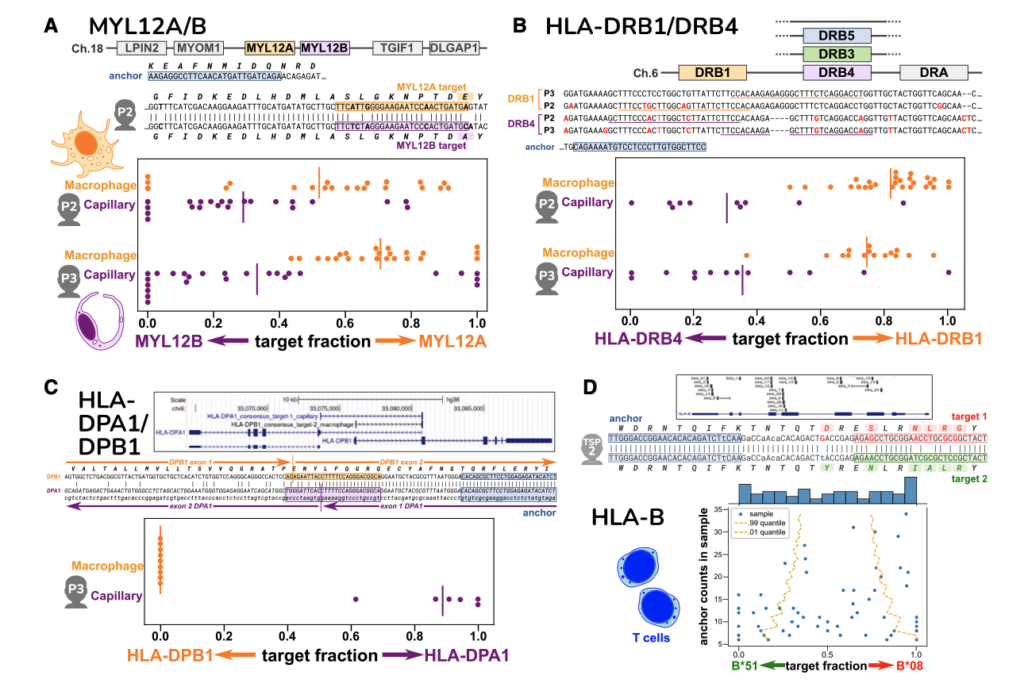
Figure 3. 단일 세포 데이터에서 paralogs와 HLA의 세포 유형별 특이적 발현
(A)-(C)는 점 플롯을 보여주며, 여기서 각 점은 단일 세포에서 상대적 아이소폼 발현을 나타내고 막대는 평균 상대 발현을 표시합니다
(D) 세포들의 다양한 allele 발현 비율에 대한 산점도
[Figure 3A] 인간의 MYL12A와 MYL12B는 18번 염색체에 인접하여 있으며, 포유류, 닭, 파충류에서 유사한 구조(syntenic)를 갖습니다. 서열 alignment은 두 유전자가 코딩 영역에서 매우 유사함을 보여주며, 개인 P2의 앵커와 두 타겟을 표시합니다(P3는 구별되는 앵커를 가짐). 대식세포는 상대적으로 더 많은 MYL12A를, 모세혈관 세포는 더 많은 MYL12B를 발현하며, 두 개인에서 일관성 있게 나타납니다.
[Figure 3B] HLA-DRB 위치는 여러 다른 합성형(haplotypes)으로 나타나며, 모두 DRB1을 포함하지만 paralogs(DRB3, DRB4, DRB5 또는 없음; hg38 참조는 DRB5를 가짐)가 다릅니다. 앵커와 그 타겟(밑줄 친 부분)은 DRB1과 DRB4의 3′ UTR에 위치하며, 두 개인은 DRB1과 DRB4 모두에서 구별되는 allele을 가집니다. 대식세포는 주로 DRB1을, 모세혈관 세포는 주로 DRB4를 발현합니다.
[Figure 3C] HLA-DPA1과 HLA-DPB1은 UCSC Genome Browser 다이어그램에 표시된 바와 같이 서로 반대 방향으로 배열되어 겹칩니다. 이는 또한 DPA1과 DPB1에 대한 앵커 합의의 BLAT 매핑을 보여주며, 이들은 반대 방향의 가닥에 위치합니다. 타겟은 반대 방향의 가닥에 가장 잘 할당됩니다. DPA1과 DPB1 모두에 대한 보고를 하는 앵커는 개인 P3에 대해서만 발견되었으며, 그 타겟은 대식세포가 독점적으로 DPB1을, 모세혈관 세포가 주로 DPA1을 발현함을 보여줍니다.
[Figure 3D] 다형성 HLA-B 유전자는 T 세포에서 많은 SPLASH 앵커를 포함하고 있으며, UCSC Browser 다이어그램에 나타납니다. hg38 참조는 B07:02 알렐이지만, 이 개인은 B08과 B*51(합의 서열 1과 2)에 가장 잘 일치합니다. 우리는 HLA-B의 앵커 중 하나를 조사했는데, 이는 exon 2에 위치합니다.Alignment에서, 앵커와 lookahead 영역에서 hg38과의 차이는 소문자로 표시됩니다. 산점도에서 세포들은 다양한 allele발현 비율을 보여주며, 일부는 단일 allele만을 발현합니다. 점선은 인구 평균 발현을 기반으로 한 이항 분포의 98% 신뢰 구간을 표시합니다(신뢰는 앵커 수에 따라 달라짐); 관찰된 데이터는 현저하게 이탈합니다(이항 p = 1.73E-25), 그리고 일부 세포는 거의 독점적으로 하나의 allele만을 발현합니다..
SPLASH는 단일 세포 RNA-seq에서 paralogs와 HLA의 조절된 발현을 식별합니다.
- SPLASH는 다양한 인간 세포에서 단일 세포 수준의 alternative splicing, 특정 paralogs 및 HLA 유전자의 조절된 발현을 효과적으로 식별하여 복잡한 유전적 변이를 감지하는 강력한 능력을 입증합니다.
- 특히 유사한 유전자 영역, 예를 들어 paralogs 간 또는 다형성이 높은 HLA 영역 내에서 세포 유형별 특정 발현 패턴을 구분하며, 참조 게놈의 한계에 의해 제시된 도전을 극복합니다.
- 이 방법은 T세포와 대식세포에서 HLA 유전자의 allele 특이적 발현 패턴을 밝혀내어, 다른 세포 유형에서 개별 allele이 어떻게 발현되는지에 있어 상당한 variation을 나타냅니다.
- SPLASH의 단일 세포 수준에서 복잡한 유전 및 전사적 조경을 식별할 수 있는 능력은 세포 특이적 및 질병 관련 유전 조절을 이해하는 데 있어 그 잠재력을 보여줍니다.
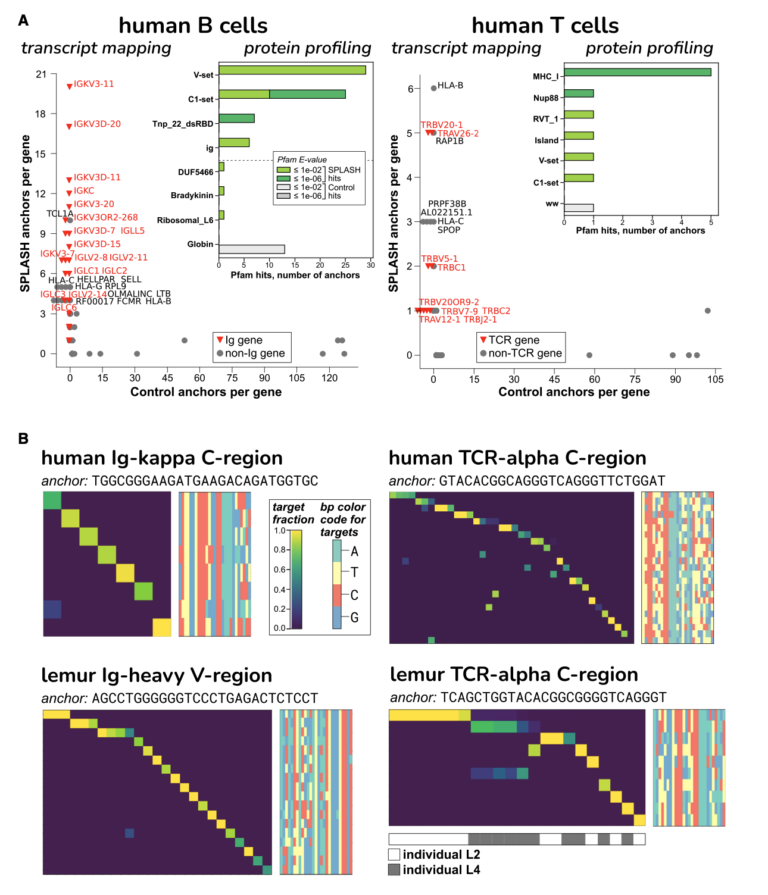
Figure 4. low- and high-affinity BCRs을 발현하는 4GC B 세포가 plasma cells로 발달하는 과정.
(A, C, E, F) recombinant HA에 대한 monovalent Fab binding의 Affinities (KD).
(A) Figure 2의 Ms#1, Ms#3, Ms#5에 대한 다양한 clones에서 채취한 GC B 세포 및 PC의 Fab KD.
(B) Ms#1의 PC Fab에서 얻은 SPR single-cycle kinetic traces 예시.
(C) 표시된 expanded clonal lineages의 GC B 세포 및 PC에 대한 Fab KD.
(D) 3 days-tamoxifen treatment 후 감염 후 14일 또는 21일째에 채취한 MedLN의 PC generation efficiency (1,000개의 tdTom+ GC B 세포당 tdTom+ PC의 수).
(E) 3 days-tamoxifen treatment 후 감염 후 21일째에 분류된 GC B 세포 및 PC의 Fab KD.
(F) 다양한 clonal lineage의 day 21 post-infection PCs의 median KD를 추론된 UCA의 KD와 비교.
[Figure 4A, B] 서로 다른 lineages의 개별 GC B 세포는 antibody affinity가 크게 달랐으며(최대 1,000배), 대부분 100~200배 범위에 걸쳐 있었음. 놀랍게도 tdTom+ PC에서 관찰된 affinity spread는 GC와 유사.
- 중요한 것은 weak affinity BCR을 가진 GC B 세포가 higher-affinity antigen receptors를 발현하는 세포와 경쟁할 때도 differentiated될수 있다는 것을 의미.
[Figure 4C] clonal lineages 내에서 BCR affinity와 PC differentiation 사이의 관계를 조사.
Individual clones은 예상대로 상당한(∼10~30배) affinity ranges를 지원했지만 lineages 간에는 덜 broad함.
somatic variants와 UCA를 비교한 결과, GC가 positive selection을 통해 비교적 완만한 affinity enhancements을 읽고 촉진한다는 사실이 입증(예: Ms#1 clone i, Ms#3 clone vii, Ms#5 clone xiii). 그럼에도 불구하고, 상대적으로 넓은 범위의 clone (예: Ms#3, KD가 ∼40배에 이르는 clone ix)과 lower-affinity의 clone (예: Ms#1 clone iii 및 Ms#5 clone xi)을 포함하여, tdTom+ PC Fab은 동일한 lineages의 GC B 세포와 affinities 측면에서 거의 유사하게 나타남.
[Figure 4D] GC 크기로 normalized된 Overall PC output은 약 4배 감소했으며, 이로 인해 뚜렷한 lineages 수와 somatic variants 모두에서 tdTom+ PC 다양성이 약간 감소함.
[Figure 4E] overall response affinity는 maturation 1주일이 추가되면서 훨씬 개선되었지만(∼600nm 범위와 비교하여 ∼20nm), large interclonal affinity spreads는 여전히 GC에서 분명하게 나타났고 PC differentiation 중에 보존됨(예: Ms#7의 clone xvi 및 xvii는 모두 clone xvii가 우세함에도 불구하고 PC를 생성함)
clonal lineages 내에서 가장 낮게 검출된 GC antibody affinities와 일치하는 PC가 여전히 발견되었으며, maturation pathway의 여러 수준에서 differentiation이 다시 발생함. 일부 clones은 균일하게 더 적은 돌연변이(예: 비교적 낮은 친화도를 가진 Ms#7 clone xvii)를 가지고 있었음.
- 이는 기존 GC에 infiltrating한 세포가 성공적으로 PC를 만들기 시작했음을 시사.
[Figure 4E] 후기 단계의 GC는 더 적은 수의 PC를 생성했지만 유사한 selection rules을 가지고 있었음. 중요한 것은 상대적으로 antibody affinity이 약한 clones에서 생성된 PC도 UCA에 비해 훨씬 개선되었다는 점임.
- 이는 이러한 방식으로 PC를 선택하는 것이 진화적으로 유리하다는 것을 나타냄.
single GC에 대한 PC differentiation tracking.
– single GC에서 low- and high-affinity PC의 concurrent generation이 사실인지 조사
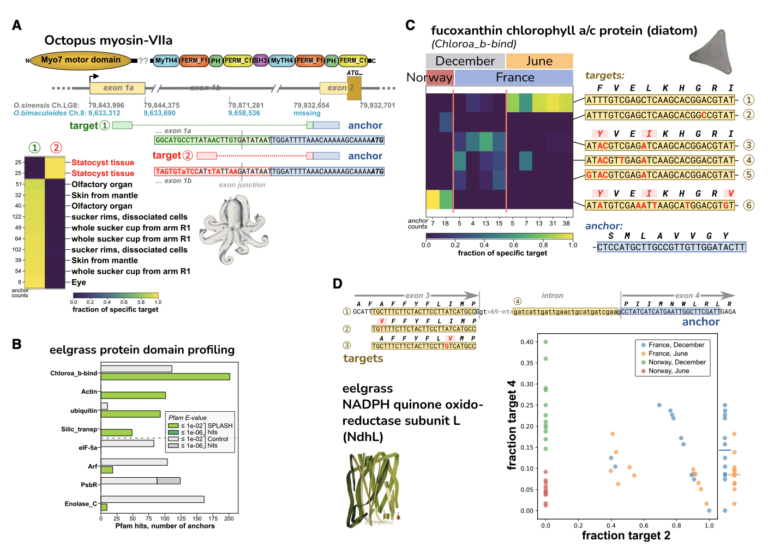
Figure 5. single GCs로 plasma cell differentiation tracking하기
(A) (B)-(F)의 실험 계획.
(B) single tdTom+ GC의 pre- and post-photoconversion의 Multi-photon microscopy image.
(C) HA+ tdTom+ PC와 HA+ PAGFP+ single GC B 세포를 분류하기 위한 Representative FACS gates.
(D) PC를 individual GC로 추적할 수 있는 두 마우스의 clonal lineages distribution을 보여주는 원형 차트.
(E) (D)에 표시된 clones에 대한 Phylogenetic maturation trees (GC).
(F) indicated clones (GC 및 PC)의 Fab의 HA에 대한 1가 Monovalent affinities (KD).
[Figure 5A-C] two-photon-microscopy-mediated photoactivatable GFP (PAGFP) approach를 채택하여 individual GC를 in situ에서 표시하고 FACS로 B 세포를 분류함.
[Figure 5D, E] PC는 2개의 GC로 성공적으로 추적되었으며, colored GC pie slices와 colored GC tree variants으로 differentiation이 발생한 위치를 표시함. PC는 dominant winner clones (예: Ms#10 clones i)과 GC에서 경쟁이 덜한 clones (예: Ms#10 clones ii 및 Ms#11 clones vi) 모두에서 차별화됨.
[Figure 5F] Ms#10 clones i-iii 및 Ms#11 clones iv-vi의 여러 GC B 세포와 PC에 대해 Fab을 발현하고 이들의 친화도를 비교함. Ms#10의 clones i과 ii는 affinities 중앙값이 ∼15배(116 nM vs 1.8 μM) 차이가 났지만 둘 다 PC를 생성함.
- 이 단일 GC에서 추적된 최고 및 최저 antibody affinity PC는 2.7 nM 및 13 μM로, 이는 동시에 ∼4,000배의 antibody affinity 차이를 가진 PC를 생성했음을 시사.
Immunization-induced GCs는 생산성은 떨어지지만 antibody affinities가 다른 PC의 development을 지원.
– subunit vaccination에 반응하여 형성된 GC에서도 유사한 PC selection rules이 적용되는지 조사
– GC B 세포와 tdTom+ PC의 HA-binding Fab의 affinities를 측정

Figure 6. GC는 immunization 후 더 적은 수의 plasma 세포를 생산하지만 여전히 서로 다른 antibody affinitie를 보임.
(A) (B)에 대한 실험 계획.
(B) infection or immunization 후 표시된 시점의 GC size-normalized PC output (1,000개의 tdTom+ GC B 세포당 tdTom+ PC의 수).
(C) (D) 및 (E)의 실험 계획.
(D) tdTom+ GC B 세포 중 Blimp1+ IRF4+ 세포의 비율을 보여주는 Representative FACS.
(E) (D)의 정량화.
(F) 3 days of tamoxifen treatment 후 day 14 post-HA/AddaS03 immunization에 tdTom+ GC B 세포 및 PC의 antibody gene을 시퀀싱했습니다.
(G) (F)에 표시된 clones에 대한 Phylogenetic maturation trees와 tdTom+ PC에 대한 관찰된 population sizes.
(H) 분석된 10개의 모든 LN에 걸쳐 GC에서 두개의 most immunodominant clones의 GC B 세포와 tdTom+ PC의 Ighv somatic mutation loads.
(I) 3 days-tamoxifen treatment 후 day 14 post-HA/AddaS03 immunization에 GC B 세포와 tdTom+ PC 중 HA-binding and non-binding cells의 비율을 보여주는 Representative FACS.
(J) (I)의 정량화.
(K) 다양한 clones을 반영하여 표시된 집단에서 Fab에 의한 HA binding에 대한 KD.
(L) (K)의 한 마우스에서 high and low affinity PC Fabs의 SPR single-cycle kinetic traces.
[Figure 6A] S1pr2tdTom Blimp1mVenus 마우스를 AS03-like adjuvant (AddaS03)에 recombinant HA protein을 subcutaneously (s.c.)로 immunized시킨 후, 새로 생성되는 PC의 특성을 다시 분석함. [Figure 6B] immunization 후 PC production efficiency는 현저하게 감소했으며, GC 크기에 따라 결과를 정상화했을 때에도 infection 시보다 훨씬 적은 수의 PC를 생성(14일째 17배 감소).
[Figure 6C] tamoxifen treatments가 어느 정도 differentiation을 허용하되 proliferation및/또는 apoptosis로 인한 secondary effects를 최소화해야 한다는 근거에 따라 tamoxifen treatments를 더 짧게(30시간) 한 추가 GC fate-mapping experiments을 수행함.
[Figure 6D, E] 유사한 tdTom-labeling kinetics에도 불구하고 infection 중에 IRF4high 및 Blimp1+인 tdTom+ GC-phenotype 세포의 비율이 훨씬 높았으며, 이는 해당 환경에서 differentiation이 더 자주 시작되는 것과 일치함
[Figure 6F] immunization-induced GC가 더 적은 수의 PC를 생성했음에도 불구하고, 우세하고 지배적인 GC clonal lineages가 다시 differentiation됨.
[Figure 6G, H] PC differentiation은 다시 overall clonal maturation process를 대략적으로 반영했는데, 이는 tdTom+ PC sequences가 phylogeny의 여러 수준에 매핑되고 Ighv somatic mutation levels이 GC에 비해 tdTom+ PC에서 비슷하거나 약간 낮았기 때문임.
[Figure 6I, J] HA/AddaS03 immunized 마우스의 GC에서 HA FACS probe negative (HA-) 세포가 분명하게 나타났으며, 이들은 또한 tdTom+ PC가 됨.
[Figure 6J] HA- 세포가 differentiation되는 상대적 효율은 마우스마다 달랐지만, 모든 마우스/실험을 합쳤을 때 HA+ GC 세포가 훨씬 더 많은 PC를 생성함 (GC에서 HA+ 중앙값 39%, tdTom+ PC에서 56%).
- damaging 또는 dead-end mutations을 지닌 세포가 GC 집단에 존재하면 이러한 효과에 기여할 수 있음. 그러나 이는 또한 HA multimers의 검출 가능한 결합을 부여하기 위해 최소 affinity thresholds에 도달하는 세포의 우선적(배타적이지는 않지만) differentiation을 반영할 수도 있음.
[Figure 6K, L] 여러 clonal lineages의 Fab을 분석했을 때, GC B 세포와 tdTom+ PC의 antibody affinities는 모두 1000배이상에 달함.
- 종합적으로, infection과 immunization에 대한 우리의 결과는 GC PC differentiation을 유도하는 선택적 기준이 affinity maturation을 유도하는 기준을 거의 따라간다는 것을 나타냄.
Immunization-induced GCs는 생산성은 떨어지지만 antibody affinities가 다른 PC의 development을 지원.
– subunit vaccination에 반응하여 형성된 GC에서도 유사한 PC selection rules이 적용되는지 조사
– GC B 세포와 tdTom+ PC의 HA-binding Fab의 affinities를 측정

Figure 7. GC는 immunization 후 더 적은 수의 plasma 세포를 생산하지만 여전히 서로 다른 antibody affinitie를 보임.
(A) (B)에 대한 실험 계획.
(B) infection or immunization 후 표시된 시점의 GC size-normalized PC output (1,000개의 tdTom+ GC B 세포당 tdTom+ PC의 수).
(C) (D) 및 (E)의 실험 계획.
(D) tdTom+ GC B 세포 중 Blimp1+ IRF4+ 세포의 비율을 보여주는 Representative FACS.
(E) (D)의 정량화.
(F) 3 days of tamoxifen treatment 후 day 14 post-HA/AddaS03 immunization에 tdTom+ GC B 세포 및 PC의 antibody gene을 시퀀싱했습니다.
(G) (F)에 표시된 clones에 대한 Phylogenetic maturation trees와 tdTom+ PC에 대한 관찰된 population sizes.
(H) 분석된 10개의 모든 LN에 걸쳐 GC에서 두개의 most immunodominant clones의 GC B 세포와 tdTom+ PC의 Ighv somatic mutation loads.
(I) 3 days-tamoxifen treatment 후 day 14 post-HA/AddaS03 immunization에 GC B 세포와 tdTom+ PC 중 HA-binding and non-binding cells의 비율을 보여주는 Representative FACS.
(J) (I)의 정량화.
(K) 다양한 clones을 반영하여 표시된 집단에서 Fab에 의한 HA binding에 대한 KD.
(L) (K)의 한 마우스에서 high and low affinity PC Fabs의 SPR single-cycle kinetic traces.
[Figure 6A] S1pr2tdTom Blimp1mVenus 마우스를 AS03-like adjuvant (AddaS03)에 recombinant HA protein을 subcutaneously (s.c.)로 immunized시킨 후, 새로 생성되는 PC의 특성을 다시 분석함. [Figure 6B] immunization 후 PC production efficiency는 현저하게 감소했으며, GC 크기에 따라 결과를 정상화했을 때에도 infection 시보다 훨씬 적은 수의 PC를 생성(14일째 17배 감소).
[Figure 6C] tamoxifen treatments가 어느 정도 differentiation을 허용하되 proliferation및/또는 apoptosis로 인한 secondary effects를 최소화해야 한다는 근거에 따라 tamoxifen treatments를 더 짧게(30시간) 한 추가 GC fate-mapping experiments을 수행함.
[Figure 6D, E] 유사한 tdTom-labeling kinetics에도 불구하고 infection 중에 IRF4high 및 Blimp1+인 tdTom+ GC-phenotype 세포의 비율이 훨씬 높았으며, 이는 해당 환경에서 differentiation이 더 자주 시작되는 것과 일치함
[Figure 6F] immunization-induced GC가 더 적은 수의 PC를 생성했음에도 불구하고, 우세하고 지배적인 GC clonal lineages가 다시 differentiation됨.
[Figure 6G, H] PC differentiation은 다시 overall clonal maturation process를 대략적으로 반영했는데, 이는 tdTom+ PC sequences가 phylogeny의 여러 수준에 매핑되고 Ighv somatic mutation levels이 GC에 비해 tdTom+ PC에서 비슷하거나 약간 낮았기 때문임.
[Figure 6I, J] HA/AddaS03 immunized 마우스의 GC에서 HA FACS probe negative (HA-) 세포가 분명하게 나타났으며, 이들은 또한 tdTom+ PC가 됨.
[Figure 6J] HA- 세포가 differentiation되는 상대적 효율은 마우스마다 달랐지만, 모든 마우스/실험을 합쳤을 때 HA+ GC 세포가 훨씬 더 많은 PC를 생성함 (GC에서 HA+ 중앙값 39%, tdTom+ PC에서 56%).
- damaging 또는 dead-end mutations을 지닌 세포가 GC 집단에 존재하면 이러한 효과에 기여할 수 있음. 그러나 이는 또한 HA multimers의 검출 가능한 결합을 부여하기 위해 최소 affinity thresholds에 도달하는 세포의 우선적(배타적이지는 않지만) differentiation을 반영할 수도 있음.
[Figure 6K, L] 여러 clonal lineages의 Fab을 분석했을 때, GC B 세포와 tdTom+ PC의 antibody affinities는 모두 1000배이상에 달함.
- 종합적으로, infection과 immunization에 대한 우리의 결과는 GC PC differentiation을 유도하는 선택적 기준이 affinity maturation을 유도하는 기준을 거의 따라간다는 것을 나타냄.
Disscussion
SPLASH는 참조 게놈에 의존하지 않고 원본 시퀀싱 데이터를 직접 분석하여 시퀀스 변이를 식별하는 새로운 통계적 프레임워크입니다. SARS-CoV-2의 복잡한 변이 감지 및 단일 세포 데이터에서의 유전자 차이 발현과 같은 다양한 유전체 분석의 하위 분야에서 그 효과를 입증했습니다. 다양한 생물체에 적용되어 유전체 감시와 생물학적 연구에 있어 잠재력을 보여주었습니다. SPLASH는 사용자 친화적이며, 깊이 있는 연구를 위한 다양한 지표를 제공하며, 원시 데이터만으로 광범위한 분석을 수행할 수 있습니다. 전통적인 참조 의존적 방법에서 벗어나 더 데이터 기반의 확장된 연구를 약속하는 유전체 분석의 독특한 접근법을 제공합니다. 이러한 전환은 유전적 다양성에 대한 더 깊은 이해를 가능하게 하며, 다양한 ‘오믹스’ 연구에 광범위한 함의를 가집니다. SPLASH는 확장되는 유전체 연구 분야에서 중요한 도구가 됩니다.
Limitation of the study
SPLASH는 현재 중요한 다양한 분야의 문제에 적용될 수 있으며, 이전에 논의된 문제들을 포함합니다(별첨 STAR Methods 참조). 당연히, 여기에서 제시된 SPLASH 분석에 직접적으로 적용하기 어려운 문제들도 있습니다. 가장 명확한 경우는 표본 특이적 RNA 또는 DNA의 양을 단독으로 정량화하고자 하는 경우입니다(예: 차등 유전자 발현 분석). 또한, SPLASH는 현재 탐지된 변이에 원인이 되는 생물학적 기작을 구별할 수 없으며, 이 문제를 해결하기 위한 작업이 진행 중입니다.