시험관 내 소형 대장 모델을 통해 시공간적으로 해석된 대장암 발생 과정 관찰
Abstract
3차원 오가노이드 배양 기술은 종양 및 미세환경 구조를 보다 현실적이고 확장 가능하게 재현할 수 있게 하여 암 연구에 혁신을 가져왔습니다. 이를 통해 비교적 짧은 기간 동안 발생하는 저복잡도의 암세포 행동을 더 잘 모델링할 수 있었습니다. 그러나 기존의 오가노이드 시스템은 조직 구조, 세포 다양성, 항상성 및 수명 측면에서 암 발달의 복잡한 진화 과정을 포착하지 못합니다. 그 결과, 종양 발생 및 형성 연구는 시험관 내에서는 불가능하며, 대신 세포 역학의 시공간적 해상도가 제한되고 상당한 자원과 동물의 생명을 필요로 하는 동물 모델을 광범위하게 사용해야 합니다. 저자는 미세가공, 광유전학 (optogenetics) 및 조직 공학 접근법을 통합하여 시험관 내에서 원하는 종양 발생 부위와 시간에 암 유전자를 활성화시켰습니다. 이렇게 만들어진 소형 대장 모델은 특정한 파장의 청색광에 노출되면 암 유전자가 활성화되어 정상 조직이 암으로 진행하게 됩니다. 또한, 유도된 소형 대장 종양을 몇 주 동안 단일 세포 해상도로 실시간 추적할 수 있습니다. 이러한 소형 대장 모델은 풍부한 종양 내 및 종양 간 다양성을 나타내며, 생체 내 대장 종양이 나타내는 주요 병리생리학적 특징을 재현합니다. 세포 내적 및 세포 외적 매개변수를 미세 조정하여 소형 대장은 종양 유발 결정 요인 및 약리학적 연구에 사용할 수 있습니다. 결론적으로, 해당 연구는 생체 외부에서 암 발생 연구의 길을 열어줍니다.
Figures
Divergent epigenome remodelling in hiPS cells
– naive and primed reprogramming 전반에 걸친 epigenome remodelling을 조사.

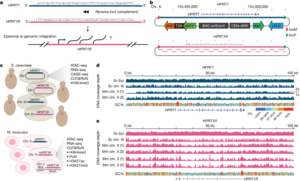
Fig. 1. 소형 대장 모델을 통해 de novo 종양을 유도할 수 있습니다.
(A) 소형 결장에서 종양 형성을 유도하기 위한 실험 작업 흐름의 개요도. CC(colonocyte)는 대장 세포, ISC (intestinal stem cell)는 장 줄기 세포, TA (transit-amplifying cell)는 전이 증폭 세포를 나타냄.
(B) 일반 오가노이드와 소형 대장 모델에서 시간 경과에 따른 종양 형성 실험의 밝은 필드 및 형광 이미지. 형광 신호는 발암 유전자의 재조합을 나타냄. 눈금 막대는 각각 200μm(왼쪽) 및 75μm(오른쪽)임.
(C) 소형 대장 모델의 밝은 필드 및 형광 근접 이미지. 빨간색 신호는 건강한 세포를, 녹색 신호는 변이된 세포를 나타냄. 눈금 막대는 25μm임.
(D) 소형 대장 모델의 면역 형광 이미지로 CD44(위, 녹색), FABP1(위, 자주색) 및 핵(아래)을 보여줌. 눈금 막대는 35μm임.
(E) 빛을 이용한 발암 유도 후 나타난 다양한 유전자형을 가진 소형 대장암 모델에서의 종양 수.
[Fig 1E] 통계 분석은 Sidak의 다중 비교 검정을 사용한 이원 분산 분석(ANOVA)로 수행됨. **P = 0.024 (6일, AKP*), **P = 0.0021 (24일, A), ***P < 0.0001 (다른 모든 조건). n = 5, 4, 3 및 10 소형 대장 모델은 각각 대조군, 빛에 의해 유도된 A, 빛에 의해 유도된 AK 및 빛에 의해 유도된 AKP 조건을 나타냄. 데이터는 평균 ± 표준 오차를 나타냄.
*AKP = Apcfl/flKrasLSL-G12D/+Trp53fl/fl
Apcfl/fl: 대장암에서 자주 돌연변이가 발생하는 Apc 유전자의 조건부 knock-out.
KrasLSL-G12D/+: 일반적으로 암에서 발견되는 G12D 돌연변이를 가진 Kras 유전자의 조건부 대립유전자.
Trp53fl/fl: 잘 알려진 종양 억제 유전자인 Trp53 유전자의 조건부 knock-out.
AKP 모델은 이러한 돌연변이를 Cre-lox 기술을 사용하여 특정 시기와 위치에서 유전자 재조합을 활성화하여 대장암을 유도하는 데 사용됨.
Emergence of aberrant DNA methylation
– hiPS 세포는 hES 세포와 비교하여 somatic cell epigenetic memory 또는 후천적 비정상 methylation states로 분류될 수 있는 differentially methylated regions (DMR)을 특징으로 하며, 이는 origin 또는 hES 세포에는 존재하지 않는 hiPS 세포 고유의 methylation 상태.
– hiPS 세포와 hES 세포 간의 DNA methylation 차이에 대한 보고에도 불구하고, reprogramming 동안의 temporal dynamics는 잘 알려져 있지 않음.
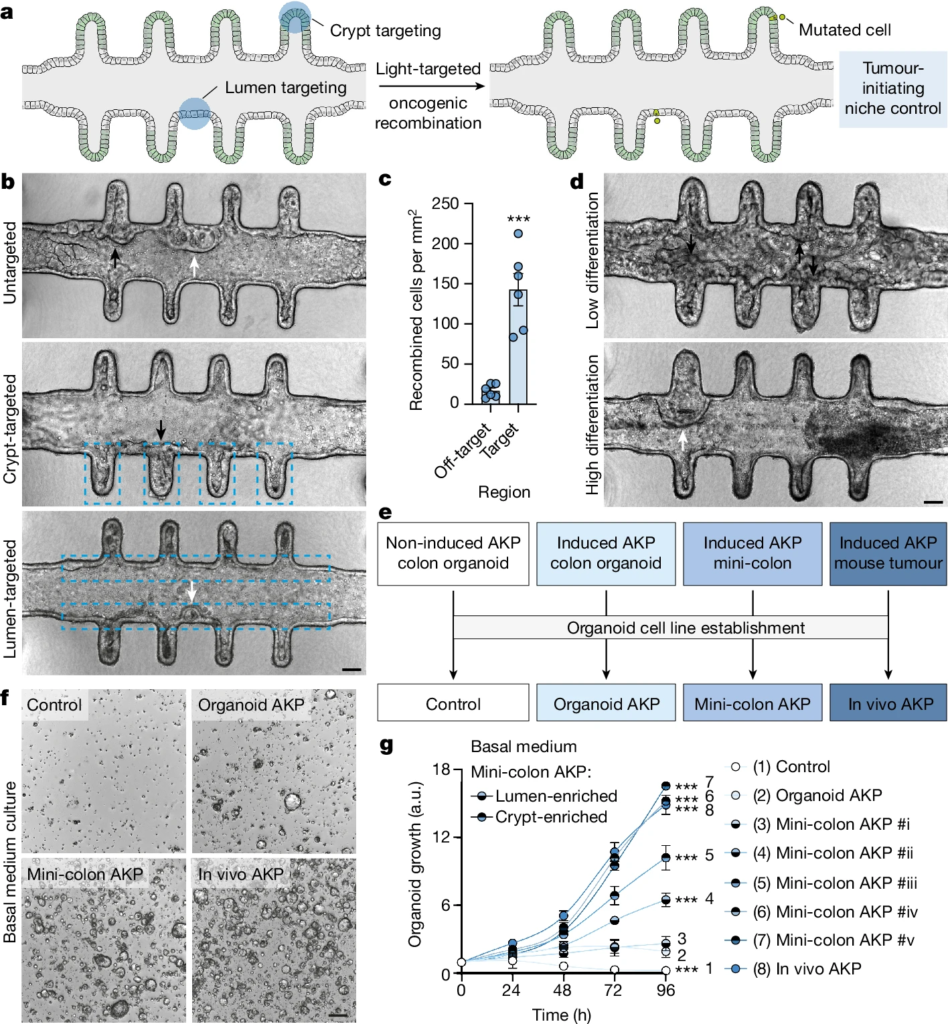
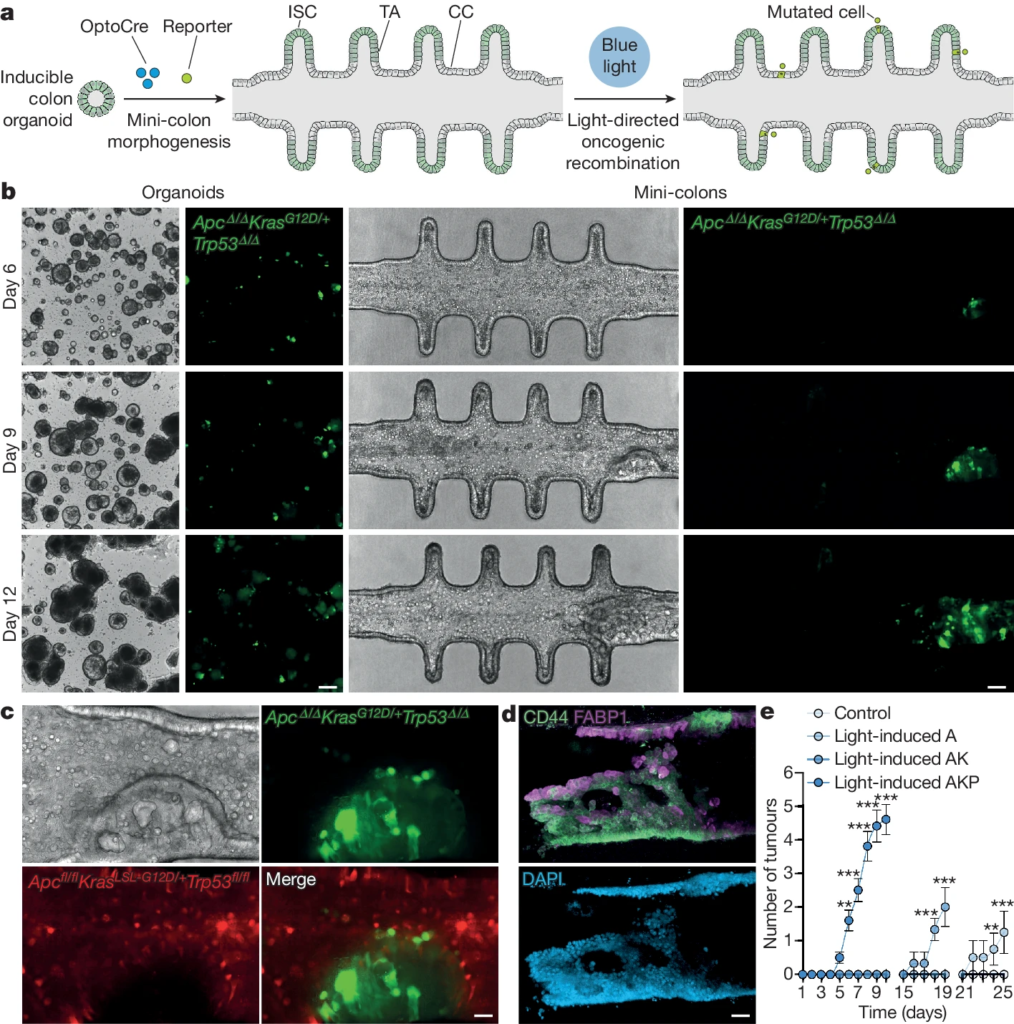
Fig. 2. 소형 대장 모델은 상황 의존적인 종양 발생 가소성을 나타냅니다.
(A) 소형 대장 모델에서 시공간적으로 종양 형성을 표적화하기 위한 실험 작업 흐름의 개요도.
(B)비표적화(상단), 크립트 표적화(중간) 및 루멘 표적화(하단) 종양 형성을 거친 소형 결장의 밝은 필드 이미지. 표적화된 영역은 파란색 점선으로 표시됨. 검은색 화살표는 밀집형 종양을, 흰색 화살표는 낭성 형태의 종양을 나타냄. 눈금 막대는 75μm임.
(C) 표적화된 영역과 비표적화된 영역에서의 소형 대장의 암 유전자 재조합 효율.
(D) 저분화(상단, WENRNi) 및 고분화(하단, ENR) 조건에서 배양된 유도 소형 대장의 밝은 필드 이미지. 검은색 화살표는 밀집형 종양을, 흰색 화살표는 낭성 형태의 종양을 나타냄. 눈금 막대는 75μm임.
(E) 본 연구에서 생성된 다양한 대장 오가노이드 라인의 개요도.
(F) 기본 배지에서 2일 동안 배양된 해당 대장 오가노이드 라인의 밝은 필드 이미지. 눈금 막대는 200μm임.
(G) 지정된 시간 동안 기본 배지에서 배양된 해당 대장 오가노이드 라인의 대사 활동(레사주린을 사용하여 측정). 세포주 식별을 용이하게 하기 위해 숫자 표기(1-8)를 사용함.
*AKP = Apcfl/flKrasLSL-G12D/+Trp53fl/fl
Apcfl/fl: 대장암에서 자주 돌연변이가 발생하는 Apc 유전자의 조건부 knock-out.
KrasLSL-G12D/+: 일반적으로 암에서 발견되는 G12D 돌연변이를 가진 Kras 유전자의 조건부 대립유전자.
Trp53fl/fl: 잘 알려진 종양 억제 유전자인 Trp53 유전자의 조건부 knock-out.
AKP 모델은 이러한 돌연변이를 Cre-lox 기술을 사용하여 특정 시기와 위치에서 유전자 재조합을 활성화하여 대장암을 유도하는 데 사용됨.
[Fig 2A] 통계 분석은 양측 t-검정을 사용함; ***P < 0.0001. 조건당 소형 결장 n = 6개. 각 점은 하나의 소형 결장을 나타냄.
[Fig 2G] 통계 분석은 Sidak의 다중 비교 검정을 사용한 이원 분산 분석(ANOVA)으로 수행됨; ***P = 0.0004 (대조군), ***P < 0.0001 (다른 모든 조건). 각 라인의 배양 n = 3개. c와 g의 데이터는 평균 ± 표준 오차를 나타냄.*
TNT reprogramming resets the epigenome
– early development 과정에서 pre-implantation embryo는 global demethylation wave와 관련된 epigenetic reset을 겪으며, 이 과정에서 genomic imprints가 demethylation으로부터 보호됨.
– embryonic development 중에 관찰되는 demethylation과 유사한 transient naive-like state를 통해 reprogramming함으로써 somatic cell epigenetic memory와 비정상적인 DNA methylation을 피할 수 있다는 가설 세움. 따라서 두 가지 실험 시스템 계획.
- fibroblasts medium에서 초기 7일간 배양한 후 5일간 transient naive culture treatment로 fibroblasts를 reprogramming한 다음 나머지 reprogramming을 위해 primed medium에서 배양하여 transient-naive-treatment hiPS 세포 (TNT-hiPS 세포)를 생성 (Fig.3a).
- 먼저 naive culturing을 연장하여 naive-hiPS cell colonies를 확립한 다음, 세포를 primed pluripotent state로 전환하여 naive-to-primed hiPS 세포 (NTP-hiPS 세포)를 생성 (Fig.3a).
Fig. 3. 소형 대장 모델은 종양 내 및 종양 간 다양성을 보여줍니다.
(A) 소형 대장 모델의 단일 세포 및 계통 추적 분석을 위한 실험 작업 흐름의 개요도.
(B) 종양 유도 후 7일째 소형 결장의 주요 세포 유형에 대한 비지도 UMAP 클러스터링.
(C) 소형 대장 모델을 구성하는 다양한 세포 집단에서 대표적인 세포 유형 특이적 마커의 발현(Exp.)을 나타냄.
(D) 소형 대장 모델에서 건강한(상단) 및 종양(하단) 클론 집단의 비지도(Unsupervised) 클러스터링(UMAP). 세포 유형(왼쪽; b와 같은 색상 코드)과 클론 정체성(오른쪽)이 표시됨.
(E) 건강한 소형 대장 클론 집단과 종양 소형 결장 클론 집단의 상대적 세포 유형 풍부도. 데이터는 평균 ± 표준 오차를 나타냄. 각각 n = 16 및 18개의 건강한 클론과 종양 클론임.
(F) 건강한 소형 대장 클론 집단과 종양 소형 결장 클론 집단의 크기.
(G) Gpx2 발현과 암 줄기 세포 전사 서명 강화(Cd44, Lgr5, Sox9) 사이의 상관관계.
(H) 건강한(오른쪽) 및 종양(왼쪽, 화살표로 표시된) 소형 대장 모델의 크립트에서 GPX2(자주색)와 핵(청록색)의 풍부함을 보여주는 밝은 필드 및 면역 형광 이미지. 눈금 막대는 35μm임.
(I) 지정된 종양 클론에서 지정된 유전자들의 발현.
[Fig 3F] 통계 분석은 양측 Mann–Whitney U-검정을 사용함; **P = 0.0011. 각각 n = 16 및 18개의 건강한 클론과 종양 클론임. 상자 그림은 중앙값(중앙 선), 제1 및 제3 사분위수(상자 경계)와 최소 및 최대 값(수염)을 나타냄. 각 점은 하나의 클론 집단을 나타냄.
[Fig 3G] 통계 분석은 양측 Pearson 상관관계 검정을 사용함; P < 0.0001. n = 540개 세포. 각 점은 하나의 세포를 나타냄. CSC는 암 줄기 세포, ES는 강화 점수를 나타냄.
[Fig 3I] 통계 분석은 양측 Wilcoxon 순위 합 검정을 사용함; ***P = 1.77 × 10^-17 (Il1a, 클론 1), 1.00 × 10^-78 (Cdkn2a, 클론 14), 3.67 × 10^-22 (Cdkn2a, 클론 48). n = 540개 세포. 각 점은 하나의 세포를 나타냄.
Correction persists through differentiation
이전 연구에 따르면 primed-hiPS 세포의 epigenetic memory와 aberrations은 differentiation 후에도 지속될 수 있으며, 이는 resulting cells에 기능적으로 영향을 미칠 수 있음.
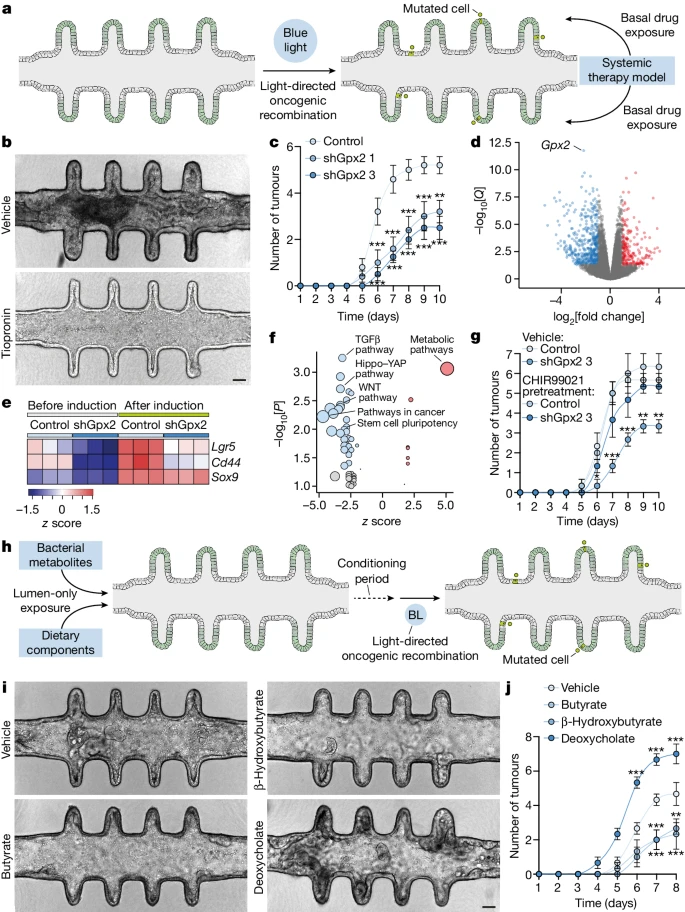
Fig. 4. 소형 대장 모델은 생리학적으로 암 유발 요인의 분석을 가능하게 합니다.
(A) 체계적 치료 모델링을 위한 실험 작업 흐름.
(B) 발암 재조합 후 차량 또는 티오프로닌으로 처리된 소형 대장 모델의 밝은 필드 이미지. 이미지는 유도 후 6일에 해당함. 눈금 막대는 75μm임.
(C) 발암 유도 후 지정된 유전자형의 소형 대장 모델에서 나타난 종양의 다발성.
(D) 빛에 의해 유도된 AKP 종양 세포에서 Gpx2 억제 후 차별적으로 발현된 유전자들.
(E) 발암 재조합 전후의 지정된 유전자형의 대장세포에서 지정된 유전자들의 발현. 색상 척도는 z 점수를 나타냄.
(F) 빛에 의해 유도된 AKP 종양 세포에서 Gpx2 억제 후 주요 강화 기능적 용어. 유전자 발현 조정 P-값(Benjamini-Hochberg 보정)을 사용한 단측 Fisher의 정확한 검정으로 결정된 유의미한 용어는 파란색 또는 빨간색으로 강조됨.
(G) 발암 유도 전(2일 전) 특정 전처리 하에서 지정된 유전자형의 소형 대장 모델에서 나타난 종양의 다발성.
(H) 미생물군 및 식이 패턴 모델링을 위한 실험 작업 흐름. BL, 청색광.
(I) 지정된 대사 산물로 처리된 소형 대장 모델의 밝은 필드 이미지. 이미지는 발암 유도 후 7일에 해당함. 눈금 막대는 75μm임.
(J) 지정된 대사 산물로 처리된 소형 대장 모델에서 나타난 종양의 다발성.
c, g 및 j의 데이터는 평균 ± 표준 오차를 나타냄.
[Fig 4C] 통계 분석은 Sidak의 다중 비교 검정을 사용한 이원 분산 분석(ANOVA)으로 수행됨; **P = 0.0034, ***P = 0.0007 (6일 및 9일, shGpx2 1), ***P < 0.0001 (다른 모든 조건). 대조군, shGpx2 1 및 shGpx2 3에 대해 각각 소형 결장 n = 5, 5 및 4개임.
[Fig 4G] 통계 분석은 Sidak의 다중 비교 검정을 사용한 이원 분산 분석(ANOVA)으로 수행됨; *P = 0.0274, **P = 0.0033 (9일 및 10일), ***P < 0.0001 (7일 및 8일). 각 조건에 대해 소형 결장 n = 3개임.
[Fig 4J] 통계 분석은 Sidak의 다중 비교 검정을 사용한 이원 분산 분석(ANOVA)으로 수행됨; **P = 0.0080, ***P = 0.0008 (7일 및 8일), ***P < 0.0001 (6일). 각 조건에 대해 소형 결장 n = 3개임.
Disscussion
본 연구에서는 소형 대장 모델이 암 발생 연구의 패러다임을 바꾸어, 시공간적 종양 형성, 실시간 단일 세포 해상도 및 폭넓은 실험적 유연성을 통해 시험관 내에서 종양 형성을 가능하게 함을 보여주었습니다. 이 시스템은 암 발생의 세포 및 분자적 결정 요인을 시험관 내에서 스크리닝할 수 있는 새로운 전망을 열어줍니다. 소형 대장 모델은 생체 내 미생물 대사 산물 및 식이 패턴에 대한 반응을 반영하고 있습니다. 또한, 글루타티온 퍼옥시다제 억제가 대장암 종양 발생을 차단한다는 발견은 유전자 표적 및 종양 억제 약물의 발견과 검증에 도움을 줍니다.
대부분의 대장암 유전 모델과 마찬가지로, 본 연구는 여러 돌연변이의 동시 획득을 기반으로 하므로, 생체 내에서 발생하는 종양 발생 과정을 완전히 재현하지는 못하나, 단계적 돌연변이 시스템을 활용하면 소형 대장의 암 발생 모델로서의 관련성이 개선될 것이라고 보여집니다. 또한, 공간 전사체학 접근법을 통해 소형 결장에서의 종양 다양성의 이해를 넓혀줄 것입니다. OptoCre 시스템에 추가적인 조절 기능을 더하여 더 정밀한 시공간적 재조합 조절을 달성하는 방법도 유망하다고 봅니다.
소형 대장 모델이 모든 암 연구 맥락에서 동물 실험을 대체할 수는 없지만, 다양한 실험 응용에서 동물 사용을 줄일 수 있는 가능성을 보여줍니다. 중요한 것은, 주변 생체 모방 세포외 기질에 기질 세포를 포함시켜 소형 대장 모델의 병리 생리학적 관련성을 쉽게 향상시킬 수 있다는 점입니다. 현재 진행 중인 연구는 이 모델이 환자 유래 대장암 표본에도 적용될 수 있음을 증명합니다. 마지막으로, 소형 대장 모델의 생체 역학적 특성, 지형 및 배양 조건을 조정하여 폐, 유방, 전립선 등 다른 주요 상피암 유형으로 확장할 수 있을 것으로 기대합니다.