complex tissue에서 단일 세포의 공간적으로 해결된 epigenomic 프로파일링
Abstract
최근 spatial omics 방법의 개발로 transcriptome 및 공간 해상도가 높은 3D genome organization의 단세포 프로파일링이 가능해졌다. spatial omics 도구의 레퍼토리를 확장하면, 공간적으로 해결된 single-cell epigenomics 방법은 세포와 tissue 기능의 공간 조절에 대한 이해를 가속화할 것이다. 여기서, 우리는 in situ tagmentation and transcription, 그리고 다중화된 이미징을 사용하여 단일 세포의 공간적으로 해결된 epigenomic profiling을 위한 방법을 보고한다. 우리는 개별 세포에서 active promoters, putative enhancers, 그리고 silent promoter를 표시하는 histone modification을 프로파일링할 수 있는 능력을 입증했고, embryonic and adult mouse brain에서 수백 개의 active promoter와 putative enhancer의 고해상도 공간 아틀라스를 생성했다. 우리의 결과는 발전적으로 중요한 유전자를 조절하는 putative promoter-enhancer pair와 enhancer hub를 제안했다. 우리는 이 접근 방식이 일반적으로 epigenetic modification과 DNA-binding protein의 spatial profiling에 적용되어 유전자 발현이 epigenome에 의해 시공간적으로 조절되는 방법에 대한 이해를 증진시킬 것으로 예상한다.
Figure
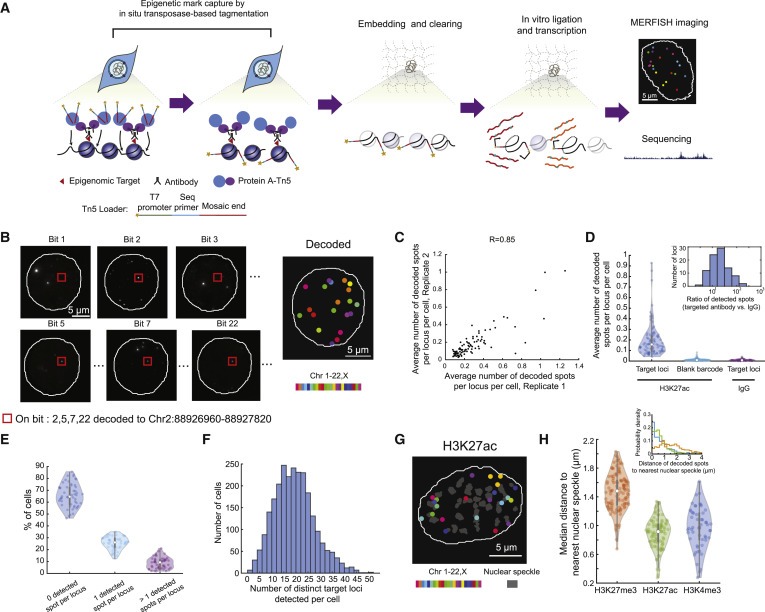
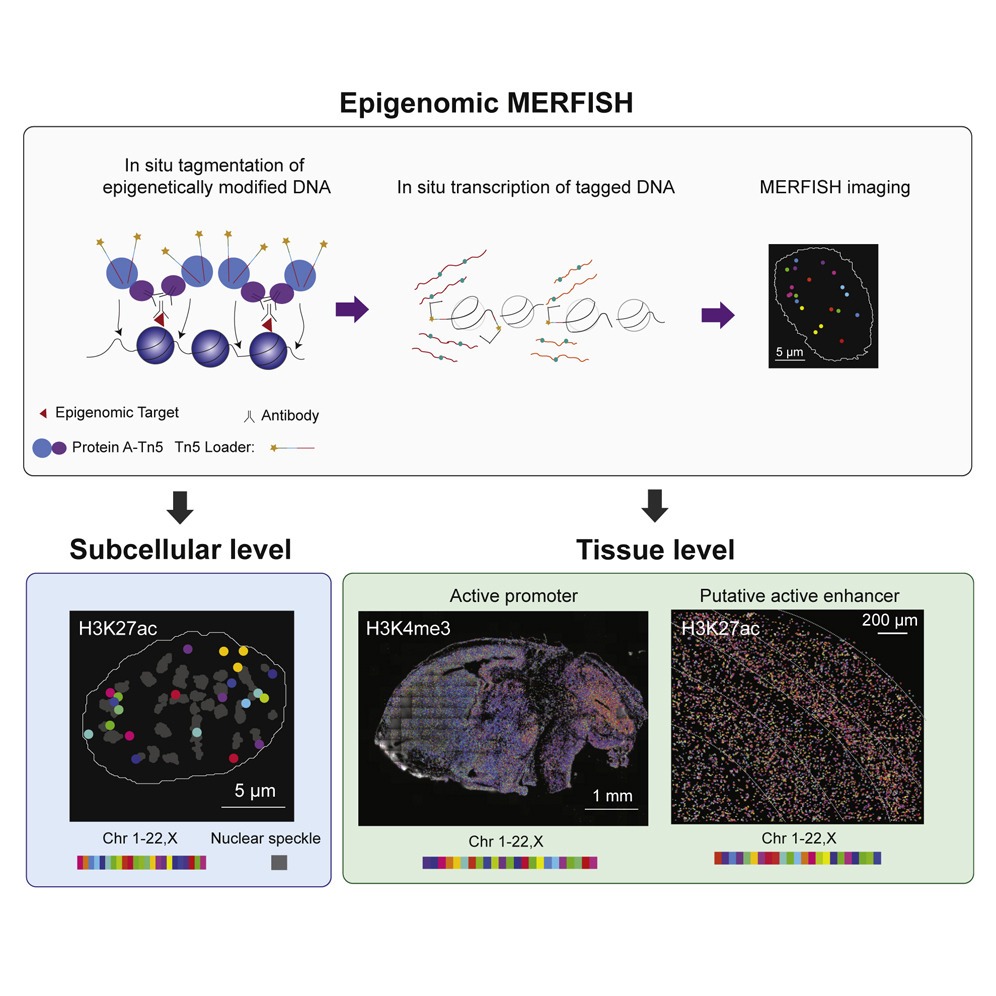
Figure 1. epigenomic MERFISH에 의한 epigenetic modification에 대한 공간적으로 해결된 single-cell profiling.
(A) 후생유전자 MERFISH의 개략도.
(B) 단일 세포에서 90개의 target H3K27ac loci의 epigenomic MERFISH image.
(C) H3K27ac imaging의 두 생물학적 복제 사이의 상관 관계.
(D) 각 target H3K27ac locus를 위한 decoded spots per cell의 평균 수를 보여주는 Violin plot (왼쪽), H3K27ac antibody가 epigenetic mark를 캡처하기 위해 사용될 때, 각 blank barcode (중간), control IgG가 대신 사용될 때, 각 target H3K27ac locus에 대한 decoded spots per cell의 평균 수 (오른쪽).
(E) 52 essential gene의 promoter들에 해당하는 개별 target H3K4me3 loci에 대해 0, 1, or >1 detected spot이 있는 cell의 비율을 보여주는 Violin plot.
(F) 셀당 뚜렷한 H3K4me3 loci detected의 개수에 대한 히스토그램.
(G) 단일 세포에서 90개의 target H3K27ac loci와 nuclear speckle의 decoded epigenomic MERFISH image.
(H) 검출된 H3K4me3, H3K27ac 및 H3K27me3 loci 와 the nearest nuclear speckle 사이의 중앙 거리를 보여주는 Violin plot.
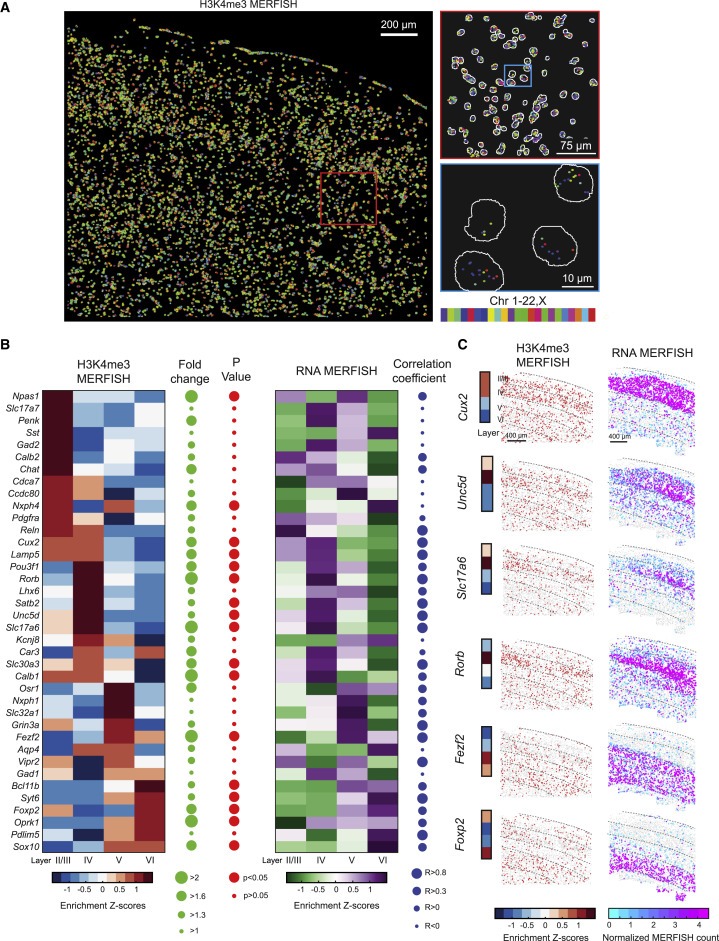
Figure 2. adult mouse cortex의 layer-specific active promoter의 공간적으로 해결된 단일 세포 프로파일링.
(A) adult mouse brain의 coronal slice의 somatosensory cortex region안에 있는 127개의 target H3K4me3 loci의 epigenomic MERFISH image (왼쪽). 왼쪽 패널에서 빨간색 상자 영역을 확대하여 표시 (오른쪽 상단). 오른쪽 상단 패널에서 파란색 상자 영역을 확대하여 표시 (오른쪽 아래).
(B) 표시된 유전자에 대해 epigenomic MERFISH에 의해 측정된 promoter H3K4me3 signal에 대한 layer enrichment Z score (왼쪽). 표시된 유전자에 대해 RNA MERFISH로 측정한 RNA 발현 수준에 대한 layer enrichment Z score (Zhang et al., 2021)(오른쪽).
(C) 6개의 표시된 유전자의 promoter에 대한 H3K4me3 신호의 layer enrichment를 보여주는 epigenomic MERFISH (왼쪽). dot으로 표시된 개별 세포에서 6개의 표시된 유전자에 대해 RNA 발현 수준을 보여주는 RNA MERFISH image (오른쪽).
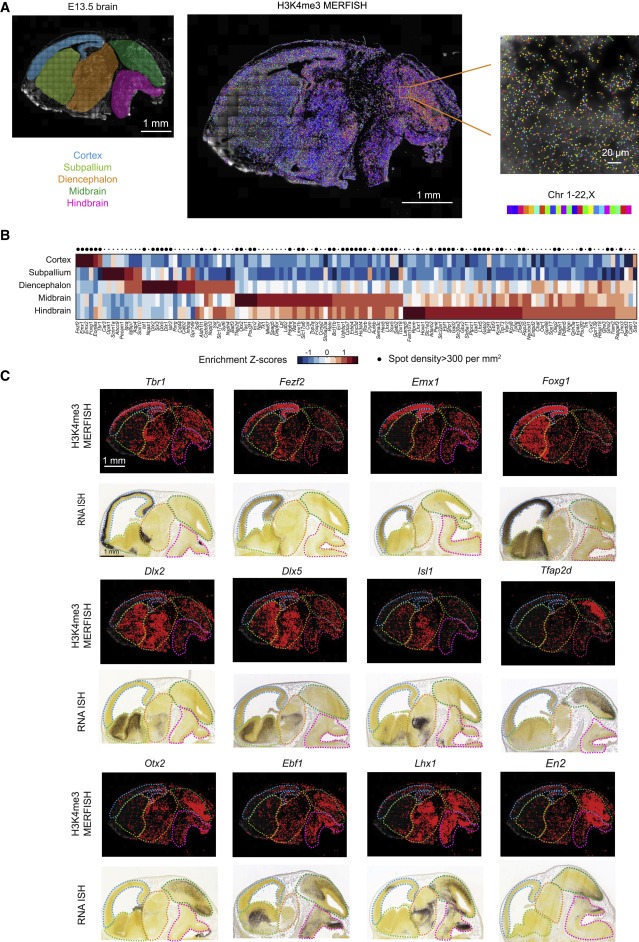
Figure 3. mouse embryonic brain의 active promoter에 대한 공간적으로 해결된 프로파일링.
(A) E13.5 마우스 뇌의 cortex, subpallium, diencephalon, midbrain, hindbrain을 도식적으로 강조 (왼쪽). 슬라이스에서 127개의 target H3K4me3 loci의 epigenomic MERFISH image (중간). 축척 막대: 1 mm.중뇌의 주황색 상자를 확대된 영역 (오른쪽).
(B) 다른 brain region안에 각 127 target H3K4me3 loci에 대한 brain region enrichment Z score.
(C) 해당 유전자의 Allen Brain RNA ISH 이미지와 비교하여 보여지는 12개의 transcription factor의 promoter의 H3K4me3 신호의 epigenomic MERFISH image.
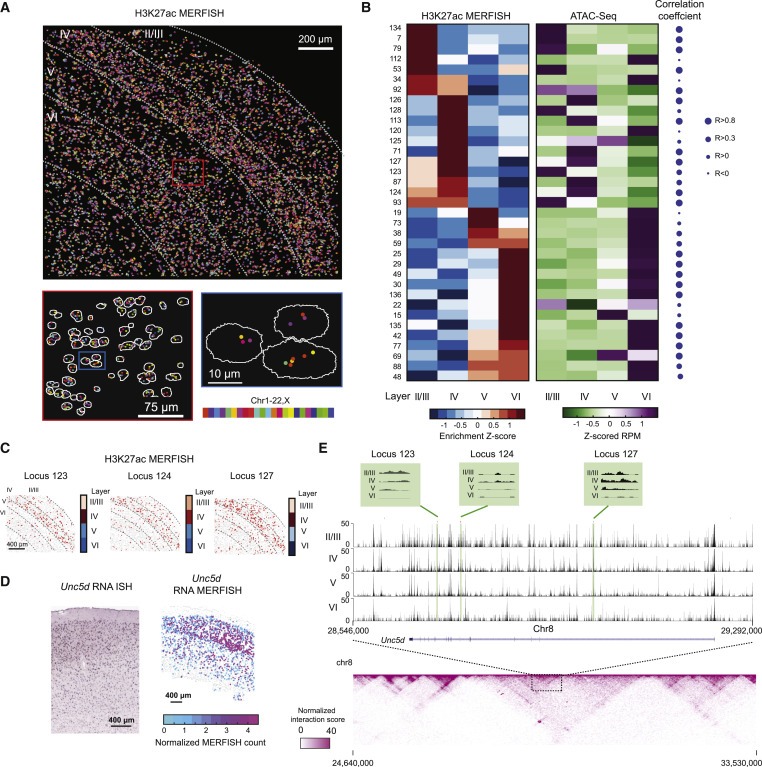
Figure 4. adult mouse cortex의 layer-specific putative active enhancer의 공간적으로 해결된 단일 세포 프로파일링.
(A) adult mouse brain의 coronal slice somatosensory cortex region 안에 있는 139개의 target H3K27ac loci의 epigenomic MERFISH image (상단). 상단 패널에서 빨간색 상자 영역을 확대하여 표시 (왼쪽 아래). 왼쪽 아래 패널에서 파란색 상자 영역을 확대하여 표시 (오른쪽 아래).
(B) 표시된 게놈 위치에 대해 epigenomic MERFISH에 의해 측정된 H3K27ac 신호에 대한 layer enrichment Z score (왼쪽). 공개된 ATAC-seq 데이터에서 각 target loci에 해당하는 Z-scored reads per million (RPM) (Gray et al., 2017) (오른쪽).
(C) layer II/III 및 IV의 enrichment을 보여주는 세 가지 예시적인 target loci에 대한 H3K27ac signal의 epigenomic MERFISH image.
(D) Unc5d 유전자의 RNA 발현 수준의 Allen RNA ISH 영상(왼쪽)과 RNA MERFISH 영상(오른쪽).
(E) Unc5d의 intronic region에서 세 개의 target loci (loci 123, 124, 127)의 위치를 보여주는 ATAC-seq 데이터 (Gray et al., 2017)를 추적하는 UCSC 브라우저 (상단). 이러한 위치를 포함하는 게놈 영역의 Hi-C 지도(Deng et al., 2015) (하단).
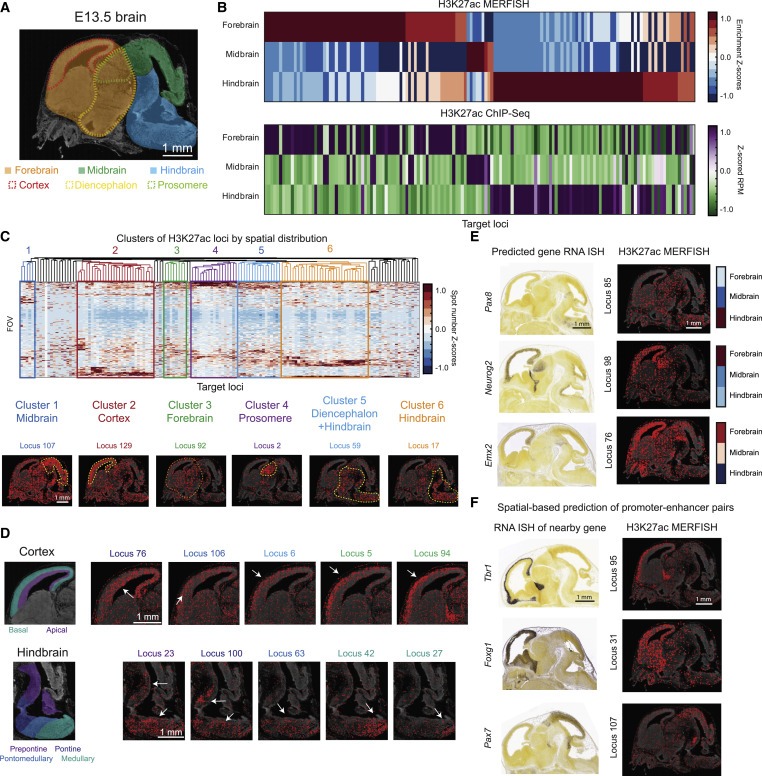
Figure 5. mouse embryonic brain에서 putative active enhancer의 공간적으로 해결된 프로파일링
(A) E13.5 마우스 뇌의 imaged sagittal slice의 다양한 뇌 영역을 도식적으로 강조.
(B) 142개의 target genomic loci에 대해 epigenomic MERFISH에 의해 측정된 H3K27ac 신호에 대한 brain region enrichment Z score (상단). E13.5 brain의 공개된 H3K27ac ChIP-seq 데이터에서 target loci에 해당하는 Z-scored reads per million (RPM) (하단).
(C) 개별 loci의 H3K27ac 신호의 측정된 공간 분포를 기반으로 142개의 target genomic loci의 계층적 cluster링 (상단). 하단: H3K27ac 신호의 epigenomic MERFISH image 각 cluster마다 하나씩 6개의 대표적인 loci.
(D) cortex와 hindbrain 내에서 미세한 공간 분포 변화를 보여주는 두 개의 위치 cluster의 loci에 대한 epigenomic MERFISH.
(E) epigenomic MERFISH에 의해 측정된 세 개의 putative enhancer loci의 H3K27ac 신호의 공간 분포 (오른쪽)와 Allen RNA ISH atlas의 해당 예측 유전자의 발현 패턴(왼쪽)의 비교.
(F) H3K27ac epigenomic MERFISH 데이터와 인근 유전자의 RNA 발현 패턴을 이용한 putative promoter-enhancer pair 예측.
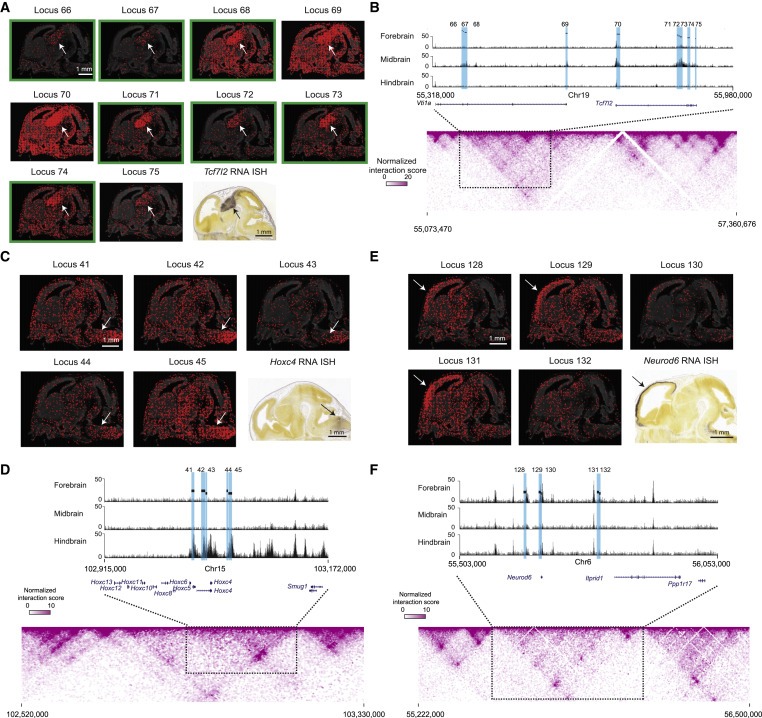
Figure 6. mouse embryonic brain에서 발달적으로 중요한 유전자를 위한 putative active enhancer hub.
(A) 근방 유전자 Tcf7l2의 Allen RNA ISH 이미지와 함께 표시된 prosomere cluster의 10개 target loci에 대한 H3K27ac 신호의 Epigenomic MERFISH 이미지.
(B) prosomere와 Tcf7l2 유전자가 풍부한 10개의 target H3K27ac를 보유하는 sub-TAD에 해당하는 영역의 H3K27ac ChIP-seq 트랙(Gorkin et al., 2020) (상단). sub-TAD와 측면 영역을 포함하는 게놈 영역의 Hi-C contact map (Deng et al., 2015) (하단).
(C) 근방 유전자 Hoxc4의 Allen RNA ISH 이미지와 함께 표시된 hindbrain cluster에 있는 5개의 target loci의 H3K27ac 신호의 후생학적 MERFISH 이미지.
(D) (B)에서와 같지만 hindbrain cluster의 5개 target loci.
(E) 근방 유전자 Neurod6의 Allen RNA ISH 이미지와 함께 표시된 cortex cluster에 있는 5개의 target loci의 H3K27ac epigenomic MERFISH 이미지.
(F) (B)에서와 같지만 cortex cluster의 5개 target loci.
Discussion
이 연구에서, 우리는 공간적으로 해결된 single-cell epigenomic 프로파일링을 위한 방법을 개발했다. 이 방법에서는 T7 프로모터를 사용하여 Tn5 기반 DNA 태깅을 사용하여 위치에서 epigenetic mark of interest in situ, in situ transcription을 사용하여 태그가 지정된 DNA 조각을 증폭한 다음, MERFISH 이미징을 사용하여 결과 RNA를 검출했다. 이 epigenetic MERFISH 방법을 사용하여 높은 공간 및 게놈 분해능과 높은 게놈 처리량을 가진 개별 세포에서 active promoters, putative enhancer 및 silent chromatin을 포함하여 크로마틴에 대한 epigenetic modification을 프로파일링할 수 있는 능력을 입증했다. 수백 개의 염기만큼 짧은 게놈 위치에 대한 Histone modification은 <1kb의 게놈 해상도를 제공하면서 이미지화할 수 있다. 이미징에 의해 제공되는 높은 공간 해상도는 이러한 게놈 위치의 하위 핵 위치 및 핵 구조와의 공간 관계를 결정할 수 있게 해주었다. 여기서의 proof-of-principle 시연에서, 우리는 수백 개의 게놈 위치의 histone modification을 이미지화했다. MERFISH는 개별 세포에서 10,000개 이상의 별개의 RNA를 이미지화하고 식별할 수 있기 때문에(Xia et al., 2019), epigenetic MERFISH의 게놈 처리량을 더욱 증가시켜 특정 epigenetic modification으로 수천 개의 게놈 위치를 동시에 프로파일링할 수 있을 것으로 기대한다.
epigenetic MERFISH가 조직 샘플에 적용될 수 있음을 입증했다. active promoters 및 putative enhancer를 표시하는 두 개의 뚜렷한 histone modification을 공간적으로 프로파일링하기 위해 이 접근 방식을 사용하여 성인 및 발달 중인 마우스 뇌 모두에서 active promoters 및 putative enhancer의 지역별 분포를 관찰했다. 이러한 측정은 기존 지식과 일치하는 active promoters 및 putative enhancer의 공간 패턴을 보여줬을 뿐만 아니라 이전에 알려지지 않은 putative enhancers의 미세한 공간 분포를 보여주었다. 우리의 데이터는 또한 뇌 발달에 관련된 유전자에 대한 putative enhancer-promoter pair과 enhancer hub를 제안했다.
세포 해리가 필요한 equencing-based single-cell epigenomic profiling 방법과 비교하여 epigenomic MERFISH는 세포의 공간적 맥락을 유지하므로 조직에서 epigenomic activity의 공간적으로 해결된 단일 세포 프로파일링을 가능하게 한다. 우리의 작업과 병행하여, 현장에서 CUT&Tag를 수행하여 sequencing-based spatial epigenomic profiling 방법(spatial CUT&Tag)이 개발되었으며, 이어서 microfluidics-assisted 공간 바코딩 및 시퀀싱이 수행되었다(Deng et al., 2022). 마이크로 유체 채널 너비에 의해 수십 미크론으로 제한된 공간 CUT&Tag의 해상도(픽셀 크기)는 단일 셀 분석을 어렵게 만든다. spatial CUT&Tag 측정의 일부 픽셀만이 단일 셀을 포함하는 반면, 대부분의 픽셀은 둘 이상의 셀로부터 기여한다(Deng et al., 2022). 이에 비해 imaging-based epigenomic MERFISH는 공간분해능이 훨씬 높아 단세포 분석이 가능할 뿐만 아니라 개별 세포 내에서 핵하위 조직을 조사할 수 있다. epigenomic MERFISH도 한계가 있다. epigenomic 부위의 표적화되지 않은 게놈 전반의 탐지를 허용하는 시퀀싱 기반 방법과 달리 epigenomic MERFISH는 표적 접근 방식이기 때문에 epigenomic loci를 선택하기 위해 사전 지식 또는 가설을 필요로 한다. 그러나 우리는 수천, 어쩌면 수만 개의 게놈 위치의 epigenomic 특성을 동시에 프로파일링하는 것이 가능해질 것으로 예상하며, 이는 이러한 한계를 완화시킬 것이다.
우리는 epigenomic MERFISH의 많은 응용을 예상한다. 여기서는 epigenomic MERFISH를 사용하여 active promoters 및 enhancer 또는 silent promoter를 표시하는 histone modification을 프로파일링했다. Tn5에 의한 현장 처리는 이러한 표시에 대한 항체 또는 다른 affinity probe가 존재하는 한 다른 epigenetic mark를 포착하기 위해 적용될 수 있다. 따라서, 우리는 epigenomic MERFISH가 히스톤과 DNA 수정뿐만 아니라 게놈 DNA를 따라 transcription factor와 non-coding RNA의 결합 패턴에 대한 공간적으로 해결된 단일 세포 프로파일링을 제공하는 많은 epigenomic 특성을 연구하는 데 적용될 수 있을 것으로 기대한다.
최근 연구에 따르면 PA-Tn5에 다른 epigenomic mark를 동시에 표적화하는 항체가 프리로드될 가능성이 입증되었다(Gopalan et al., 2021). 우리는 이 접근법이 epigenomic MERFISH에도 적용될 수 있어, 예를 들어 promoter and enhancer activity를 동시에 표시하여 세포 내 enhancer activity과 유전자 조절에 대한 보다 포괄적인 그림을 제공할 수 있을 것으로 예상한다.
이미징 접근법으로서, 우리는 epigenomic MERFISH가 3D 게놈 및 transcriptome 이미징과 같은 이미지 기반 omics 측정의 다른 양식과 결합하여 동일 세포내에서 크로마틴의 epigenetic and protein-binding 프로필, 크로마틴의 3D organization 및 유전자 발현 프로필을 동시에 detection 할 수 있도록 할 수 있다고 상상한다. 이러한 single-cell spatial multi-omics approach는 transcriptional regulation의 기초가 되는 메커니즘과 조직 발달 및 기능에서 유전자 조절의 역할에 대한 우리의 이해를 실질적으로 가속화할 것을 약속한다.