공간적으로 조직된 세포 커뮤니티가 발생 단계의 인간 심장을 형성한다
Abstract
심장은 첫 번째로 발달하는 기관으로, 그 형태에 따라 기능이 크게 좌우된다. 하지만, 다양한 심장 세포 유형이 어떻게 공간적으로 조정되어 심장 기능에 필수적인 복잡한 형태학적 구조를 만드는지는 명확하지 않다. 여기서 우리는 single-cell RNA-sequencing과 high-resolution multiplexed error-robust fluorescence in situ hybridization (FISH)을 통합하여 인간 심장을 발달시키는 심장 세포 유형의 정체성을 해결했다. 이 접근법은 또한 개별 세포의 공간적 매핑을 제공하여 그들이 독특한 심장 구조를 형성하는 세포 커뮤니티로 조직되는 방식을 밝힌다. 우리는 이러한 심장 세포 유형 중 많은 수가 특정 커뮤니티에 독점적인 세부 분화군으로 더 세분화되며, 이는 세포 생태계 및 해부학적 지역에 따른 그들의 전문화를 지원한다는 것을 발견했다. 특히, 심실 심근세포 세부 분화군은 심실 벽을 통해 예상치 못한 복잡한 층상 조직을 보이며, 다른 세포 세부 분화군과 함께 여러 세포 커뮤니티를 형성했다. 이 커뮤니티 내의 세포 간 상호작용을 생쥐 모델에서의 in vivo 조건부 유전적 모델과 인간 다능성 줄기 세포 시스템에서의 in vitro 모델을 사용해 조사함으로써, 심실 벽 형태형성 동안 심장 세포 세부 분화군의 공간적 조직을 조율하는 다세포 신호 경로를 밝혔다. 이러한 심장 세포 유형의 사회적 상호작용과 인간 심장을 구축 및 리모델링하는 전문화에 대한 상세한 발견은 구조적 심장 질환과 인간 심장 수리를 위한 복잡한 다세포 조직 공학에 대한 새로운 통찰력을 제공한다.
Figures
Cell lineages in the developing human heart
– 심장 기능 조절에 필수적인 복잡 구조의 형성을 연구하기 위해 임신 후 9-16주 사이의 인간 심장 발달 과정의 세포를 조사 및 식별함
– scRNA-seq을 통해 개별 세포 구획에서 주요 세포 클래스를 식별함.
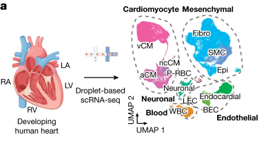
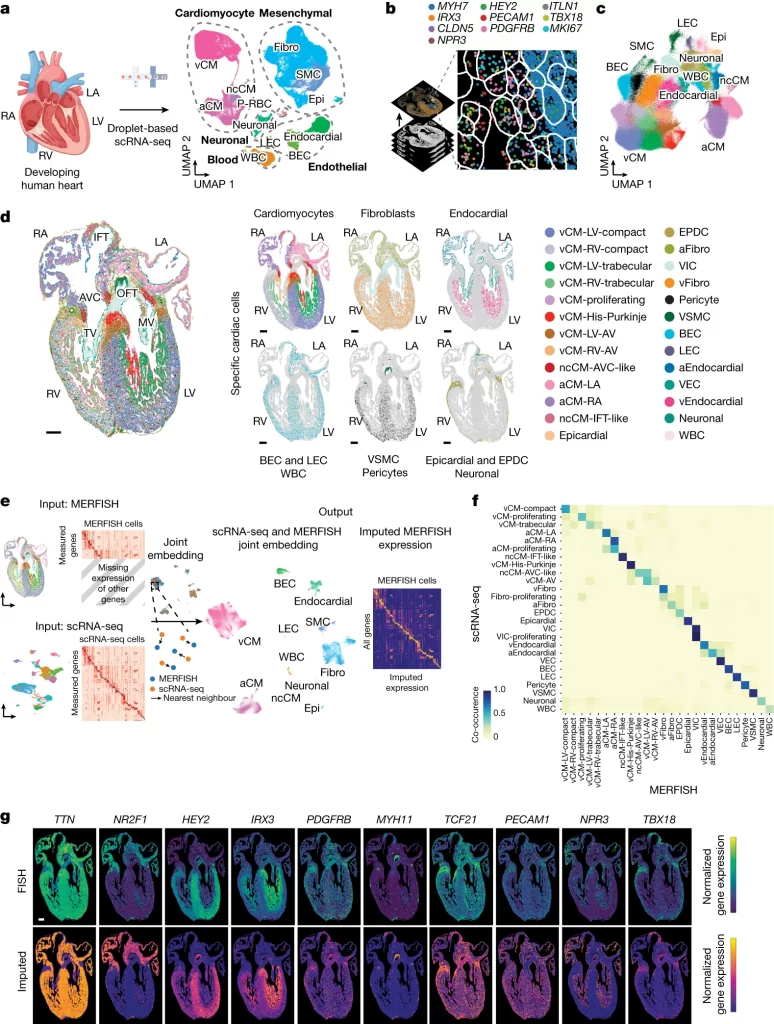
Fig. 1(A). molecular and spatial 인간 심장 세포 atlases는 심장 발달 동안 다양한 범위의 세포 집단을 밝혀낸다.
(A) (좌측) 실험의 개요도. (우측) scRNA-seq은 uniform manifold approximation and projection (UMAP)을 통해 약 143,000개의 세포로 표시된 발달 과정의 인간 심장을 만드는 다양한 범위의 독특한 심장 세포를 식별함.
[Fig 1A] 임신 후 9~16주 사이의 인간 심장을 해부한 후 scRNA-seq을 통해 5개 개별 세포 구획 (심근세포, 중간엽, 내피, 혈액, 신경세포) 및 12개의 주요 세포 클래스를 식별.
MERFISH spatially maps heart cells & Diverse cardiomyocytes in specific heart structures & Spatial relationships of heart lineages
– 앞서 조사한 다양한 심혈관 세포의 상호작용 및 복잡한 형태학적 공동체, 심장 기능 및 세포 전문화에 중요한 구조로 구성되는 과정을 확인하고자 함.
– *MERFISH 및 scRNA-seq 정보를 매칭, 심혈관 세포의 공간적인 구성을 더 다양한 유전자 수준에서 확인하여 심근세포가 심장 전체에 걸쳐 뚜렷한 지역적 및 구조적 분포를 나타냄을 밝힘.
*Multiplexed Error-Robust Fluorescence in situ Hybridization (MERFISH): 다조직 유래 개별 세포로부터 공간적으로 매핑된 표적 RNA (수백 개) 정량화를 수행할 수 있는 분석 방법.

Fig. 1(B-G). molecular and spatial 인간 심장 세포 atlases는 심장 발달 동안 다양한 범위의 세포 집단을 밝혀낸다.
(B) MERFISH를 사용하여 238개의 심장 세포 특이 유전자가 공간적으로 어떻게 식별되었는지 보여주는 개략도. 각 색상은 10가지 특정 RNA 전사체의 개별 분자 위치를 표시.
(C) MERFISH로 식별된 약 250,000개의 심장 세포들이 UMAP에 의해 특정 세포 집단으로 군집화. 군집화 결과는 (D)에서 실제 심장 절단면에 세포 분포를 나타내기 위해 사용됨.
(D) (좌측) 식별된 MERFISH 세포들이 13주차 심장의 전면 절단면에 공간적으로 매핑되었으며, (우측) 주요 세포 클래스에 따라 표시.
(E) MERFISH와 age-matched scRNA-seq 데이터 세트 간의 joint embedding을 통해 세포 라벨 transfer과 MERFISH 유전자 추정이 가능해짐.
(F) Co-occurrence heatmap은 MERFISH 세포의 cell annotation과 13주차 scRNA-seq 데이터 세트에서 transfer된 annotation의 correspondence를 보여줌.
(G) 유전자 추정 성능은 MERFISH로 측정된 마커 유전자의 normalized gene expression profile과 해당 cell의 추정 gene expression profile을 공간적으로 비교함으로써 검증.
[Fig 1B-D] MERFISH를 사용하여 공간에 따른 심장 세포의 세포 분포를 확인함. 심장 세포와 연관된 주요 gene들의 공간적 분포를 확인하고, 이를 통해 각 세포가 속한 클래스를 식별한 후 다시 공간 이미지에 매핑함.
[Fig 1E-G] MERFISH는 측정할 수 있는 유전자의 종류가 한정됨. 이를 확장하기 위해 scRNA-seq 데이터와 MERFISH에 공통으로 존재하는 유전자(238개)로 가장 유사한 MERFISH의 세포와 scRNA-seq의 세포를 매칭한 후 매칭된 세포들의 cell annotation, gene expression의 유사성을 확인함.
Cell communities form cardiac structures
– 심혈관 세포가 심장 기능에 중요한 조직화된 다중 세포 계통 구조를 형성하기 위해 어떻게 assemble되는지 분석.
– 이웃한 세포와 유사한 contents를 가지는 그룹을 cellular communities (CCs)로 정의하고 13개의 개별 CC를 식별. 각 CC는 특정 세포의 독특한 조합 및 양으로 구성되어 최소 1~2개에서 10개 이상의 세포 집단을 포함.
– 발달 중인 심실, 특히 심실 벽 CC의 경계 영역에서 세포의 다양성이 증대되며, 이은 발달 단계의 심실이 이전에 설명된 것보다 더 구분되는 심장 세포 및 복잡한 조직을 표현할 수 있음을 시사함.
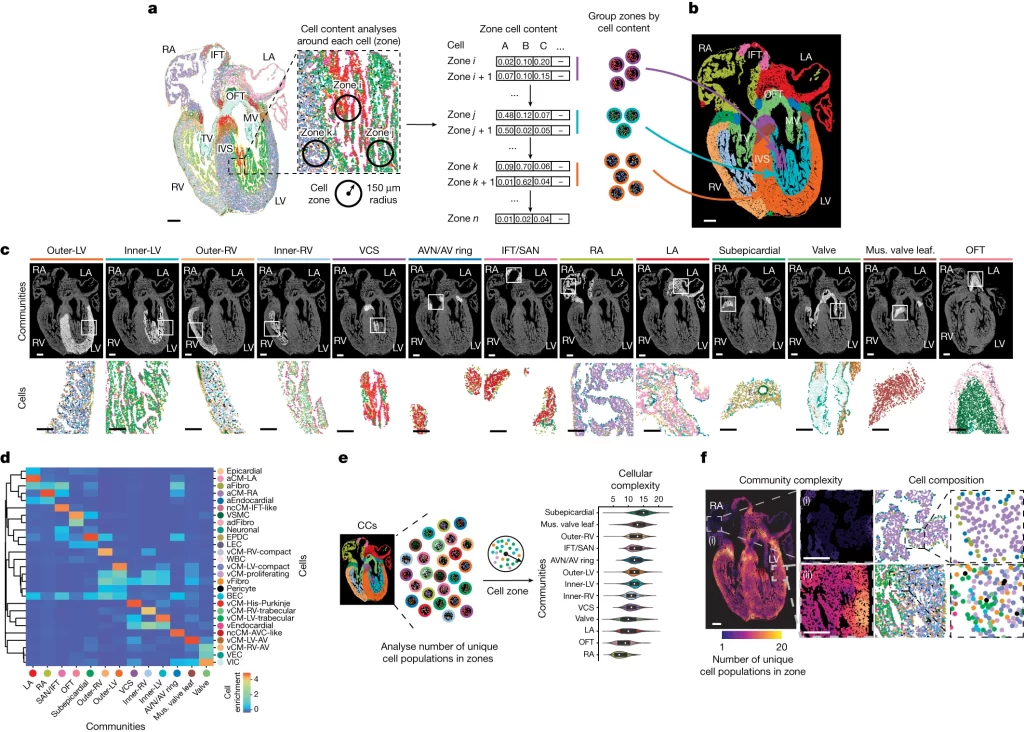
Fig. 2. 구별된 심장 세포 집단은 특수한 심장 구조를 형성하는 CC로 공간적으로 조직된다.
(A) 각 개별 세포 주변의 세포 contents에 대한 조사를 통해 정의된 CC를 형성하는 세포 구역 또는 이웃이 식별됨.
(B) 13주차 심장에 대한 CC의 공간 매핑은 그들이 구별된 해부학적 심장 구조에 해당함을 드러냄.
(C) 각 CC의 공간 위치를 세포 구성 및 분포의 예시와 함께 표시함.
(D) Heatmap은 정의된 각 CC 내에서 식별된 MERFISH 세포의 구성을 보여줌.
(E, F) 각 구역 내의 고유한 세포 집단의 수에 대한 분석. 각 CC와 심장 지역의 세포 복잡성을 (E) 정량적으로 보여주는 violin plot 및 (F) 공간적으로 보여주는 spatial complexity map.
[Fig 2A-C] 심장 이미지에서 식별된 CC들로 새롭게 심장 구조를 매핑.
[Fig 2D] CC를 구성하는 다양한 세포 구성을 보여줌.
[Fig 2E, F] 각 지역의 세포 복잡성을 보여주는 violin plot 및 complexity map에서 LV는 상대적으로 높은 복잡성을, RA는 가장 낮은 복잡성을 보여주고 있음. 지역별로 서로 다른 CC 및 세포 복잡성을 가질 수 있음을 의미함.
Multilayered organization of ventricles
– 심실 세포 및 조직의 복잡성을 탐색하기 위해 심실 세포들을 분리 및 식별하여 집중적으로 분석함.
– 좌심실이 우심실보다 먼저 발달하는 것과, 심실벽의 vCMs이 깊이별로 연속적인 집합체를 형성함을 밝힘.
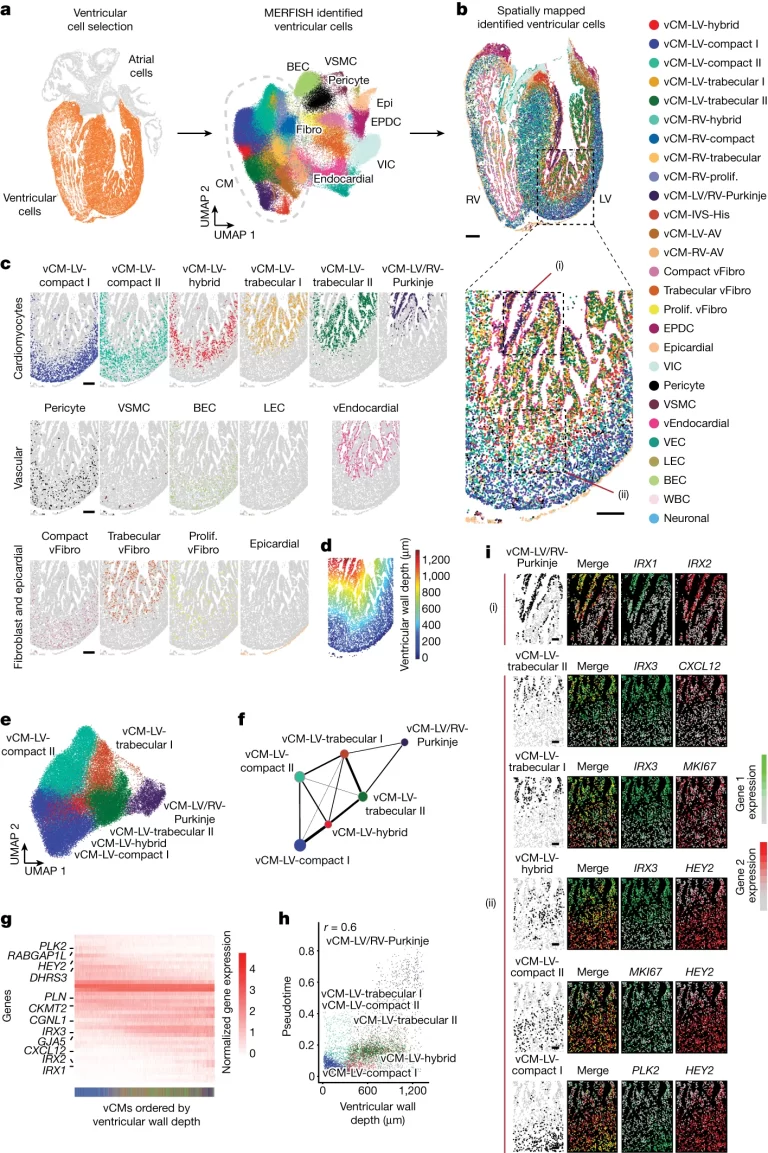
Fig. 3. 심실 벽에는 예상 밖의 복잡한 laminar layer로 공간적으로 조직된 구분된 특수 심장 세포가 포함된다.
(A) (좌측, 오렌지색) 심실을 구성하는 MERFISH 세포들이 (우측) UMAP을 사용하여 군집화됨.
(B) 식별된 심실 세포들이 13주차 심실에 공간적으로 매핑.
(C) (B)의 MERFISH 공간 지도에서 윤곽이 그려진 지역의 좌측 심실벽에 대한 특정 심실 세포들의 공간 분포.
(D) (B)의 이미지 영역에 대해 심실의 심장외막/외부 표면으로부터 측정된 거리로서 심실 세포의 심실벽 깊이 분포를 보여줌.
(E) LV vCMs가 구별된 vCM 소집단으로 분리됨.
(F) 유전자 발현 유사성에 기반한 연결성을 나타내는 nodes간 weighted edge. 이를 통해 구별된 vCM의 분자 관계가 연결성 지도에서 보여짐.
(G) 심실벽 깊이가 증가함에 따라 정렬된 vCM의 Differentially expressed gene의 normalized expression을 보여주는 heatmap. 하단의 색깔 바는 (B)에서 표시된 특정 vCM을 나타냄.
(H) 왼쪽 심실에서 개별 vCM의 심실벽 깊이와 pseudotime 사이의 관계를 밝히는 scatter plot.
(I) (C)에서 윤곽이 그려진 지역의 MERFISH 이미지는 녹색과 빨간색으로 보여진 특정 유전자 마커의 조합이 특정 vCM을 공간적으로 식별함을 보여줌.
[Fig 3A, B] 심실 세포만을 MERFISH로 분류한 후 세부적인 세포 클래스를 다시 확인 및 매핑함.
[Fig 3C, D] 심실 세포별로 심실 내 위치를 식별하고, 심실벽 깊이의 분포를 확인하여 심실의 다층 조직이 우심실보다 좌심실에서 더 많은 층과 vCM을 포함하는 것을 확인함. 이는 좌심실이 우심실보다 더 일찍 발달한다는 증거임.
[Fig 3E-G] 좌심실의 vCM의 복잡성 및 laminar layer를 이해하기 위해 해당 세포들을 분리하여 분석함. 인접한 vCM 사이의 강력한 연결성 및 높은 유사도의 유전자 발현을 확인함.
[Fig 3H] 두 가지 유전자의 조합으로 vCM들을 정의한 후 해당 gene들의 pseudotime에 따른 expression 변화를 확인 (extended figure 6c) 한 결과 vCM의 순서가 심실 벽 깊이에 따라 배열되는 연속적인 집합체를 형성함을 확인.
Multicellular signaling forms ventricles
– 성인 및 선천성 심장 질환을 유발할 수 있는 외부의 치밀층 (outer compac) 및 내부의 소주층 (inner trabecular layers) 등 심실 벽 발달 단계의 결함을 이해하기 위해 인간의 심장 심실이 재구성되기 시작하는 12주~13주차의 심근층 구성 과정을 조사함.
– 심실 내의 세포 분포를 CC 단위로 묶어 분석함으로써 세포 구성의 이질성 및 이에 관여하는 신호 전달 과정을 조사함.
– 다양한 상호작용 중 심실 벽 vCM 구성에 영향을 줄 수 있을 요소로 PLXN-SEMA 신호 전달 pathway 등을 식별함.
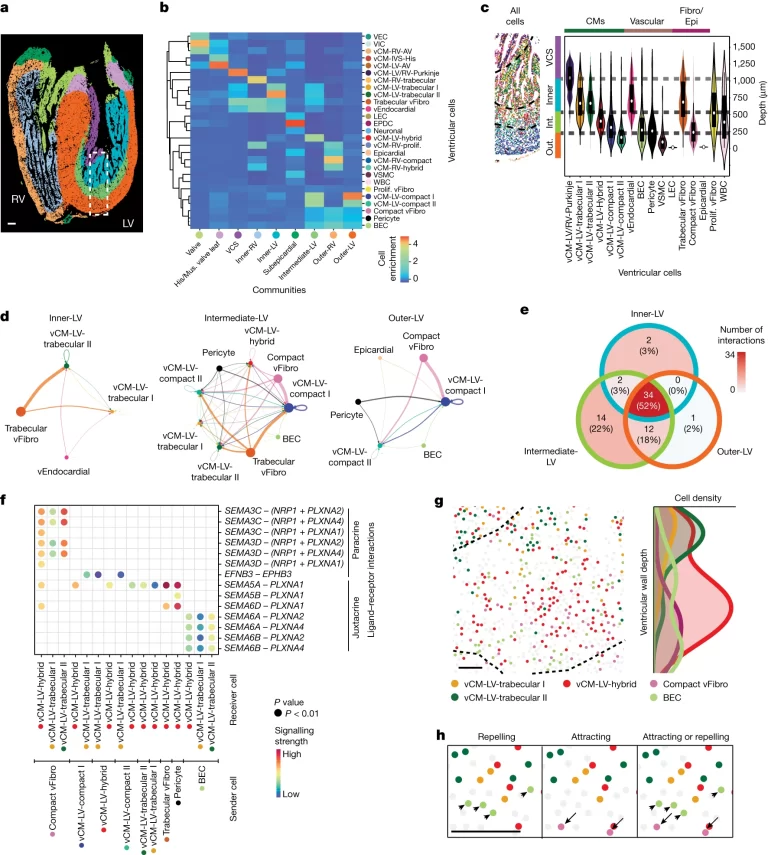
Fig. 4. i다세포 상호작용이 심실벽 내 특정 CC의 조직을 지시함
(A) MERFISH로 식별된 심실 세포들이 심실 내에서 9개의 더 세분화된 CC로 assemble됨.
(B) Heatmap은 각 심실 CC 내에서 구별된 심실 세포의 구성을 보여줌.
(C) (A)에서 윤곽이 그려진 영역의 MERFISH 이미지는 CC 층과 그들의 세포 구성을 드러냄. Violin plot은 이러한 층 내에서 구별된 심실 세포의 심실벽 깊이 분포를 보여줌. 중앙의 흰색 점은 중앙값을, 굵은 검은색 선은 사분위 범위를, 가장자리는 분포의 최소값과 최대값을 정의. 점선은 CC 층의 경계를 나타냄.
(D) code diagram은 내부-LV, 중간-LV 및 외부-LV CC에서 특정 vCM이 받는 세포 간 신호 전달 상호작용의 강도를 보여줌. node의 크기는 구별된 심실 세포의 세포 수를 나타내며, edge의 너비는 특정 심실 세포 쌍 간의 상호작용 강도를 나타냄.
(E) 벤 다이어그램은 내부-LV, 중간-LV 및 외부-LV 커뮤니티 내에서 vCM이 받는 특정 및 공유된 CCI의 수를 보여줌.
(F) dot plot은 중간-LV CC 내에서 구별된 심실 세포 간의 특정 신호 전달 상호작용을 보여줌.
(G) (좌측) 중간-LV CC에 대해 SEMA3C, SEMA3D, SEMA6A 또는 SEMA6B와 PLXNA2 또는 PLXN4 간의 상호작용에 참여하는 세포들의 공간 지도. (우측) 이러한 세포의 정규화된 심실벽 깊이 분포를 보여주는 히스토그램.
(H) 중간-LV CC의 고해상도 공간 세포 지도는 SEMA3C, SEMA3D, SEMA6A 또는 SEMA6B와 PLXNA2 또는 PLXN4 신호와 상호작용하는 세포들이 유인 또는 반발 상호작용을 매개하기 위해 공간적으로 어떻게 분포될 수 있는지 보여줌. 화살표와 화살표 머리는 각각 SEMA3C+SEMA3D+ 밀집 vFibro 세포와 SEMA6A+SEMA6B+ BEC를 가리킴.
[Fig 4A-C] Figure 3의 분석과 동일하게 심실을 대상으로 하지만, 개별 세포를 CC 단위로 묶어 분석을 진행하는 점에서 차이가 있음. 심실 벽의 세포를 CC 단위로 통합한 후, 깊이에 따른 CC 층별로 세포 구성을 조사함. 좌심실, 특히 중간 layer가 높은 이질성을 지녀 심실 벽의 동적 발달 및 리모델링을 조절하는 복잡한 상호 작용 다세포 사건이 발생할 수 있다고 추론함.
[Fig 4D] 심실 심장 세포가 어떤 협력 과정으로 성숙한 심실 벽을 구성하는지 살펴보기 위해 cell-cell interaction 분석을 통해 신호 전달 과정을 조사함. 광범위한 심실 심장 세포 중 섬유아세포가 vCM에 가장 활발한 신호를 보내 좌심실 벽 발달에 중요한 역할을 할 수 있음을 시사함.
[Fig 4E, F] 서로 다른 vCM에 의해 차등적으로 존재하는 interaction을 식별함. 특히 중간 LV CC에서 세포 이동 조절에 기여할 수 있는 여러 PLXN–SEMA axon guidance signalling pathways를 식별함.
[Fig 4G, H] 심실 벽 세포가 LV의 벽을 가로질러 상보적으로 분포하면서도, 중간 LV 내에서 병합되는 것을 관찰함. 또한, LV CC 내에서 특정한 세포 클래스의 존재를 확인함.
PLXN–SEMA directs ventricle organization
– PLXN-SEMA 신호 전달 경로가 심실 벽 내 vCM 구성에 기여하는지 확인하기 위해 3D 바이오 프린팅 기술을 통해 다층 심실 벽 모델을 개발하고 관련 유전자의 기여를 확인함.
– SEMA의 종류에 따라 바이오프린팅 모델에서 vCM의 심실 벽으로의 이동을 촉진 또는 방해할 수 있었으며, mouse 실험에서 특정 SEMA gene의 deletion이 실제 심실 벽의 발달 결함으로 이어질 수 있음을 밝힘.
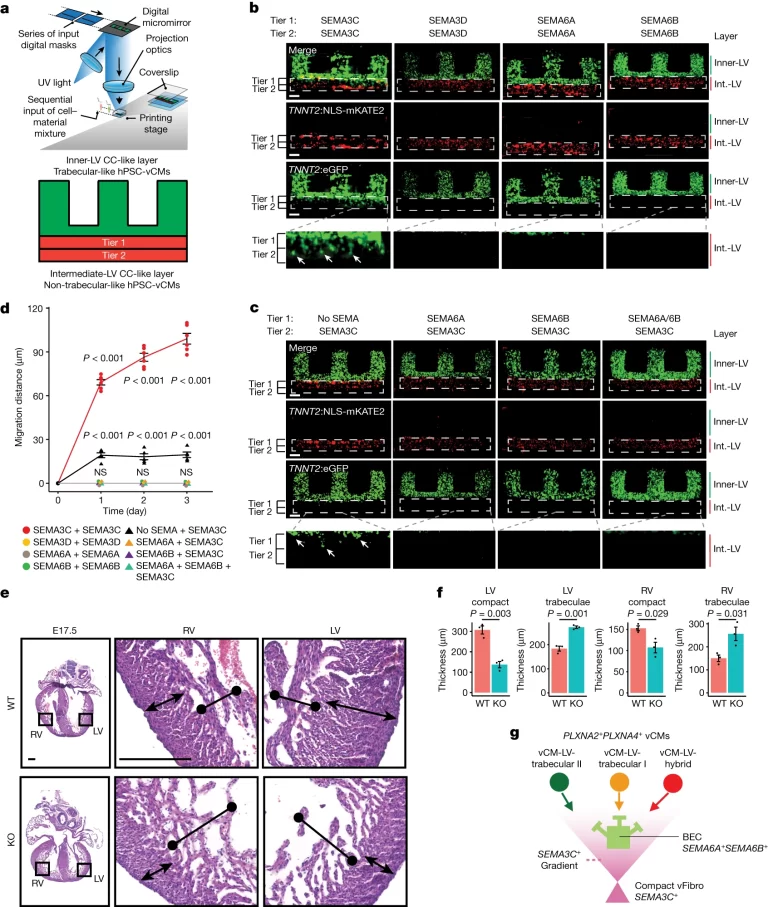
Fig. 5. PLXN–SEMA 신호 전달은 trabecular vCM의 이동을 매개한다.
(A) NLS-mKATE2+ non-trabecular 및 GFP+ trabecular-like hPSC-vCM들이 다이어그램에 표시된 대로 심실벽을 모델링하는 다층 구조물로 바이오프린팅 됨.
(B) GFP+ trabecular-like hPSC-vCM들은 NLS-mKATE2+ non-trabecula-like hPSC-vCM들과 SEMA3C가 혼합된 중간 심실 유사층(Int.-LV)으로 이동하지만, SEMA3D, SEMA6A 또는 SEMA6B와 혼합되었을 때는 그렇지 않음. 하얀 화살표는 중간 심실 유사층으로 이동하는 GFP+ trabecular-like hPSC-vCM을 가리킴.
(C) 중간 심실 유사층에서 다양한 조합으로 혼합된 SEMA6A 또는 SEMA6B는 SEMA3C 매개 GFP+ trabecular-like hPSC-vCM의 이동을 방지. 하얀 화살표는 중간 심실 유사층으로 이동하는 GFP+ trabecular-like hPSC-vCM을 가리킴.
(D) 다른 중간-LV CC 유사층 조건 하에서 GFP+ trabecular-like hPSC-vCM 이동 측정. SEMA3C+SEMA3C 조건에 대해 N=6회, no SEMA+SEMA3C 조건에 대해 N=5회, 모든 다른 조건에 대해 N=3회 독립 실험.
(E) E17.5 야생형 (WT) 및 Tcf21-creERT2;Sema3cfl/fl Knockout (KO) 마우스 배아의 심장 절편은 E10.5부터 Tcf21+ 세포에서 Sema3c deletion이 심장 심실벽의 밀집되지 않은 형태로 이어진다는 것을 보여줌.
(F) WT 및 조건부 deletion된 Sema3c KO 마우스 심장의 조밀하고 trabecular 심근의 두께를 보여주는 그래프. 조건 당 N=3 마리의 마우스.
(G) 모델은 다른 vCM, 섬유아세포 및 내피세포 간의 PLXN–SEMA 상호작용이 심실벽 내에서 vCM의 조직을 어떻게 조정하는지 보여줌.
[Fig 5A-C] 좌심실 벽의 중간 및 내부에 각각 위치하는 CC들을 바이오프린팅 기술로 재현함. 중간 LV CC에서 유래한 서로 다른 SEMA 분자를 두 개의 서로 다른 층에 배열하여 어떤 SEMA 종류가 심실 벽의 형성 과정에 기여하는지 확인함.
[Fig 5D] SEMA3C만이
vCM이 중간LV CC로 정상적으로 이동하는 데 기여했으며, 1) SEMA3C 외의 다른 종류의 SEMA를 첨가한 경우, 또는 2) 내부 LV와
직접 맞닿은 tier 1에 다른 종류의 SEMA를 첨가하거나
SEMA가 존재하지 않는 경우 모두 정상적인 세포 이동이 이뤄지지 않음.
[Fig 5E, F] Mouse
실험에서 실제로 Sema3c의
deletion이 심실 벽 발달의 결함으로 이어지는 것을 확인.
[Fig 5G] vCM의 재배치 메커니즘 정리. SEMA3C+ compact vFibro
cell이 심실 벽으로 배치되는 것을 촉진하며, SEMA6+ SEMA6B+ BEC는 vCM의 이동을 오히려 방해할 수 있음.
Disscussion
우리의 단일 세포 심장 multimodal 연구는 scRNA-seq와 MERFISH 이미징의 결합된 힘을 활용하여 spatial & molecular single-cell resolution에서 발달 과정에 있는 인간 심장의 포괄적인 세포 atlas를 구축했다. 이러한 multimodal 분석은 심장 발달과 형태 형성에 참여하는 다양한 심혈관 계통을 밝혀냈다. 또한, 연구 결과 심장 판막과 전도 시스템과 같이 중요하지만 과소평가된 심장 부위에서 새로운 심장 세포 집단을 발견하는 데 기여함으로써 인간 심장을 구성하는 세포 유형과 상태에 대한 현재 지식을 확장했다(Supplementary Discussion). 이러한 세포 집단이 그들의 세포 및 지역 환경에 따라 어떻게 전문화되는지 이해하기 위해, 우리는 MERFISH 기반 high-resolution 공간 심장 세포 atlas를 분석했으며, 이는 구별되는 심장 구조와 관련된 CC 내에서 형성되고 상호 작용하는 개별 세포들이 무엇인지 질문할 수 있게 했다. 이 MERFISH 이미징 분석을 해당 scRNA-seq 데이터와 통합하여 spatillay-resoved된 개별 세포들의 transcriptional profile과 고유한 유전자의 imputation을 밝혔다.
이 특정 유전자들을 CCI 알고리즘으로 검사하는 것은 공간적으로 인접한 세포 집단 사이에 상호 작용을 매개하는 것으로 표현된 구별되는 세포 신호 ligand-receptor 쌍을 식별하는 데 도움이 되었다. 이러한 식별된 신호 경로들은 심장 전반에 걸쳐 다양한 세포 유형에서 예측되었지만, 우리는 그것들이 구별되는 CC 내의 특정 CCI 사이에서 차별적으로 발생한다는 것을 발견했다. 예를 들어, 우리는 심실벽의 특정 층 내에서 상호 작용하는 여러 조합의 세포 집단 사이에 PLXN–SEMA 신호 경로가 구별되는 것을 관찰했으며, 이는 이전에 심실에서 보고된 plexins와 semaphorins를 포함했다. 그러나, 우리는 또한 PLXNA2+PLXNA4+ 심실 심근세포, SEMA3C+SEMA3D+ 섬유아세포 및 SEMA6A+SEMA6B+ 내피세포 사이에 이전에 특성화되지 않은 다세포 상호 작용을 식별했으며, 이는 심실벽 압축의 중요한 형태학적 과정 동안 심근세포의 할당을 제어할 수 있다(Fig. 5g 및 Supplementary Discussion). 전반적으로, 이러한 발견들은 우리의 high-resolution molecular & spatial 심장 세포 atlas가 심장 기능 유지에 필수적인 심장 구조로 전문화되고 조직되는 구별되는 세포 유형 간의 상세한 사회적 상호 작용에 대한 통찰력을 제공하는 방법을 강조한다. 이러한 정보는 미래에 선천적 및 성인 구조적 심장 질환의 병리학적 메커니즘을 이해하는 데 사용될 뿐 아니라, 심장 치료를 위한 복잡한 다세포 심장 조직을 엔지니어링하는 새로운 전략을 개발하는 데 사용될 수 있다.