공간 유전체학은 암 클론의 구조, 특성, 진화를 매핑합니다
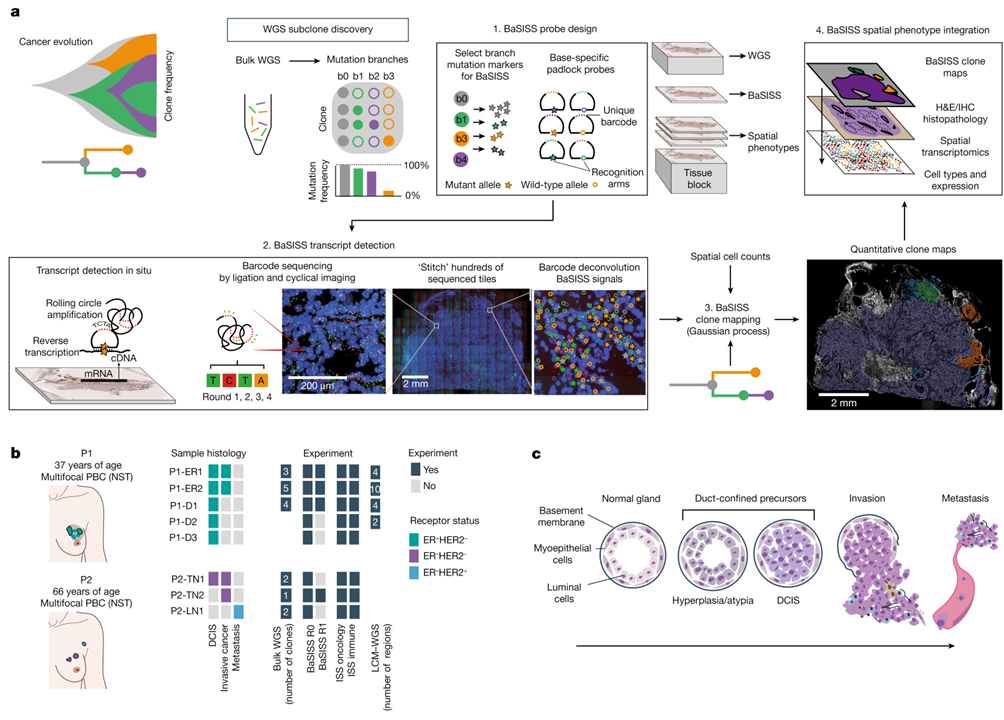
암의 게놈 시퀀싱은 종종 동일한 종양에 존재하는 서로 다른 서브클론의 모자이크를 보입니다. 이들은 체세포 진화의 원리에 따라 발생한다고 생각되지만 정확한 공간적 성장 패턴과 기본적인 메커니즘은 파악하기 어렵습니다.
우리는 이러한 요구를 해결하기 위해 전체 종양 섹션에 걸쳐 유전적 서브클론 구성의 상세한 정량적 지도를 생성하는 워크플로우를 개발했습니다. 이 워크플로우는 각 클론의 클론 성장 패턴, 조직학적 특성, 미세해부학 및 미세환경 구성을 연구하기 위한 기초를 제공합니다. 이 접근 방식은 전체 게놈 시퀀싱에 이어 고도로 다중화된 염기별 in situ 시퀀싱, 단일 세포 분해 transcriptomics 및 이러한 레이어를 연결하는 전용 알고리즘에 의존합니다.
2개의 multifocal primary breast cancer의 8개 조직 절편에 염기별 in situ 시퀀싱 워크플로우를 적용한 결과 microdissection에 의해 검증된 복잡한 하위 클론들의 성장 패턴이 밝혀졌습니다. Ductal carcinoma in situ의 경우, polyclonal neoplastic expansion은 거시적 규모에서 발생했지만 미세 해부학적 구조 내에서 구분되었습니다. Ductal carcinoma in situ, 침윤성 암, 림프절 전이의 단계에 걸쳐 서브클론 영역은 뚜렷한 전사체적, 조직학적 특징과 세포 미세 환경을 보이는 것으로 나타났습니다.
이러한 결과는 암 진화 및 미세 환경 생태학의 기본 메커니즘을 이해하기 위해 공간 유전체학이 제공하는 이점의 예를 보입니다.
