암의 돌연변이 과정에 의해 유도된 single-cell genomic variation
Abstract
인간 암의 유전적 불안정성을 뒷받침하는 cell-to-cell copy number alteration이 어떻게 유전형과 표현형 변이를 유도하고 결과적으로 암의 진화를 유도하는지에 대해서는 연구되지 않은 상태로 남아 있습니다. 이 연구에서는 스케일링된 single-cell whole-genome sequencing을 wild-type, TP53-deficient, TP53-deficient;BRCA1-deficient, TP53-deficient;BRCA2-deficient mammary epithelial cell (13,818 genomes)와 primary triple-negative breast cancer (TNBC), high-grade serous ovarian cancer (HGSC) (22,057 genomes)에서 진행했고 세포 간 구조적 변이에 의해 정의되는 3개의 뚜렷한 ‘foreground’ 돌연변이 패턴을 식별했습니다. Cell-specific과 clone-specific high-level amplification, parallel haplotype-specific copy number alterations, copy number segment length variation (serrate structural variations)는 측정 가능한 표현형 및 진화적으로 유의미한 결과를 가졌습니다. TNBC와 HGSC에서 알려진 종양유전자의 clone-specific high-level amplification은 homologous recombination deficiency를 가진 종양에 비해 fold-back inversion이 있는 종양에서 매우 만연했으며 클론 간 표현형의 변이 증가와 관련이 있었습니다. Parallel haplotype-specific alterations도 일반적으로 관찰되어 phylogenetic evolutionary diversity와 clone-specific mono-allelic expression으로 이어졌습니다. Serrate variants는 fold-back inversions이 있는 종양에서 증가했으며 세포 집단의 증가된 유전적 다양성과 높은 상관관계가 있었습니다. 우리의 연구 결과는 세포 간 구조적 변이가 TNBC 및 HGSC의 표현형 및 진화적 다양성의 기원에 기여하고 개별 암세포의 게놈 및 돌연변이 상태에 대한 통찰력을 제공한다는 것을 보여줍니다.
Figures
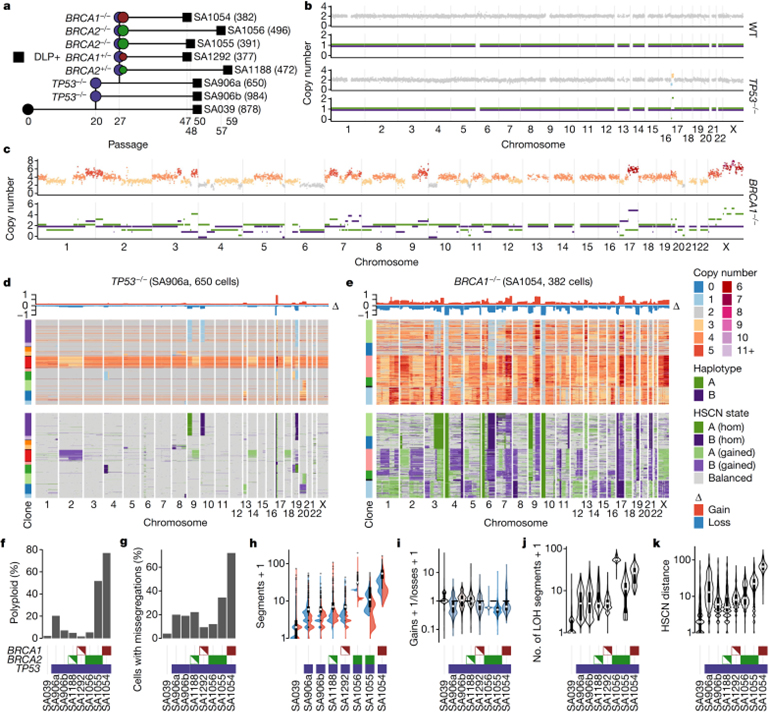
Fig 1. 184-hTERT mammary epithelial cell line의 CRISPR–Cas9 유래 isogenic genotype의 single-cell genome 특성.
(A) WT → TP53 → BRCA1/BRCA2 대립형질을 보여주는 genotype lineage diagram.
(B, C) WT, TP53-/- (b)와 BRCA1-/- (c) DLP+로 시퀀싱된 184-hTERT single-cell genome.
(D, E) TP53-/- (d)와 BRCA1-/- (e) 계통의 세포 집단에서 copy number profiles에 대한 heat map.
(F-K) Polyploidization 속도 비교 (f), chromosome missegregation이 있는 세포의 비율 (g), gain이나 loss가 있는 segment 분포 (h), gain/loss 비율에 대한 분포 (i), heterozygosity를 상실한 segment 수 (j), 250개의 subsampled 세포 사이의 pairwise HSCN 거리 분포 (k).
Fig 2. Single-cell genome에서 세포 간 변이를 생성하는 프로세스.
(A) 세포의 총 비율에서 세포 당 HLAMP의 수.
(B-D) 세포주에서 발견되는 Oncogenic amplification.
(E) SA906b의 parallel copy number 증가를 보이는 heat map.
(F) e의 두 가지 세포에서의 분포.
(G) 184-hTERT mammary epithelial cell line에서 세포 당 parallel copy number 사건의 수.
(H) 세포 사이에 breakpoint 위치 변화를 보여주는 copy number heat map.
Fig 3. PDX 모델과 환자 조직에서의 single-cell genome 특성.
(A) TNBC HRD-Dup (SA501)과 HGSC FBI (SA1049)로부터 2개의 single-cell에 대한 총 copy number (상단), HSCN (하단) 프로파일.
(B) SA1049의 1,283개 세포에 대한 total copy number heat map (왼쪽), HSCN heat map (오른쪽).
(C, D) polyploid 세포의 비율 (c)과 missegregation event가 있는 세포 비율 (d).
(E) 상이한 ploidy state와 mutational signature 그룹에 걸친 chromosomal gains vs losses의 비율.
Fig 4. HLAMP copy number variation.
(A) FBI 종양 (SA1049) 클론 A와 C에서 chromosome 12에 대한 클론 및 single-cell whole-genome consensus copy number 프로파일.
(B) SA1049로부터 얻은 scRNA-seq 데이터의 Uniform manifold approximation and projection (UMAP) 차원 감소를 보이는 유전자 발현 cluster와 KRAS cluster 당 유전자 발현의 violin plot.
(C) Primary human tissue (왼쪽), PDX tissue (오른쪽)에서 KRAS 단백질의 immunohistochemistry staining.
(D) 각 데이터 세트 내의 HLAMP bin에 대한 세포 간 copy number 차이.
(E) 8개의 HRD-Dup case vs 12개의 FBI case에서 mean copy variance의 분포.
(F) 증폭을 포함하는 genomic segment의 너비.
(G) 유전자당 maximum cell copy number (CN).
(H) HLAMP 영역과 겹치는 암 유전자의 최대/최소 copy number 비율.
(I) 가변 종양 유전자 vs 비가변 종양 유전자에 대한 일치하는 scRNA-seq의 유전자 발현 cluster 사이의 최대 logFC 분포.
(J) SV를 나타내는 라인이 겹쳐진 SA1096의 2개 클론에서 consensus copy number 프로파일.
Fig 5. Haplotype-specific parallel copy number 진화.
(A-C) 계통수에 의해 정렬된 chromosome 1q (a), 10q (b), 8 (c)의 heat map.
(D) 우성 대립 유전자가 두 상태 사이를 전환하는 두 가지 haplotype-specific states에서 parallel copy number event에 대한 SNV의 VAF.
(E) SA535 chr의 데이터를 계산적으로 혼합한 후 대립유전자 A에 클론으로 존재하는 돌연변이의 VAF.
(F) Chromosome 10의 말단에 있는 유전자의 대립형질 상태에 대한 SA1053의 scRNA-seq 데이터 UMAP.
(G) f에서 세포의 BAF (B-allele frequency) 분포.
(H) RNA vs DNA에서 계산된 모든 데이터 세트에 걸쳐 segment 당 평균 BAF의 scatter plot.
(I) 일치하는 scRNA가 있는 모든 종양 및 cell line에서 chromosome 17 상의 BAF 분포 vs WT cell line에서의 분포.
(J) 2배체(1|1)와 4배체(2|2)에서 전체 chromosome, chromosome arm, segment 내의 gain, loss 비율.
(K, L) Copy number distance (k), phylogenetic distance (l)와 parallel copy number event 수의 상관관계.
Fig 6. Breakpoint serriform 가변성.
(A) 수직 축을 따라 단일 세포에 걸쳐 수평 축에서 breakpoint 위치의 변화를 보여주는 숫자 heat map.
(B) a. heat map 상단의 세포 번호는 프로파일 오른쪽에 표시하여 분석. 상단: 총 copy number & 하단: HSCN.
(C) FBI (갈색), TD (빨간색), HRD (파란색)로 분리된 점수를 계산할 수 있는 모든 암 데이터 세트에 대한 breakpoint serration distribution.
(D) 경우에 따른 serration score의 분포.
(E) 케이스당 serration score 대 배수체 세포 백분율.
(F) 케이스당 평균 세포간 HSCN 거리 (짝수 염색체만) 대 serration (홀수 염색체만).
Discussion
우리의 연구 결과는 구조적인 면과 copy number alteration (CNA)의 정도에서 세포 간 변이가 유전적 불안정성의 뚜렷한 endogenous mutation 과정에 대해 나타나는 TNBC와 HGSC 암의 만연한 ‘foreground’ 특징임을 보여줍니다. CNA는 수백 개의 유전자 발현 수준에 영향을 미칠 수 있기 때문에 foreground mutation 패턴 각각은 selection이 작용할 수 있는 광범위하고 뚜렷한 유전적 다양성을 제공합니다. 발암성 HLAMP는 종양 진행의 주요 원인으로 알려져 있으며 FBI와 함께 co-localization 될 때 HGSC에서 예후가 됩니다. 이 연구에서 HLAMP의 진폭이 세포 간에 상당히 다를 수 있음을 발견하면서 복잡성의 추가적인 계층을 보였습니다. 이것이 extrachromosomal DNA 증폭을 정의할 수 있는 특징으로 인식되었지만, 우리는 이것이 BFBC, inter-chromosomal rearrangement processes에 의해 매개되는 것과 같은 다른 부류의 HLAMP의 일반적인 특성이기도 하다고 제안합니다. 이것은 높은 유전적 불안정성을 가진 암 유형이 치료에 내성을 가진 클론을 포함하는 경향이 있기 때문에 자주 변경되는 종양 유전자를 표적으로 하는 치료 전략에서 중요한 의미를 갖습니다. 동일한 locus 내에서 Multi-allelic variation은 유방암 및 난소암의 매우 만연한 특징이며, 이는 다른 암에서 이전에 알려진 일부 연구 결과와 일치합니다.
특히, 총 copy number 수준에서 클론으로 나타나는 이벤트는 종종 얻거나 잃는 서로 다른 대립형질을 가진 별개의 분기군으로 구성됩니다. 이것은 전사 표현형 수렴에 의해 나타난 바와 같이 총 copy number 수준에서 유리한 karyotype에 대한 진화적 수렴을 반영할 수 있습니다. Evolutionary time series modelling은 병렬 CNA에서 표현형 선택 패턴을 해결하는 것에 추가적인 도움이 될 것입니다. 우리는 이배체에 비해 전체 유전자가 두 배인 상황에서는 비율이 증가하여 유전자 중복으로 인해 증가된 적합성 내성을 잠재적으로 반영하는 두 대립형질 모두에서 산발적인 이득과 손실이 발생한다는 점을 강조합니다. 마지막으로, single-cell level (SSV)에서 megabase 규모의 copy length variation이 cell-selected single-cell sequencing으로 in vitro에서 관찰되었습니다. 우리는 SSV가 실제로 TNBC와 HGSC에서 널리 퍼져 있고 종양 내의 클론에 걸쳐 분포한다는 것을 cell-population level에서 single-cell genome sequencing으로 보여줍니다. SSV를 생성하는 기본 메커니즘은 알려져 있지 않지만 FBI background와 polyploid genome states가 풍부한 종양의 구조적 복제 진화에 기여할 수 있는 새로운 종류의 변이를 보입니다. 우리는 모든 돌연변이 과정에서 foreground mutation 패턴 각각을 관찰했지만 FBI-type 종양은 세 가지 foreground mutation 패턴 모두에서 상당한 enrichment를 보여주었습니다. 이와 같이, FBI는 foreground mutation 패턴이 불량한 예후에서 다양성을 생성하는 별개의 표현형 클래스를 포함하여 중요하게 작용할 수 있습니다. 우리는 확장된 single-cell sequencing이 유전적으로 불안정한 종양에서 structural copy number diversity의 숨겨진 세포 상태를 밝히는 유용한 수단이라고 결론지었습니다. 우리가 여기에 제시하는 데이터는 foreground mutation 패턴이 암에서 유전체로 인코딩된 표현형의 다양성과 결과적으로 ‘evolvability’의 핵심 결정 요인임을 보여줍니다.