성체 생쥐 뇌의 Single-cell DNA 메틸옴 및 3D 다중-오믹스 아틀라스
Abstract
Cytosine DNA 메틸화는 뇌 발달에 필수적이며 다양한 신경학적 장애와 관련이 있습니다. 전체 뇌에서 DNA 메틸화 다양성을 공간적 맥락에서 이해하는 것은 뇌 세포 유형과 그들의 유전자 조절 환경의 완전한 분자 아틀라스를 위해 필수적입니다.
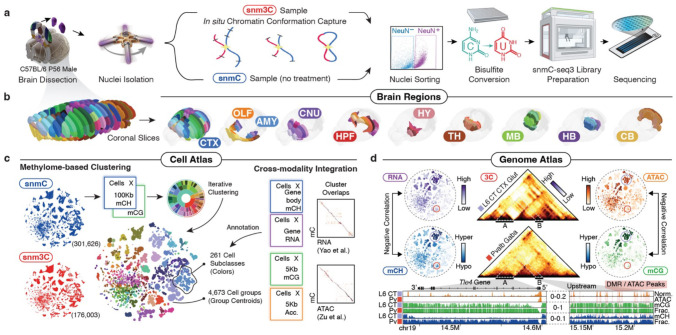
여기에서 우리는 단일핵 메틸화 염기서열 분석 (snmC-seq3)과 다중-오믹 염기서열 분석 (snm3C-seq) 기술을 사용하여 성체 생쥐 뇌의 117개 해부된 영역에서 301,626개의 메틸화 게놈과 176,003개의 크로마틴 구조-메틸화 공동 프로필을 생성했습니다. 반복 클러스터링을 사용하고 전체 뇌 전사체 및 크로마틴 접근성 데이터 세트와 통합하여, 우리는 4,673개의 세포 그룹과 274개의 교차 모달리티-주석된 하위 분류를 가진 메틸화 기반 세포 분류법을 구축했습니다.
우리는 유전자 조절 요소를 나타내는 2.6백만 개의 차등적으로 메틸화된 영역을 전체 게놈에서 식별했습니다. 특히, 우리는 뇌 영역 내부와 걸쳐 있는 세포 유형에서 유전자 및 조절 요소의 공간적 Cytosine 메틸화 패턴을 관찰했습니다. 뇌 전체 공간 전사체 데이터는 공간적 후성유전적 다양성과 유전자 발현의 관계를 확인하고 우리의 후성유전적 데이터 세트의 해부학적 매핑을 향상시켰습니다. 또한, 중요한 뉴런 유전자에서 크로마틴 구조 다양성이 발생하며 DNA 메틸화 및 유전자 발현 변화와 높은 연관성을 가지고 있습니다. 뇌 전체 세포 유형 비교는 전사 인자, 조절 요소 및 잠재적인 하류 유전자 목표를 포함하는 조절 네트워크 구축을 가능하게 했습니다. 마지막으로, 유전자 내부의 DNA 메틸화 및 크로마틴 구조 패턴은 전체 뇌 SMART-seq2 데이터 세트에서 관찰된 대안적 유전자 아이소폼 발현을 예측했습니다.
우리의 연구는 뇌 전체, 단일 세포 DNA 메틸화 및 3D 다중-오믹 아틀라스를 확립하고 생쥐 뇌의 세포-공간 및 조절 게놈 다양성을 이해하는데 귀중한 자원을 제공합니다.