고품질diploid human reference genome의 반자동 조립
현재 human reference genome인 GRCh38은 고품질의 assembly를 만들기 위해 20년 이상의 노력했으며, 이는 사회에 혜택을 주었다. 그러나, 그것은 여전히 많은 차이와 오류를 가지고 있고, 다수의 개체의 혼합이기 때문에 생물학적 게놈을 나타내지 않는다. 최근, 최신의 long-read technology로 고품질의 telomere-to-telomere reference CHM13이 생성되었지만, 그것은 거의 homozygous genome을 가진 hydatidiform mole cell line으로부터 파생되었다.
이러한 한계를 해결하기 위해 Human Pangenome Reference Consortium은 인간의 유전적 다양성을 나타내는 Pangenome Reference를 위한 고품질, 효율적인 비용, diploid genome assembly를 만드는 것을 목표로 결성되었다.
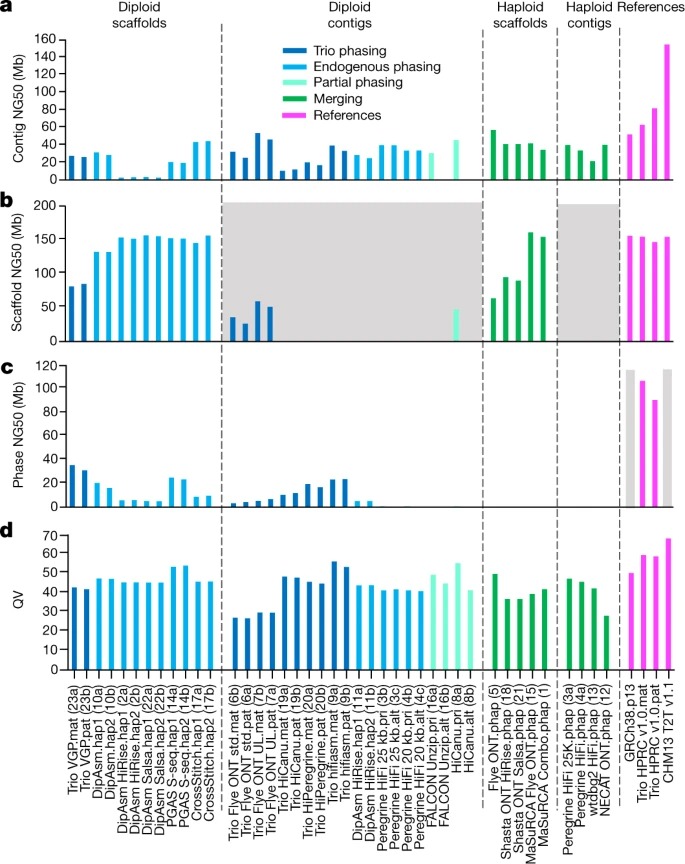
여기서, 우리의 첫 번째 과학 보고서에서, 우리는 최소한의 수동 큐레이션으로 가장 완전하고 정확한 diploid genome assembly를 산출하는 현재의 게놈 시퀀싱과 assembly approach의 조합을 결정했다. assembly 중에 graph-based haplotype 단계별로 매우 정확한 긴 읽기 및 부모-자녀 데이터를 사용한 접근 방식은 그렇지 않은 접근 방식보다 성능이 우수했다. 최고 성능의 방법의 조합을 개발하면서, 우리는 염색체당 평균 약 4개의 간격만 포함하는 첫 번째 고품질 diploid reference assembly를 생성했고, 대부분의 염색체는 CHM13 길이의 ±1% 내에 있었다. 단백질 코딩 유전자의 거의 48%는 haplotype간에 non-synonymous amino acid change를 가지며 centromeric region 이 가장 높은 다양성을 나타낸다.
우리의 발견은 단일 뉴클레오타이드에서 구조 재배열까지의 전체 유전자 변이를 포착하기 위해 pangenome reference를 위해 거의 완전한 diploid human genome을 규모에 맞게 조립하는 기초 역할을 한다.