Cephalopod의 RNA recoding에 의한 microtubule 모터 단백질 기능 조절
Abstract
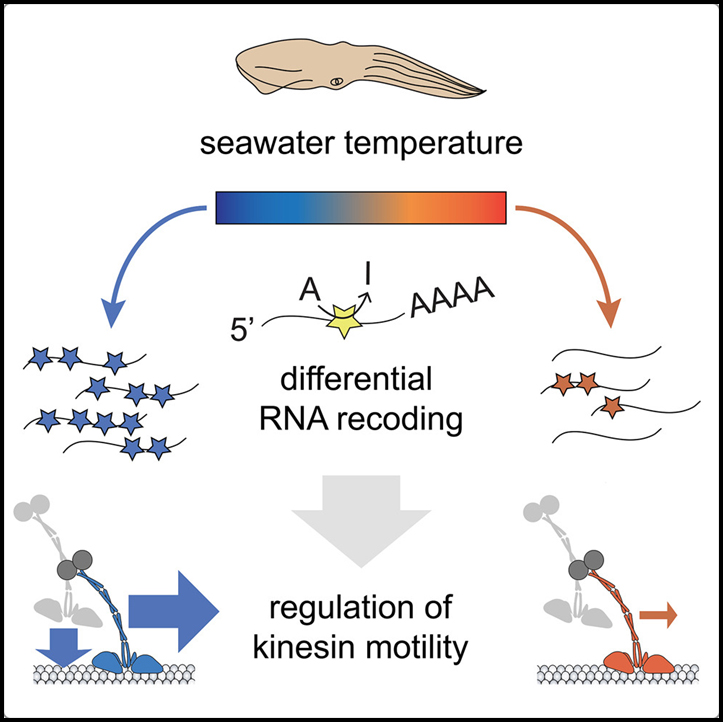
RNA 편집은 “recoding”라고 하는 단백질의 아미노산 서열을 변경할 수 있는 광범위한 후생유전학적 과정입니다. cephalopod에서 대부분의 전사체는 recoding되며, recoding은 표현형 가소성을 생성하기 위한 적응 전략인 것으로 추측됩니다. 그러나 동물이 RNA recoding을 동적으로 사용하는 방법은 대부분 연구되지 않았습니다.
이 연구에서는 microtubule 모터 단백질 kinesin과 dynein에서 cephalopod RNA recoding의 기능을 조사했습니다. 우리는 오징어가 해수 온도 변화에 반응하여 RNA recoding을 빠르게 사용하고 차가운 바닷물에서 생성된 kinesin 변이체가 추위에서 수행된 단일 분자 실험에서 향상된 운동 특성을 나타냄을 발견했습니다. 우리는 또한 독특한 운동 특성을 나타내는 tissue-specific recoded 오징어 kinesin 변이체를 확인했습니다. 마지막으로, 우리는 cephalopod 기록 사이트가 non-cephalopod kinesin과 dynein의 기능적 치환 발견을 안내할 수 있음을 보여주었습니다. 따라서 RNA recoding은 cephalopod에서 표현형 가소성을 생성하고 보존된 non-cephalopod 단백질의 특성화를 알릴 수 있는 동적 메커니즘입니다.
Figure
[Figure 1] Cephalopod recoding 기능 연구 모델로써의 microtubule 운동 단백질
(A) 전체 D. pealeii 전사체와 비교하여 D. pealeii 편집된 전사체 사이에서 상당히 풍부한 상위 20개의 GO 용어.
(B와 C) Liscovitch-Brauer 등이 보고한 D. pealeii, S. officinalis, O. bimaculoides와 O. vulgaris에서 recoding 사이트의 합계. (B) dynein 수송 복합 단백질, (C) kinesin 수송 복합 단백질.
[Figure 2] 오징어 kinesin의 조직별 기록의 기능적으로 구별되는 모터 생성
(A) 오징어 D. pealeii kinesin-1 모터 도메인 (아미노산 1-554)을 따라 recoding 된 아미노산을 나타내는 개략도.
(B) 시신경엽과 성상 신경절이 있는 D. pealeii의 그림.
(C와 D) 시신경엽 (녹색)와 성상 신경절 (주황색)에서 D. pealeii kinesin cDNA의 클론 시퀀싱 요약.
(E와 F) 시신경엽 (E)와 성상 신경절 (F)로부터의 D. pealeii kinesin 모터 도메인을 따라 기록하는 부위 사이의 쌍별 상관관계.
(G) 인간 kinesin (회색)와 편집되지 않은 D. pealeii kinesin (갈색)의 속도 분석.
(H) 인간 kinesin (회색)와 편집되지 않은 D. pealeii kinesin (갈색)의 실행 거리 분석.
(I) D. pealeii kinesin에서 생성된 D. pealeii 기록 부위 변이체.
(J) 편집되지 않은 D. pealeii kinesin와 변형 OL-1, SG-1, SG-2의 대표적인 kymograph.
(K) 편집되지 않은 D. pealeii kinesin (회색)과 비교한 OL-1 (녹색)의 속도 분석.
(L) 편집되지 않은 D. pealeii kinesin (회색)과 비교한 OL-1 (녹색)의 실행 거리 분석.
(M) 편집되지 않은 D. pealeii kinesin과 비교한 SG-1 (주황색)와 SG-2 (갈색)의 속도 분석.
(N) 편집되지 않은 D. pealeii kinesin과 비교하여 SG-1 (주황색)와 SG-2 (갈색)의 실행 거리 분석.
[Figure 3] 해수 온도에 따른 오징어 kinesin 전사체의 차등적 기록
(A) D. opalescens 해츨링의 사진.
(B) 온도 분석의 개략도.
(C) 서로 다른 온도에 노출된 개별 동물의 D. opalescens kinesin-1 모터 도메인을 따라 recoding 사이트에서 편집 비율.
(D와 E) 6°C (파란색) 또는 20°C (빨간색) 물에 노출된 동물의 D. opalescens kinesin cDNA의 클론 시퀀싱 요약.
(F) 6°C (파란색) 또는 20°C (빨간색) 물에 노출되거나 둘 다 (보라색) 물에 노출된 동물의 D. opalescens kinesin에서 고유한 기록 부위 조합.
(G와 H) 6°C (G) 또는 20°C (H) 물에 노출된 동물의 D. opalescens kinesin 모터 도메인을 따라 기록 부위 간의 쌍별 상관관계.
[Figure 4] 추위에 특화된 오징어 kinesin 변이체의 운동성 향상
(A) 25°C (빨간색) 대 8°C (파란색)에서 편집되지 않은 D. opalescens kinesin의 속도 분석.
(B) 8°C (파란색)와 25°C (빨간색)에서 편집되지 않은 D. opalescens kinesin의 실행 거리 분석.
(C) D. opalescens는 D. opalescens kinesin: Cold-1, Cold-2와 Cold-3에서 생성된 사이트 변형 존재.
(D) 편집되지 않은 D. opalescens kinesin와 Cold-1, Cold-2와 Cold-3 변이체의 대표적인 kymograph.
(E) 편집되지 않은 D. opalescens kinesin과 비교한 Cold-1, Cold-2와 Cold-3의 속도.
(F) 편집되지 않은 D. opalescens kinesin과 비교하여 Cold-1, Cold-2와 Cold-3의 실행 거리.
(G) 편집되지 않은 D. opalescens kinesin과 비교하여 Cold-1, Cold-2와 Cold-3의 방문율.
(H) 인간 kinesin 치환 K67R, Y77C와 N117D의 대표적인 kymograph.
(I) 야생형 인간 kinesin과 비교한 K67R, Y77C와 N117D의 속도.
(J) 야생형 인간 kinesin과 비교한 K67R, Y77C와 N117D의 주행 거리 분석.
(K) 야생형 인간 kinesin과 비교한 K67R, Y77C와 N117D의 착지율.
[Figure 5] Cephalopod recoding 부위는 dynein과 kinesin의 보존된 잔기에서 발생
(A와 B) kinesin-1 중쇄 (A)와 세포질 dynein-1 중쇄-1 (B)에 걸쳐 분포된 cephalopod 기록 부위.
(C와 D) kinesin (C)와 dynein (D) 운동성에 중요한 기계화학적 요소와 관련된 선택된 cephalopod 기록 부위를 보여주는 개략도.
[Figure 6] 인간 kinesin과 효모 dynein의 운동성을 조정하는 부위 대체 recoding
(A) 각 인간 kinesin 돌연변이체에 대한 분획 진행.
(B) 각 인간 kinesin 돌연변이체의 속도.
(C) 각 인간 kinesin 돌연변이체의 주행 거리.
(D) 각 효모 dynein 돌연변이에 대한 분획 진행.
(E) 각 효모 dynein 돌연변이의 속도.
(F) 각 효모 dynein 돌연변이체의 주행 거리.
Disscussion
우리의 결과는 cephalopod가 표현형 가소성을 생성하는 메커니즘으로 RNA recoding를 사용한다는 것을 뒷받침합니다. 우리는 오징어가 생리적 요구와 변화하는 환경 조건에 대한 순응을 지원할 수 있는 다양한 조직과 환경 온도에서 RNA recoding를 통해 kinesin 기능을 유연하게 조정할 수 있음을 보여줍니다. 이러한 데이터는 RNA recoding이 온도 적응에 관여할 수 있다는 오랜 가설을 뒷받침하는 증거를 제공합니다. 유기체는 환경 온도 변화를 보상하기 위해 다양한 분자 메커니즘을 사용하고 kinesin 또는 기타 단백질의 온도 의존적 recoding은 Drosophila와 동면하는 포유동물과 같은 다른 동물에서도 온도 순응의 중요한 구성 요소일 수 있습니다.
오징어 kinesin 전사체에 대한 우리의 분석은 조합 RNA 편집을 조절하는 메커니즘을 탐구하는 미래 작업에 동기를 부여합니다. 편집 사이트는 일반적으로 short-read RNA 시퀀싱에서 결정되므로 대부분의 전사에서 동일한 전사에서 편집 사이트가 어떻게 조합되어 발생하는지 알 수 없습니다. 인간에서 세로토닌 2C 수용체의 조합 편집은 기능적으로 구별되는 수용체 변이체를 생성할 수 있으며 조절 장애 편집은 신경 장애와 관련이 있습니다. 우리는 고유한 운동 특성을 가진 변이체를 인코딩하는 오징어 kinesin에서 조직별와 온도별 기록 사이트 조합을 확인했습니다. 이전 연구와 유사하게 편집 사이트 간의 중요한 상관관계를 감지했습니다. 우리의 작업은 또한 사이트 간 recoding의 상관관계가 조합 편집의 조건별 규정을 암시하는 조건일 수 있음을 시사합니다. 이러한 조절은 ADAR 발현 또는 국소화, 차등 RNA 국소화 또는 접힘 수준에서 또는 다른 RNA 결합 단백질과의 상호작용을 통해 매개될 수 있습니다. 실제로 Drosophila에서 Shaker 칼륨 채널의 조합 편집에서 조직 특이적인 차이는 ADAR 발현 수준만의 차이를 넘어서는 조절을 포함하는 것으로 보입니다. 오징어 kinesin의 recoding의 경우, 어떤 ADAR이 어떤 사이트에서 편집을 담당하는지, 또는 recoding이 발생할 수 있는 위치 (핵 vs 세포질)는 알 수 없습니다. 예를 들어, 포유류 ADAR2와 달리 오징어 ADAR2는 핵과 세포질 모두에 국한되어 잠재적인 조절의 추가 모드를 여는 것으로 나타났습니다. 새로운 long-read RNA 시퀀싱 방법은 미래에 다양한 세포 유형, 세포 내 구획과 조건에 걸쳐 조합 RNA 편집을 광범위하게 평가할 수 있는 기회를 제공해야 합니다.
인간 kinesin와 효모 dynein의 코딩 부위 치환에 대한 우리의 조사는 보존된 단백질의 기능을 조사하기 위해 cephalopod 편집체가 어떻게 사용될 수 있는지를 강조합니다. 다중 단백질 서열 정렬은 보존된 기능 영역을 추론하기 위한 비교 유전적 접근법을 제공합니다. cephalopod RNA 편집은 보존된 잔기에서 기능적으로 중요한 치환을 추가로 정확히 찾아낼 수 있는 후생유전학적 렌즈를 제공하여 상동성만으로 얻은 통찰력을 보완합니다. 이 접근법은 기능에 중요한 아미노산 잔기의 선험적 식별이 어려운 단백질 영역에서 특히 유용할 수 있으며 다른 상황에서 단백질 기능을 향상시키거나 맞춤화하는 아미노산 치환을 식별하는 데 사용될 수 있습니다. 따라서, 우리는 cephalopod recoding 부위가 다른 단백질을 특성화하기 위한 일반적인 자원과 가이드를 나타낸다고 제안합니다. 예를 들어, 수많은 질병 관련 단백질은 cephalopod (예: Tau, Amyloid beta, LRRK2)에서 고도로 기록되며 기록 위치를 특성화하면 이러한 단백질의 조절과 기능에 대한 새로운 시각을 제공할 수 있습니다.