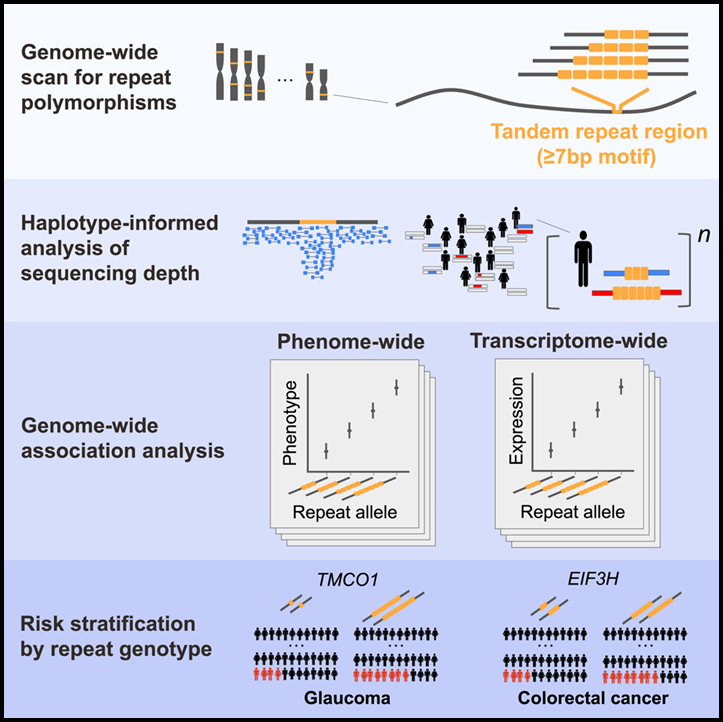
녹내장과 대장암에 대한 가장 높은 유전적 위험을 가진 loci의 기초가 되는 repeat polymorphism
Abstract
인간 유전체의 많은 영역은 variable numbers of tandem repeat (VNTR)으로 인해 개인마다 길이가 다릅니다. VNTR 유전체 전체의 표현형 영향을 평가하기 위해 통계적 대조 접근법을 적용하여 서로 관련 없는 영국 Biobank 참가자 418,136명과 GTEx 참가자 838명에서 9,561개의 상염색체 VNTR loci의 길이를 추정했습니다.
연관 및 통계적 fine-mapping 분석을 통해 UK Biobank의 복잡한 특성에 영향을 미치는 것으로 보이는 58개의 VNTR이 확인되었으며, 그 중 18개는 인근 유전자의 발현 또는 접합을 조절하는 것으로 나타났습니다. TMCO1 및 EIF3H의 non-coding VNTR은 각각 녹내장과 대장암 발병 위험에 대한 일반적인 인간 유전적 변이에 가장 큰 기여를 하는 것으로 나타났습니다. 또한, 이 두 VNTR은 각각 개인 전체에 걸쳐 2배 이상의 위험 범위와 연관되어 있습니다.
이러한 결과는 인간 건강 및 유전자 조절에서 non-coding VNTR의 실질적이고 과거에 인정받지 못했던 역할을 보여줍니다.
Figures
Ascertainment and genotyping of 15,653 VNTR polymorphisms genome-wide
– 이 연구에서는 HGSVC2 (Human Genome Structural Variation Consortium)의 long-read sequencing에서 생성된 64개의 haploid genome assembly와 함께 GRCh38 reference genome을 분석하여 인간 유전체 전체에서 VNTR loci를 식별했습니다.
[Figure 1] 15,653개의 다중 대립유전자 VNTR loci의 확인, 유전형 분석 및 대조
(A) HGSVC2의 n=64개 long-read haploid genome assemblies 중에서 관찰된 뚜렷한 대립유전자의 수와 대립유전자당 반복 횟수의 중앙값으로 계층화된 VNTR loci의 수.
(B) HGSVC2 대립유전자 length distribution width (SD)에 의해 계층화된 VNTR loci의 수와 VNTR 유전자형 사전 정제의 정확도 추정.
(C) 각 VNTR에 대해 best tag SNP를 사용한 대조 정확도 vs 연관 불균형 수준의 분산.
[Fig. 1A] VNTR의 중간 반복 단위 길이는 34bp이고 HGSVC2 assembly 간에 표시되는 6개의 개별 대립유전자의 중앙값을 가졌습니다. 또한, 더 많은 반복을 가진 VNTR은 일반적으로 대립유전자 다양성이 컸습니다.
[Fig. 1B] HGSVC2 assembly의 VNTR 대립유전자 길이 분포는 중앙값 범위가 199bp이고 중앙값 표준 편차가 46.8bp였습니다. 이러한 초기 VNTR 길이 유전자형은 highly length-polymorphic인 VNTR에 대해 대립형질 변이를 정확하게 포착했지만 less-variable length인 VNTR의 경우 정확도가 떨어졌습니다.
[Fig. 1C] 대부분의 다중 대립유전자 VNTR의 경우, haplotype-sharing 분석과 sequencing read-depth 분석의 조합은 근처의 이중 대립유전자 SNP보다 더 강하게 실제 대립유전자 길이 변화와 상관관계가 있는 강력한 통계적 대조를 가능하게 했습니다.
Exploring the effects of non-coding VNTRs phenome-wide
– VNTR이 보다 광범위하게 복잡한 특성을 형성하게 만드는지 평가하기 위해 UKB에서 측정된 668개의 추가 표현형으로 분석을 확장하여 각 표현형과 9,561개의 귀속된 VNTR과의 연관성을 테스트했습니다.
[Figure 2] 현상 전반에 걸친 연관과 통계적 fine-mapping 분석을 통해 복잡한 특성과 연결된 58개의 VNTR 식별
(A) Bonferroni 유의성 (p<5×10^-9)에 도달하고 FINEMAP (PIP>0.5)에 의해 VNTR에 높은 사후 인과 확률이 할당된 107개의 VNTR 표현형 연관성을 표시하는 Manhattan plot.
(B) 연관 및 통계적 fine-mapping 상태별로 그룹화된 VNTR에 대한 GeneHancer promoter와 enhancer (왼쪽), GENCODE(v26) exon (중간), GENCODE(v26) transcript (오른쪽)와 겹치는 VNTR의 빈도.
[Fig. 2A] 분석을 통해 4,910개의 중요한 VNTR 표현형 연관성이 확인되었으며, 그 중 107개의 연관성에는 통계적 fine-mapping에 의해 높은 인과 확률을 확인했습니다.
[Fig. 2B] 이러한 연관성에 관련된 많은 VNTR은 promoter, enhancer, exon 등과 같은 조절 요소나 코딩 요소와 겹쳤습니다.
> 이러한 연관성에는 이전에 보고된 인슐린 유전자 INS와 제1형 당뇨병의 VNTR upstream 연관성을 비롯하여 녹내장, 대장 용종 및 고혈압 위험이 있는 VNTR 길이 다형성 연관성을 포함한 비코딩 VNTR 다형성과 인간 질병 사이의 5가지가 포함되었습니다.
Repeat expansion at TMCO1 associates with glaucoma risk more strongly than any SNP or indel in the genome
–
[Figure 3] 녹내장 위험 및 안압과 연관된 TMCO1의 intronic repeat expansion
(A) 1000 Genomes Project에 나타난 각 대륙 인구의 1, 2, 및 ≥5 반복 단위 대립유전자의 빈도.
(B-C) 녹내장 (B) 및 안압 (C)과 SNP, VNTR의 연관성.
(D-E) 녹내장 위험에 대한 VNTR 대립유전자의 효과 크기 (D) 및 각 대립유전자 운반체의 평균 안압 (E).
[Fig. 3A-3B] 녹내장 위험과 관련된 이 VNTR은 전체 유전체의 모든 SNP 또는 indel보다 더 강력합니다. 이는 녹내장에 대한 일반적인 유전적 변이의 처음으로 확인되고 가장 강력하게 알려진 영향 중 하나입니다.
[Fig. 3C] 녹내장 사례를 제외한 후에도 TMCO1 VNTR 길이는 유전체 내의 어떤 SNP보다 더 강력하게 IOP와 연관되어 있어 인근 SNP가 아닌 VNTR이 TMCO1 VNTR은 IOP에 대한 영향을 통해 녹내장 위험에 영향을 미칩니다.
[Fig. 3D-3E] TMCO1 VNTR의 반복 대립유전자는 더 긴 반복 길이에서 IOP 및 녹내장 위험에 대한 영향이 증가하는 대립유전자 시리즈를 형성했습니다. 확장된 VNTR 대립유전자에 태그를 붙인 TMCO1의 SNP는 독립적이고 성능이 뛰어난 녹내장 및 IOP 유전 연관성 데이터 세트에서 이러한 연관성을 복제할 수 있는 기회를 제공했습니다.
Common repeat polymorphism at EIF3H associates with a 2-fold range of colorectal cancer risk
-대장암은 100개 이상의 공통 위험 대립유전자가 확인된 유전성 복합 질환으로, 각각은 질병 위험에 미묘한 영향을 미칩니다. 이를 확인하고자 EIF3H 연관 유전자들을 측정했습니다.
[Figure 4] 대장암 위험 및 결장 폴립과 연관된 EIF3H downstream의 repeat expansion
(A-B) EIF3H locus (상단)와 유전체 전체 (하단)에서 유전 변이체와 대장암 (A) 및 결장 폴립 (B)의 연관성.
(C) 유럽계 UKB 참가자에서 관찰된 VNTR 대립유전자의 빈도와 대장암 및 결장 폴립에 대한 효과.
[Fig. 4A-4B] 대조적으로, 27bp 반복 길이 EIF3H downstream에서 ~20kb는 대장암과 대장 폴립의 위험에 강력하게 연관되어 있습니다. VNTR은 대장암에 대해 보고된 최초의 연관성 중 하나인 인근 SNP 연관성을 설명하는 것으로 나타났습니다. 이 loci는 유전체 전반에 걸쳐 모든 대장암 관련 loci 중 1위를 차지했습니다.
[Fig. 4C] 공통 대립유전자는 유전체의 일반적인 SNP 또는 indel보다 더 높은 대장암 위험을 나타냅니다. UKB에서 VNTR의 연관성은 대장암 및 대장 폴립의 위험에 증가하는 영향을 나타내는 일련의 4가지 공통 대립유전자에 의해 주도되었습니다. 질병 위험은 VNTR 길이에 따라 선형적으로 증가했으며, 각 추가 반복 단위는 대장암 위험이 14% 증가했습니다.
> 이러한 결과는 이 loci의 위험 대립유전자는 EIF3H 발현과 연관되어 있는 것으로 나타나지 않았습니다. VNTR을 이 loci의 원인 변이에 대한 유망한 후보로 확인했지만 대장 폴립과 분자 메커니즘을 해독하려면 새로운 종류의 데이터가 필요합니다.
Intronic repeat expansion in CUL4A influences alternative splicing and erythrocyte traits
-GTEx RNA sequencing 데이터 분석에 따르면 VNTR 대립유전자 길이는 CUL4A의 명백한 스플라이스 결함과 밀접한 관련이 있는 것으로 나타났습니다. 대신 훨씬 더 가까운 서열로 스플라이싱하여 15개의 downstream 표준 exon 없이 CUL4A 판독 프레임이 조기에 잘리는 것을 확인합니다.
[Figure 5] 적혈구 특성 및 splice isoform 사용과 연관된 CUL4A의 intronic repeat expansion
(A) 일반적으로 발현되는 두 가지 CUL4A isoform의 대체 스플라이싱.
(B) CUL4A locus에서 평균 미립자 hemoglobin과 VNTR, SNP의 연관성.
(C) 유럽계 UKB 참가자의 VNTR 대립유전자 길이 분포 및 강하게 연관된 4개의 혈액 세포 특성에 대한 VNTR 대립유전자 보유자의 평균 표현형.
(D) 배양된 fibroblast에서 CUL4A 대체 스플라이싱 사용량과 VNTR, SNP의 연관성.
(E) GTEx의 VNTR 대립유전자 분포 및 VNTR 연관이 가장 강한 5개 조직에 대한 VNTR 대립유전자 운반체의 평균 대체 스플라이싱 사용량.
(F) GTEx로 분석한 49개 조직 각각에서 VNTR 결합 강도 vs 대체 스플라이싱을 사용한 가장 강한 SNP 결합 강도의 산점도.
(G) 49개 조직 각각에 대한 중앙 대체 스플라이싱 사용량 vs 중앙 CUL4A 발현의 분산.
[Fig. 5A-5C] CUL4A에서 평균 미립자 hemoglobin 감소와 관련된 고도로 polymorphism intron 반복의 확장 및 9개의 기타 적혈구 관련 특성을 보입니다. VNTR 연관성은 인근 SNP보다 3배 이상 더 강했고 관련 표현형에 단조롭게 강화되는 효과를 갖는 일련의 대립유전자에 의해 주도되었습니다.
[Fig. 5E-5F] 이 스플라이싱은 변형이 Bonferroni 중요성에 도달한 30개 조직 각각에서 어떤 SNP보다 훨씬 더 강력하게 VNTR과 연관되어 있다는 것을 보입니다. 각각의 경우에, 긴 대립유전자는 단백질 절단 isoform의 더 큰 사용과 연관됩니다.
[Fig. 5G] VNTR의 영향을 넘어서, 이러한 방식으로 잘못 접합된 CUL4A 전사체의 비율은 조직에 따라 상당히 다양합니다. 또한, VNTR 길이뿐만 아니라 컨텍스트도 접합 결과에 영향을 미칩니다.
> 이러한 결과는 CUL4A 유전자가 생쥐의 조혈에 필요한 orthologue (Cul4A)가 있는 ubiquitin ligase를 암호화하며, 이는 VNTR 길이 polymorphism이 CUL4A 스플라이싱을 방해하여 적혈구 특성에 영향을 미칠 수 있는 분자 메커니즘을 암시하고 그에 따라 절단된 CUL4A isoform의 생성을 조절합니다. 이러한 방식으로 잘못 접합된 CUL4A 전사체의 비율은 조직에 따라 상당히 다양합니다. VNTR 길이뿐만 아니라 컨텍스트도 접합 결과에 영향을 미칩니다.
Exploring VNTR effects on gene expression and splicing
-유전자 조절에서 반복 polymorphism의 역할을 체계적으로 탐구하고 복잡한 특성과 VNTR 연관성의 기본이 되는 잠재적인 분자 메커니즘을 식별하기 위해 9,561개의 귀속된 VNTR을 테스트하여 인근 유전자에 대한 발현 및 스플라이싱 정량적 특성과의 연관성을 테스트했습니다.
[Figure 6] 유전자 조절과 관련된 VNTR은 복잡한 특성에 대한 연관을 중재하는 유전자와 관련되며 해당 유전체 요소 근처에 풍부
(A) eVNTR과 관련된 유전자의 전사 시작 위치 사이의 거리 분포.
(B) sVNTR과 영향을 받은 스플라이싱 위치 사이의 거리 분포.
(C) GeneHancer 주석이 달린 promoter 또는 enhancer와의 중복 빈도.
(D) 스플라이싱 또는 발현 정량적 특성과의 fine-mapping 지원 연관에 관여하는 VNTR의 비율.
[Fig. 6A] cis 조절과 관련된 VNTR은 관련 유전체 특징 근처에서 농축을 나타냈습니다. 발현 관련 VNTR (eVNTR)은 이전 eVNTR 분석과 일치하여 전사 시작 사이트 근처에서 농축되었습니다.
[Fig. 6B] 스플라이스 관련 VNTR (sVNTR)은 영향을 받은 근처에서 농축되었습니다.
[Fig. 6C] eVNTR과 sVNTR 모두 주석이 달린 조절 영역이 풍부했습니다
[Fig. 6D] 이러한 데이터를 VNTR 연관성에 대한 현상 전반의 스캔과 통합하면 UKB의 유전자 조절과 복잡한 특성 모두에 영향을 미치는 것으로 보이는 18개의 VNTR이 확인되었습니다. 이 분석은 절제와도 관련된 PLEC의 인트론 VNTR을 포함하여 UKB의 fine-mapping 지원 연관과 관련된 VNTR의 31%에 대한 조절 기능을 제공했습니다.
Hundreds of repeat polymorphisms influence splice site usage by diverse mechanisms
-각각 조직 전반에 걸쳐 연관성과 인과성에 대한 일관된 증거를 나타내는 22개의 sVNTR 세트가 포함되었습니다. 이들 중 21개는 영향을 받은 스플라이스 부위의 1kb 내에 위치했고, 20개는 스패닝 인트론의 변경된 스플라이싱과 관련이 있었습니다. 영향을 받은 스플라이스 사이트와 관련된 이러한 sVNTR의 위치 및 명백한 전사 효과에 대한 추가 조사를 통해 반복 polymorphism이 스플라이싱에 영향을 미칠 수 있는 다양한 방식에 대한 통찰력이 밝혀졌습니다.
[Figure 7] 다양한 메커니즘에 의한 반복 polymorphism 접합 영향
UPF3A (A), NOC4L (B), PLIN5 (C), PLQC1 (D)의 VNTR이 여러 조직에 걸쳐 스플라이싱을 조절함을 보이는 증거.
왼쪽: 각 loci에서 짧거나 (상단) 긴 (하단) VNTR 유전자형을 가진 개인의 GTEx 샘플에 대한 RNA sequencing depth와 LeafCutter intron 절제 수를 표시하는 Sashimi plot.
오른쪽: 왼쪽 패널에서 별표가 표시된 절단 횟수가 있는 intron에서 파생된 스플라이싱 정량적 특성에 대한 VNTR vs SNP 연관 통계의 산포도.
[Fig. 7A] 반복 대립유전자에는 UPF3A와 TUBGCP2의 대체 스플라이스 사이트가 포함되어 있습니다. UPF3A에서 32bp 인트론 반복은 두 가지 유형의 대체 전사체 사용에 영향을 미치는 것으로 나타났습니다.
[Fig. 7B] 정규 스플라이스 기증자 사이트를 포함하는 반복은 NOC4L 및 RSPH1의 다중 exon 건너뛰기와 연관됩니다. NOC4L의 44bp 반복과 RSPH1의 41bp 반복의 긴 대립유전자는 표준 스플라이싱을 방해하고 ≥5 건너뛴 exon이 부족한 스플라이스 형태의 빈도를 증가시키는 것으로 나타났습니다.
[Fig. 7C] 표준 스플라이스 수용체에 가까운 반복은 PLIN5의 인트론 유지와 연관됩니다. 이 24bp 반복의 3′ 끝은 표준 스플라이스 수용체에서 5bp 떨어진 곳에 위치합니다.
[Fig. 7D] 흥미롭게도 PQLC1에서 VNTR은 대안적으로 발현된 exon 자체 내에 포함되어 있습니다.
> 이러한 결과 각 loci에서 VNTR와 가깝거나 그 downstream intron 전사는 대립유전자 길이에 따라 증가하며 명백한 대체 스플라이스 수용체가 없다는 것을 보입니다.
Disscussion
이 연구 결과는 일반적인 질병의 위험과 관련된 5개의 VNTR 길이 다형성을 포함하여 인간 표현형과 유전자 발현에 강력한 영향을 미치는 것으로 보이는 많은 VNTR을 식별합니다. 우리가 관찰한 TMCO1, EIF3H의 VNTR과 관련된 두 가지 질병 연관성은 각각 녹내장과 대장암 위험에 대한 공통 유전 변이의 가장 강력하게 알려진 유전적 영향인 것으로 나타났습니다. 또한 GTEx의 분석에 따르면 VNTR은 다양한 메커니즘 세트를 통해 영향을 받은 스플라이싱 사이트의 근위와 원위 위치에서 스플라이싱 isoform 사용을 조절할 수 있는 것으로 나타났습니다. 이 발견은 다른 유전자 데이터 세트에도 유사하게 적용할 수 있는 프레임워크인 SNP 배열 유전형 분석 데이터에 대한 통계적 위상 조정 및 대체와 sequencing depth-of-coverage 분석을 통합한 VNTR 유전자형 추정에 대한 전산적 접근 방식을 통해 가능해졌습니다.
GTEx에 대한 분석을 통해 UKB의 복잡한 특성과 연결된 VNTR의 상당한 부분 (18/58)의 기본이 되는 그럴듯한 전사 메커니즘과 인과 유전자가 확인되었지만 대부분의 표현형 관련 VNTR은 유전체의 non-coding 영역에 위치하여 DNA 서열 길이를 조절합니다. 수백에서 수천 개의 염기쌍이 존재하지만 표현형에 대한 명백한 영향을 설명할 수 있는 명확한 분자 메커니즘은 없습니다. 표현형과 연결된 non-coding 변이체는 인간 유전학에서 중요한 문제를 제기합니다. “missing regulation”을 밝히고 여기에서 관찰된 연관성의 기본 메커니즘을 식별하려면 다른 기술과 추가 연구가 필요할 것입니다.