Amylase 유전자좌에서 재발생하는 진화와 선택에 의한 구조적 다양성 형성
Abstract
농업의 채택은 인간 집단에서 녹말이 풍부한 식단으로의 급격한 전환을 초래했습니다. Amylase 유전자는 녹말 소화를 돕는데, 녹말 섭취량이 많은 현대 인구 집단에서 amylase 유전자의 복제 수가 증가한 사례가 관찰되었지만, 최근 선택에 대한 명확한 증거는 부족했습니다.
여기서 우리는 94개의 long-read haplotype-resolved assembly와 약 5,600명의 현대 및 고대 인간으로부터의 short-read 데이터를 사용하여 amylase 유전자좌에서 구조적 변이의 다양성과 진화 역사를 해결했습니다. 우리는 농업 집단에서 amylase 유전자의 복제 수가 어업, 수렵, 목축 집단보다 높다는 것을 발견했습니다. 28개의 독특한 amylase 구조적 형태를 확인하고, 거의 동일한 구조가 최근 인류 역사에서 여러 haplotype 배경에서 반복적으로 발생했음을 증명했습니다. AMY1과 AMY2A 유전자는 여러 번의 복제 및 삭제 사건을 겪었으며, 돌연변이율은 단일 염기 다형성 (single-nucleotide polymorphism: SNP) 돌연변이율보다 최대 10,000배 이상 높았던 반면, AMY2B 유전자 복제는 단일 기원을 공유했습니다.
Pangenome 기반 접근 방식을 사용하여 수천 명의 인간에서 구조적 haplotype을 추정하고, 현대 농업 집단에서 복제가 많이 된 haplotype이 더 높은 빈도로 나타남을 확인했습니다. 533개의 고대 인간 유전체를 활용하여, 복제된 haplotype (조상 haplotype보다 더 많은 유전자 복사본을 가진)이 지난 12,000년 동안 서유라시아인들 사이에서 빠르게 빈도를 높였다는 것을 발견했으며, 이는 긍정적 선택을 시사합니다. 우리의 연구는 농업 혁명이 인간 유전체에 미친 잠재적 영향을 강조하며, 인간 적응에서 구조적 변이의 중요성을 보여줍니다.
Figures
Amylase copy number diversity worldwide

[Figure 1] 전 세계 amylase 복제 수 다양성
(A-C) 147개 다른 인간 집단에서 AMY1 (a), AMY2A (b) 및 AMY2B (c)의 평균 복제 수를 나타내는 세계 지도.
– 점의 크기: 집단 샘플 크기 (1~134)
– 색상: 평균 복제 수
(D) 농업에 전통적으로 의존하는 33개의 현대 인간 집단에서 AMY1 (상), AMY2A (중간), AMY2B (하)의 복제 수 분포를 어업, 사냥, 목축 기반 식단과 비교한 결과.
– 괄호 안 숫자: 샘플 크기
Twenty-eight distinct structural haplotypes

[Figure 2] Amylase 구조적 haplotype 다양성의 pangenome 기반 식별
(A) AMY1, AMY2A, AMY2B 복제 수 간의 관계.
(B) 계층적 MAP-graph (상)와 variation graph (하) 아키텍처.
(C) 94개의 haplotype에서 식별된 28개의 고유한 amylase 구조적 haplotype.
(D) Amylase 유전자에 대해 독립적으로 assemble 된 35명의 개인 (70개의 haplotype)에서 read-depth 기반 복제 수와 assembly 기반 복제 수 간의 관계.
Evolution of structural haplotypes
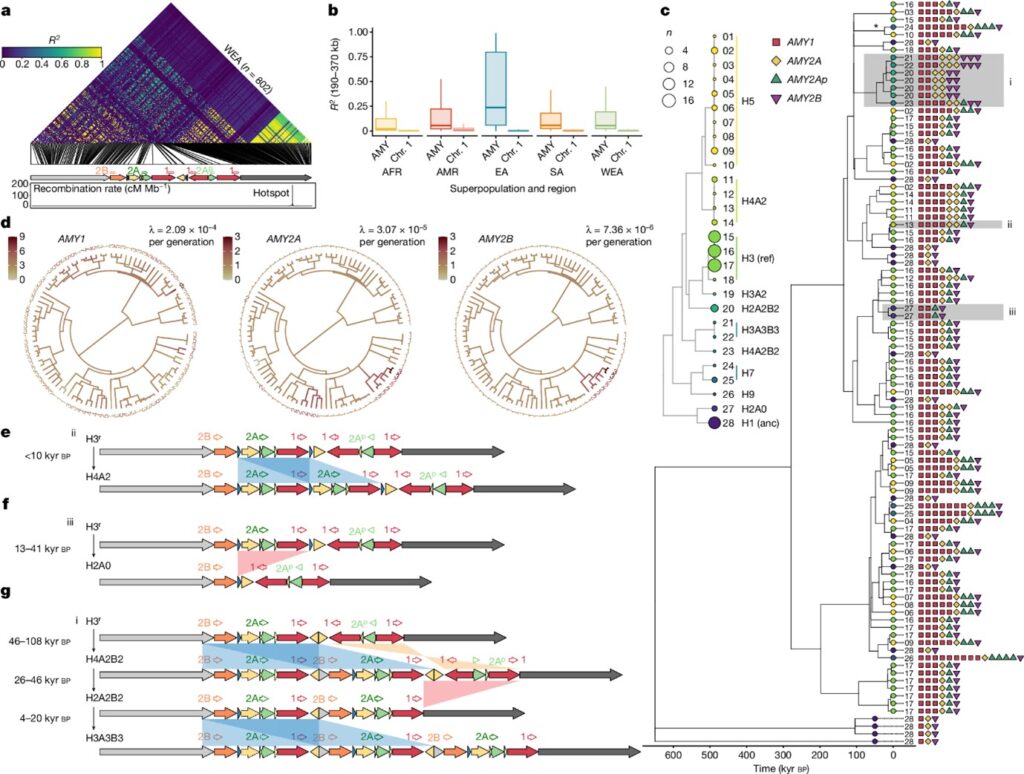
[Figure 3] Amylase 구조적 haplotype의 진화사
(A) 서유라시아인 802명의 amylase 구조적 변이 영역 양쪽의 고유 서열을 포함하는 약 406-kb 구역에 걸친 SNP에 대한 연관 불균형 히트맵 (Heatmap of linkage disequilibrium).
(B) 주요 인간 집단에서 구조적 변이 영역 양쪽의 SNP 쌍과 염색체 1의 동일한 간격으로 떨어진 SNP 간의 연관 불균형을 비교한 boxplot.
– AFR: 아프리카. AMR: 아메리카. CAS: 중앙아시아&시베리아. EA: 동아시아. OCN: 오세아니아. SA: 남아시아.
(C) 94개의 assemble 된 haplotype에 걸친 구조적 변이 영역 (SVR)의 non-duplicated region을 둘러싸는 영역에서 time-calibrated coalescent tree.
(D) Amylase 유전자 복제 수에 대한 조상 상태 재구성과 돌연변이율 추정.
(E-G) 가장 최근의 AMY2A 유전자 복제, AMY2A 유전자의 완전한 소실, 그리고 AMY2A와 AMY2B 유전자의 연속적이고 공동적인 복제의 설명도.
– 파란색: 복제, 빨간색: 삭제, 주황색: 역위.
Pangenome-based haplotype deconvolution

[Figure 4] Short-read 데이터를 통한 복잡한 구조적 haplotype 추론
(A) Short-read로 시퀀싱된 개인에서 존재하는 구조적 haplotype 쌍을 추론하기 위한 haplotype deconvolution 접근 방식의 개략도.
(B) 대륙별 집단에서 구조적 haplotype 빈도(7,188개의 haplotype에서 3,594명의 다양한 인간).
(C) 농업에 전통적으로 의존하는 개인의 haplotype(대립 유전자) 빈도를 어업, 사냥, 목축 기반 식단과 비교한 결과.
[Fig. 4A] 접근 과정
Assemble 된 haplotype 세트가 변이 그래프에 매핑되고, 그래프의 모든 노드에 걸친 커버리지 벡터 정량화 (1)
모든 haplotype 벡터 쌍을 합산하여 합성 유전자형 벡터 생성 (2)
Short-read 게놈이 변이 그래프에 매핑되고, 그래프의 모든 노드에 걸쳐 리드 깊이 정량화 (3)
Short-read 커버리지 벡터를 모든 합성 유전자형 벡터와 비교 (4)
short-read로 시퀀싱된 개인에서 존재하는 가장 가능성 높은 haplotype 쌍을 식별하기 위해 점수 부여 (5)
Recent selection in West Eurasia
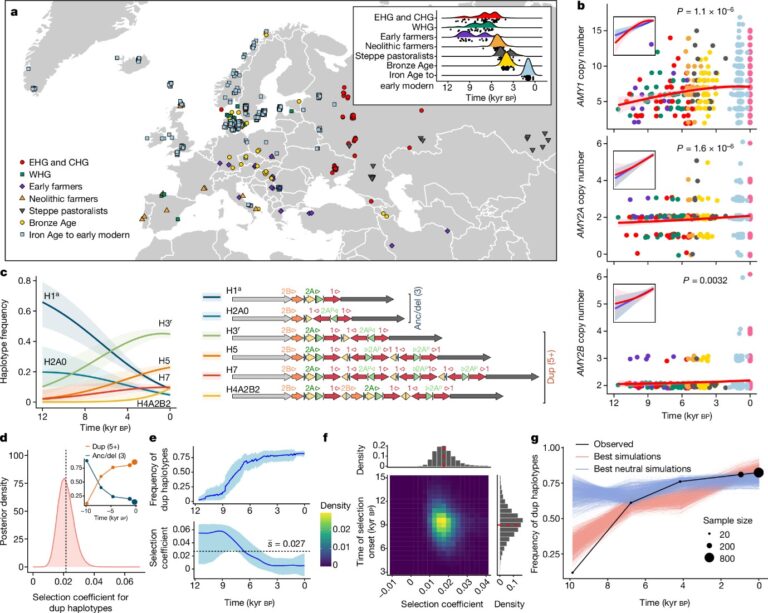
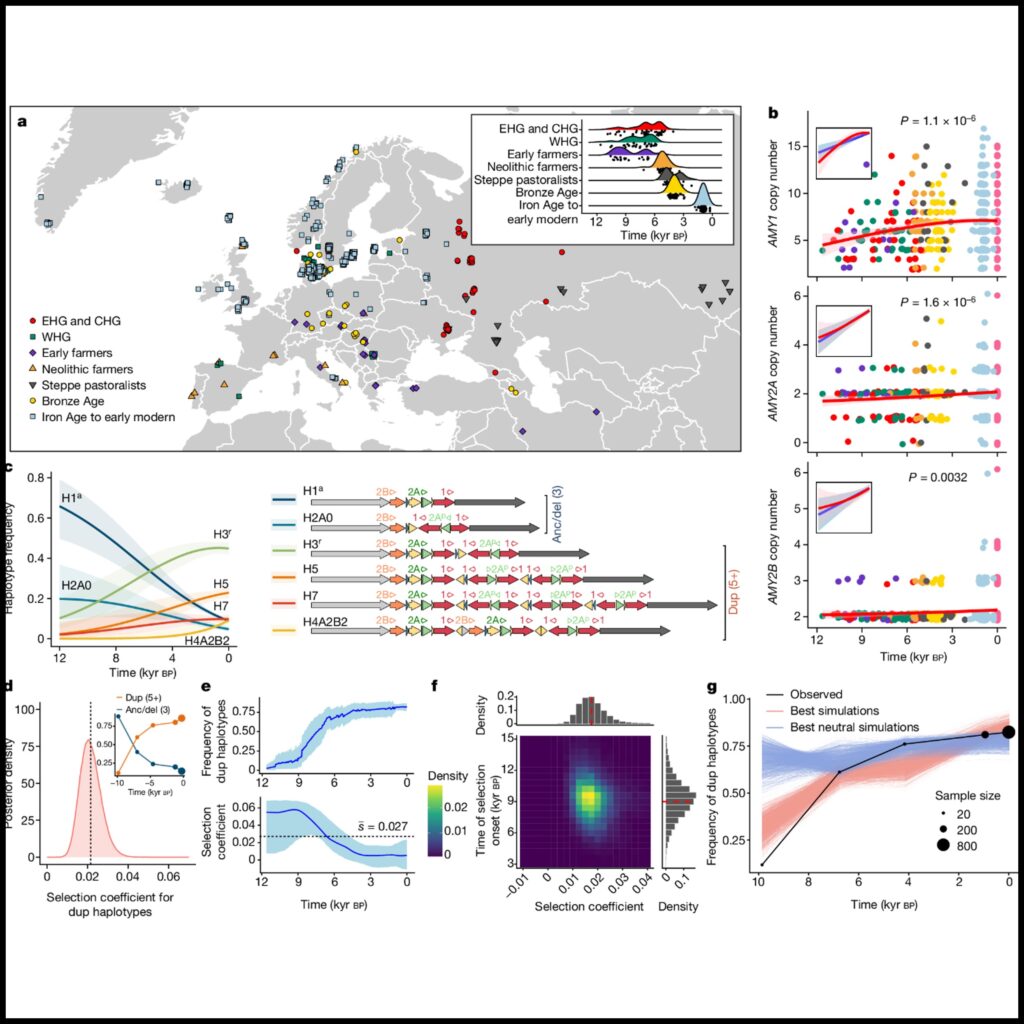
[Figure 5] 서유라시아에서 amylase 유전자군에 대한 최근 선택
(A) Amylase 복제 수가 추정된 533개의 서유라시아 고대 게놈 위치.
– 삽입 그림: 샘플의 추정 연대
– CHG: 코카서스 수렵채집인. EHG: 동부 수렵채집인. WHG: 서부 수렵채집인.
(B) 나이에 따른 복제 수 유전자형을 부드러운 일반화 가법 모형으로 피팅한 그래프.
– 삽입 그림: 데이터에 피팅된 선형 모형 (파란색)과 일반화 가법 모형 (빨간색)
(C) 고대와 현대 서유라시아인에서 1% 이상 빈도로 존재하는 6개의 haplotype (오른쪽)에 대해 다항 로지스틱 회귀로 피팅된 haplotype 궤적.
(D) ApproxWF에서 지난 12,000년 동안 추정된 dup haplotype의 선택 계수에 대한 후방 밀도.
– 삽입 그림: dup 대 anc/del haplotype 빈도 궤적의 구간 관측값
(E) dup haplotype (파란색 선)에 대한 빈도 및 선택 계수 궤적.
(F) ABC 접근 방식을 기반으로 한 선택 계수 및 선택 시작 시기의 후방 분포.
(G) 상위 1,000개의 모든 시뮬레이션 및 상위 1,000개의 중립 시뮬레이션에서 예상된 대립 유전자 빈도 궤적과 관찰된 대립 유전자 빈도 궤적.
Disscussion
작물의 가축화와 그에 따른 농업의 부상은 인간의 사회 구조, 생활 방식, 식단을 근본적으로 재편했습니다. 이 전환의 진화적 흔적은 고대와 현대 서유라시아인의 게놈에서 확인되었습니다. 그러나 amylase 유전자군이 이 전환으로 인해 선택을 받았을 가능성이 제기되어 왔음에도 불구하고, 최근의 긍정적 선택의 흔적은 아직 발견되지 않았습니다. 여기서는 long-read assembly를 활용하여 amylase 유전자군에서 복잡한 haplotype 구조를 현재까지 가장 높은 해상도로 분석하여, short-read 시퀀싱으로는 해결할 수 없는 구조적 및 서열 복잡성을 밝혀냈습니다. 또한, 이 long-read haplotype은 이 복잡한 구조와 연결된 주변 단일 염기 다형성 (SNP)에 대한 새로운 정보를 제공합니다. 이를 통해 최근 인류 역사에서 이 유전자군에서 급속하고 반복적인 중복 및 삭제 사건을 보여주는 공동 진화 나무를 구축할 수 있었습니다. 특히, 이러한 사건의 대부분은 지난 5만 년 동안 발생했으며, 따라서 이러한 사건은 주변 지역의 희귀 변이로만 표시될 것입니다. 따라서 이 지역에서의 광범위한 동형성 및 높은 돌연변이율은 주변 SNP가 고전적인 선택적 스윕 검사의 부적합한 태그가 될 수 있으며, 이는 이 유전자군에서 선택을 감지하려는 이전의 노력이 실패한 이유를 설명할 수 있습니다. 마지막으로, 우리는 long-read assembly를 활용하여 기존의 short-read 데이터를 개선하고, amylase 유전자군의 pangenome 그래프를 구축하여 short-read로 시퀀싱된 개인의 haplotype 구조를 추론했습니다. 이러한 그래프 기반 접근 방식인 haplotype deconvolution을 통해 이전에 short-read로 접근할 수 없었던 영역을 현대 및 고대 데이터 세트에서 다시 탐색할 수 있게 되었습니다.
Haplotype deconvolution 접근 방식을 사용하여 amylase 유전자군에서 288개의 고대 샘플의 haplotype 구조를 자신 있게 재구성할 수 있었습니다. Amylase 유전자의 중복된 사본을 포함하는 haplotype이 지난 12,000년 동안 빈도가 7배 증가한 것을 발견했습니다. 우리의 분석은 상대적으로 적은 샘플 수와 서유라시아에서 haplotype 할당에 적합한 고품질 고대 게놈의 불균형한 샘플링으로 제한된다는 점을 지적해야 합니다. 또한 선택을 테스트하는 여러 접근 방식은 다양한 모델 가정과 유전자형 정확성에 의존합니다. 그럼에도 불구하고 우리는 amylase 유전자군에서 서유라시아인들에게서 최근 선택이 일어났음을 일관되게 지지하는 여러 증거를 제시하며, 이는 농업 채택과 연관될 가능성이 있습니다.
인간의 식단 적응에 대한 가장 잘 연구된 예 중 하나는 젖당 분해 지속성의 진화입니다. 우리가 추정한 선택 계수 (sdup)는 많은 연구에서 보고된 MCM6/LCT 유전자군의 선택 계수 (s) 추정치와 크기가 유사합니다. 그러나 amylase 사본 수의 증가는 충치와 같은 구강 건강 문제와도 연관되어 있어, 이는 LCT와 같은 다른 식단 관련 유전자군과는 다른 선택 역학을 초래할 수 있는 잠재적인 진화적 균형을 나타냅니다. Amylase 유전자군에서 발견된 반복적인 돌연변이와 동형성은 점 돌연변이에 의해 주도된 유전자군과는 달리 더 복잡한 진화적 특성을 추가합니다. 우리는 amylase 유전자의 중복/삭제 돌연변이율이 평균 SNP 돌연변이율의 약 10,000배에 이른다는 것을 발견했으며, 이는 짧은 반복 서열과 유사합니다. 이는 최근에 설명된 Y 염색체 영역에서의 구조적 변이 돌연변이율과 유사합니다. 두 경우 모두 이 유전자군의 중복 구조는 같은 염색체나 자매 염색체의 긴 상동 서열 간 비대립 유전자 상동 재조합 또는 비교차 유전자 변환을 통해 새로운 구조적 변이를 형성할 가능성을 높이며, 이로 인해 유사한 구조적 변이가 발생할 수 있습니다. 따라서 이 유전자군에서는 빠르고 반복적인 구조적 변화가 발생하더라도 연관 불균형이 유지됩니다.
MCM6/LCT 유전자군과 amylase 유전자군 사이의 또 다른 흥미로운 유사점은 우유를 소화하는 능력이 다른 인구에서 독립적으로 발생했다는 점입니다. 마찬가지로 농업도 인류 역사에서 여러 번 독립적으로 채택되었습니다. 여기서 우리는 서유라시아 인구에서 긍정적 선택의 증거를 보여주는 것 외에도, amylase 사본 수가 많은 haplotype이 전 세계적으로 전통적으로 농업에 의존하는 여러 인구에서 더 자주 발견된다는 사실을 발견했습니다. 이러한 결과는 amylase 사본 수 증가에 대한 선택이 인류 역사에서 여러 차례 발생했을 가능성을 시사하며, 이는 농업이 독립적으로 여러 번 채택된 시기와 일치합니다. 유럽 이외의 지역에서 고대 샘플이 부족하기 때문에 다른 농업 채택과 관련된 선택을 추론할 수는 없었습니다. 이러한 가설을 더 검증하려면 다양한 고대 게놈 샘플링과 현대의 long-read assembly가 더 필요합니다. 녹말이 풍부한 식단으로의 전환에 따른 amylase 유전자의 확장은 개, 돼지, 쥐, 생쥐 등 여러 다른 공생 종에서도 독립적으로 발생했을 가능성이 있으며, 이는 이 유전자군의 반복적인 진화와 농업 혁명이 인간뿐만 아니라 다른 종의 유전학과 진화에도 미친 광범위한 영향을 강조합니다.
