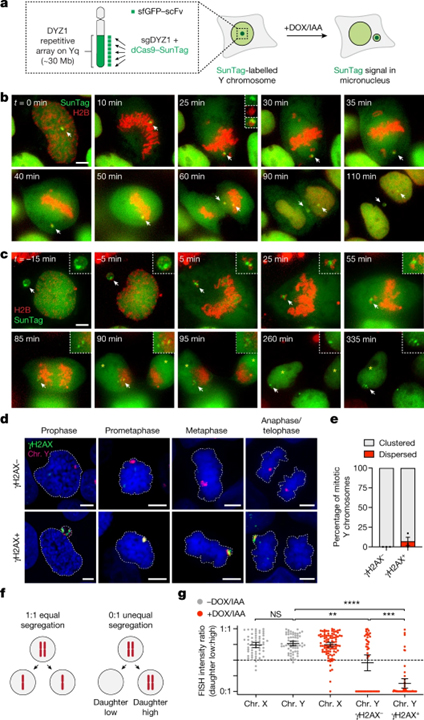
Micronuclei에서 분쇄된 염색체의 유사분열 클러스터링
Abstract
복잡한 게놈 재배열은 chromothripsis로 알려진 과정을 통해 micronuclei 내에 갇힌 잘못 분리된 염색체의 치명적인 분쇄에 의해 생성될 수 있습니다. 각 염색체에는 단일 중심체가 포함되어 있기 때문에 부서진 염색체에서 파생된 비중심성 단편이 유사분열 동안 딸세포 간에 어떻게 유전되는지는 불분명합니다.
이 연구에서는 라이브 셀 이미징 micronucleated 염색체를 추적하고 단일 딸세포에서 유사분열 과정 중 비대칭 전달에 대한 가까운 공간에서 acentric 조각 클러스터를 보여줍니다. 기계적으로, CIP2A-TOPBP1 복합체는 간기 (interphase) 동안 파열된 micronuclei 내 DNA 병변과 조기에 결합하여 유사분열 진입 시 클러스터링을 위해 분쇄된 염색체를 안정화합니다. CIP2A-TOPBP1의 불활성화는 비중심성 단편을 유사분열 세포질 전체에 분산시키고 확률적으로 두 딸세포의 핵으로 분할 시 비정상적인 세포질 DNA 축적이 일어납니다. Mitotic 클러스터링은 정식 chromothripsis의 특징인 광범위한 DNA 사본 수 손실이 없는 재배열된 염색체로 중심이 없는 단편의 재조립을 용이하게 합니다. 범종양 유전체에 대한 종합적인 분석 결과 다양한 종양 유형에 걸쳐 DNA 복사 수 중립적 재배열 클러스터가 밝혀져 알려진 종양 드라이버 이벤트를 획득했습니다. 따라서 chromothripsis의 뚜렷한 패턴은 micronuclei에서 분쇄된 염색체의 공간적 클러스터링으로 설명될 수 있습니다.