Microbial peptides는 glioblastoma에서 tumour-infiltrating lymphocytes를 활성화합니다.
Abstract
Microbial organisms은 인체의 수많은 생리적 과정에서 중요한 역할을 하며 최근에는 immune checkpoint inhibitors에 대한 반응을 조절하는 것으로 밝혀졌습니다. 본 연구에서는 glioblastoma에 대한 immune reactivity에서 microbial organisms의 역할과 잠재적 역할에 대해 다루고자 합니다. 우리는 glioblastoma 조직과 tumour cell lines 모두의 HLA 분자가 bacteria-specific peptides를 존재한다는 것을 입증했습니다. 이 발견은 tumour-infiltrating lymphocytes (TIL)가 tumour-derived bacterial peptides를 인식하는지 여부를 조사하도록 유도했습니다. HLA class II molecules에서 eluted된 Bacterial peptides는 매우 약하게나마 TIL에 의해 인식됩니다. 편향되지 않은 unbiased antigen discovery approach를 사용하여 TIL CD4+ T cell clone의 특이성을 조사한 결과, pathogenic bacteria, commensal gut microbiota 및 glioblastoma-related tumour antigens의 광범위한 peptide를 인식하는 것으로 나타났습니다. 이러한 peptide는 또한 대량 TIL과 peripheral blood memory cells를 강력하게 자극하여 tumour-derived target peptides에 반응합니다. 우리의 데이터는 bacterial pathogens and bacterial gut microbiota가 tumour antigens의 specific immune recognition에 어떻게 관여할 수 있는지를 암시합니다. TIL에 대한 microbial target antigens의 편견 없는 식별은 향후 개인 맞춤형 tumour 백신 접근법에 대한 가능성을 제시합니다.
Figure
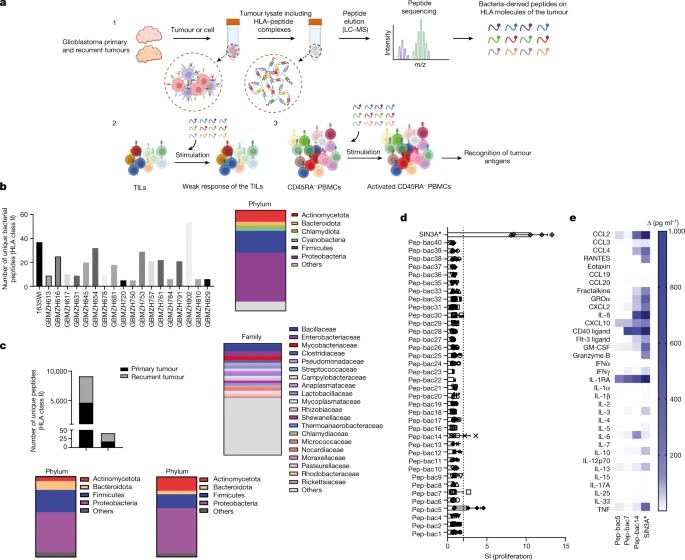
Fig. 1: Elution of HLA class II-presented bacterial peptides from 19 glioblastomas and the stimulatory effect of IPdBPs on bulk TILs.
그림 1: 19개의 glioblastomas에서 HLA class II-presented bacterial peptides의 Elution과 bulk TIL에 대한 IPdBP의 자극 효과.
a, glioblastomas 조직의 immunopeptidome analyses 모식도.
b, glioblastomas 환자(n = 19)의 고유 IPdBP 수 (왼쪽). 이 bacteria의 분류학에 따르면 이들은 주로 phylum 수준에서 Proteobacteria와 Firmicutes에서 유래한 것으로 나타났습니다(오른쪽).
c, primary and recurrent tumours의 IPdBP(위). primary (왼쪽 하단) 및 recurrent (오른쪽 하단) tumours의 IPdBP는 phylum 수준에서 source bacteria에 개별적으로 주석을 달았습니다.
d, Bulk TIL을 각각 5개의 replicate wells에서 IPdBP로 자극하고, 3H- thymidine incorporation을 통해 5일 후 증식을 측정했습니다.
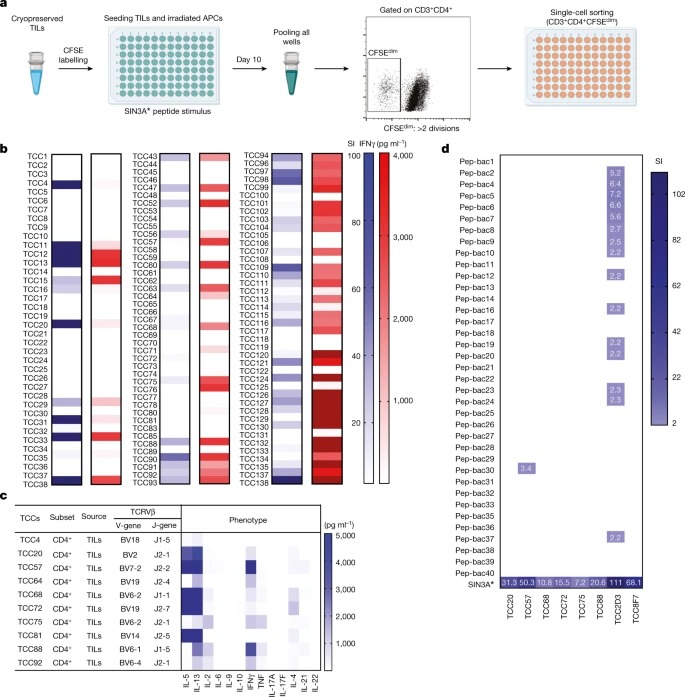
Fig. 2: SIN3A* peptide로 TIL을 자극하여 tumour-specific TCCs를 분리 및 characterization합니다.
a, Bulk TIL을 CFSE로 표지하고 SIN3A* peptide로 pulsed 된 APC로서 조사된 autologous PBMC로 자극했습니다.
b, 분리된 모든 TCC를 cognate antigen (SIN3A* peptide)과 대조하여 proliferation 및 IFNγ production을 테스트했습니다.
c, TCRB V-gene 및 J-gene 및 몇몇 TCC의 기능적 표현형.
d, tumour antigen (SIN3A*)에 대한 반응성을 가진 몇몇 TIL-derivedTCC를 대상으로 IPdBP에 대한 반응성을 테스트했습니다.
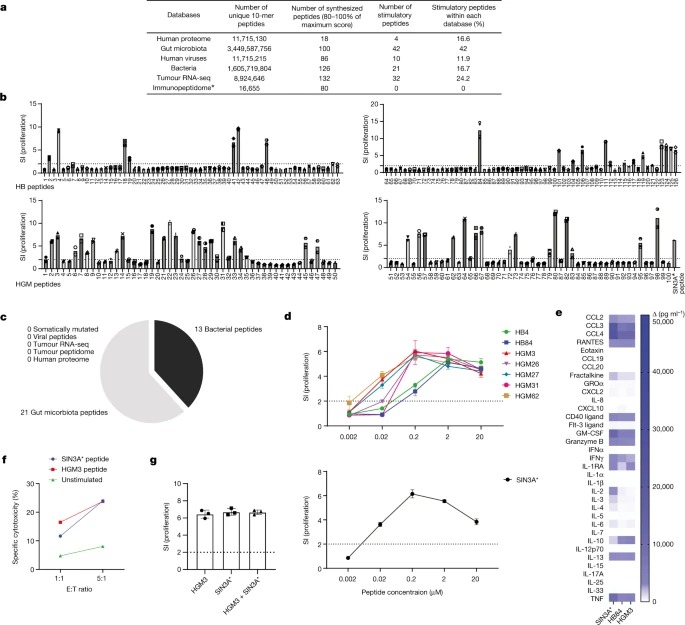
Fig. 3: Bacteria-derived and microbiota-derived peptides는 the tumour antigen뿐만 아니라 TCC88을 자극할 수 있습니다.
a, predicted stimulatory scores가 가장 높은 peptide는 다양한 데이터베이스를 검색하여 확인했습니다.
b, bacteria-derived (위) 및 microbiota-derived (아래) peptide로 3일간 TCC88을 자극한 후 3H- thymidine incorporation을 통해 증식을 측정했습니다.
c, 10µM 농도에서 cognate antigen (SIN3A* tumour 항원)과 동등하게 또는 더 잘 자극하는 모든 데이터베이스의 peptide 수를 표시하는 파이 차트입니다.
d, TCC88은 20, 2, 0.2, 0.02, 0.002 µM 등 농도를 낮추면서 여러 가지 강력한 stimulatory bacteria-derived and microbiota-derived peptides와 SIN3A* peptides로 테스트했습니다.
e, SIN3A*, HGM3 및 HB84 peptides로 자극한 후 TCC88에서 Supernatants을 수집했습니다.
f, 2µM의 SIN3A* 및 HGM3로 자극한 후 autologous tumour cell line에 대한 TCC88의 Cytotoxicity.
g, TCC88을 HGM3(10µM) 또는 SIN3A*(10µM) peptide 또는 둘 다(각각 10µM)와 함께 테스트하고 3일 후 3H-thymidine incorporation을 측정했습니다.
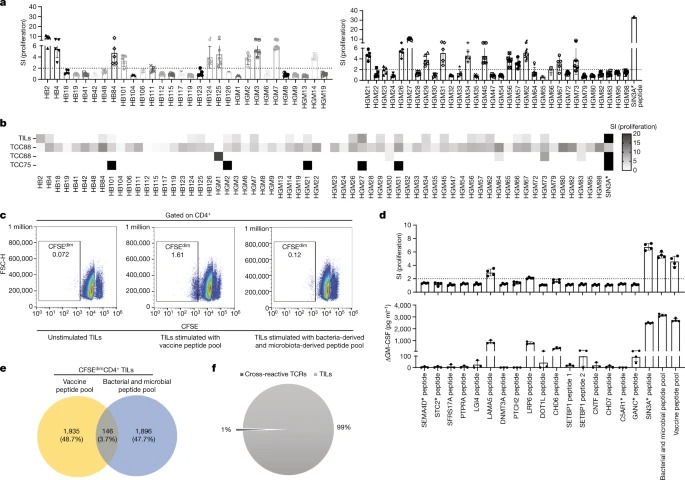
Fig. 4: bacteria-derived and microbiota-derived peptides에 대한 bulk TIL의 반응.
a, bulk TIL을 bacteria-derived and microbiota-derived peptides로 자극하고 autologous PBMC를 APC로 조사했습니다.
b, heatmap에 표시된 bacteria-derived and microbiota-derived peptides에 대한 여러 TCC 및 bulk TIL의 proliferative 반응에 대한 개요.
c, bulk TIL을 CFSE로 표지하고 bacteria-derived and microbiota-derived peptides pool과 autologous tumour에서 overexpressed된 somatic mutations및 genes의 peptides를 포함하는 백신 또는 tumour peptide 풀로 개별적으로 자극했습니다.
d, bacteria-derived and microbiota-derived peptides pool에 반응하는 확장된 CFSEdimCD4+ TIL을 여러 tumour antigens, bacteria-derived and microbiota-derived peptides pool, vaccine peptides pool로 자극했습니다.
e, bacteria-derived and microbiota-derived peptides pool과 vaccine peptides pool에 반응한 CFSEdimCD4+ TIL의 일부가 TCRVβ 시퀀싱에 사용되었습니다.
f, vaccine peptides pool과 bacteria-derived and microbiota-derived peptides pool이 모두 전체 TIL의 1%를 차지한다는 것을 인식하는 TIL에서 cross-reactive T cells의 TCR 서열 중첩.
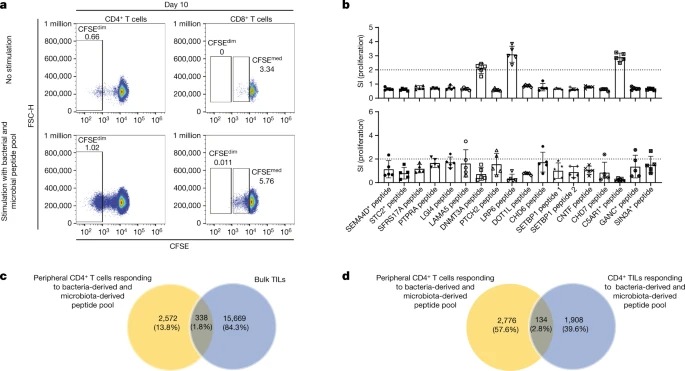
Fig. 5: Peripheral memory T cells는 bacteria-derived and microbiota-derived peptides로 활성화된 후 여러 tumour antigens을 인식합니다.
a, CD45RA- peripheral T cells를 분리하고 CFSE로 표지했습니다. 그런 다음 bacteria-derived and microbiota-derived peptides pool로 10일 동안 세포를 자극했습니다. 다음으로, 60개의 replicate wells을 풀링하고 proliferative (CFSEdim) CD4+ T 세포를 분류하고 short-term expanded했습니다.
b, Bacteria-reactive or microbiota-reactive peripheral CD4+ T cells는 조사된 autologous PBMC가 APC로 제시한 peptide로 자극했을 때 autologous tumour의 여러 tumour antigens에 반응했습니다(위쪽). Peripheral blood memory T cells를 분리하여 tumour antigens으로 직접 테스트했습니다(아래).
c,d, bacteria-derived and microbiota-derived peptide pool에 반응하는 peripheral CD4+ T cells의 일부(즉, proliferating)를 TCRVβ 시퀀싱에 사용했습니다.
Disscussion
본 연구는 예후가 좋지 않고 미충족 의료 수요가 높은 glioblastoma에서 tumour antigen-specific immune responses에 대한 bacteria의 역할을 조사합니다. 우리는 bacteria, 특히 gut microbiota의 관여와 이들이 면역 반응을 어떻게 형성하는지에 대한 최근 관찰을 기반으로 합니다. glioblastoma을 포함한 tumour의 bacterial communities, tumour의 immunopeptidome 내 bacterial peptides, tumour-specific immune responses을 자극하는 commensal bacteria and a bacteriophage의 영향에 대한 발견이 연구에 동기를 부여했습니다. 먼저 glioblastoma에서 bacteria 서열이 발견된다는 Nejman 등의 데이터와 glioblastoma 환자의 HLA class II immunopeptidomes에서 bacteria-derived and microbiota-derived peptides에 관한 Kalaora 등의 데이터를 확인했지만, 여기서는 glioblastoma에서 확인했습니다. IPdBP가 유래한 peptide와 bacteria 균주는 5개에서 50개 이상까지 다양했습니다. 따라서 일반적으로 수천 개인 tumour 유래 인간 peptide 서열과 비교했을 때(그림 1c, 상단), HLA class II immunopeptidomes에서 bacterial peptides의 풍부도는 낮았습니다. 이는 tumour 세포 및/또는 tumour-infiltrating APC에서 bacterial peptides가 HLA class II 분자의 소수만을 차지하고 검출 한계 이하로 떨어질 수 있는 bacteria 부담이 낮기 때문일 수 있습니다. Nejman 등20의 연구와 일관되게, 우리는 glioblastoma에서 Proteobacteria와 Firmicutes phyla가 가장 널리 퍼져 있는 것을 발견했습니다. 처음에는 IPdBP에 초점을 맞추었지만, 두 분석 모두 Enterobacterales, Burkholderiales, Bacillales, Clostridiales등의 bacterial antigens을 주문 수준의 계통형에서 확인했습니다. 이전에 tumour neoepitope peptide cocktails을 사용한 맞춤형 백신으로 치료한 적이 있는 환자를 대상으로 IPdBP가 TIL에 의해 인식되는지 여부를 테스트했습니다. 백신 peptide 중 하나인 돌연변이 SIN3A* peptide와 비교했을 때, TIL은 증식을 통해 37개의 IPdBP 중 3개에 매우 약하게 반응했지만, tumour으로의 cytotoxic CD8+ T cell 이동을 강화하는 CXCL10과 GM-CSF 및 TNF의 분비42가 반응성을 뒷받침합니다. CXCL10은 여러 tumour에서 항 PD1 치료에 대한 반응을 예측하는 데 사용되었습니다. 더 많은 pro-inflammatory SIN3A* 반응성 T세포를 조사했을 때, 10개 중 2개가 단일(TCC57) 또는 다중(TCC2D3) IPdBP를 인식하여 tumour과 IPdBP에 대한 cross-reactivity을 가진 T세포가 TIL에서 발견될 수 있음을 입증했습니다. 편향되지 않은 항원 발견 접근법을 통해 우리는 이전에 autoantigen-specific, virus-specific and bacteria-specific CD4+ TCC의 표적을 확인했으며, 이들 모두는 여러 항원을 인식했습니다. 따라서 우리는 이제 tumour-infiltrating T 세포에서 시작하여 TIL-derived TCC가 viral-derived, bacterial-derived, microbiome-derived, human protein-derived or tumour antigen-derived peptides에 반응하는지 여부를 다루었습니다. TIL-derived, SIN3A* 특이적 TCC88은 인간에 대한 영양성을 가진 모든 human proteins, viruses and bacteria with tropism for humans, gut microbiota, the translated proteome of the autologous primary and recurrent tumours and the tumour immunopeptidome 서열 중에서 표적 항원을 검색하는 프로토타입의 예로 사용되었습니다. 이러한 데이터베이스의 크기는 16,000개(immunopeptidome)에서 34억 개(human gut microbiota) 이상의 decamer peptide에 달했습니다(그림 3a). 우리는 TCC88에 대한 높은 자극 점수가 예측되는 다양한 소스에서 542개의 peptide를 합성했습니다(그림 3a). TCC88은 109개의 peptide에 반응했으며, 이 중 bacterial and microbiota-derived peptides에 가장 광범위하게 반응했지만 많은 tumour proteome-derived peptides에도 반응했습니다(그림 3a). 226개의 bacterial and microbiota-derived peptides 중 63개를 인식했으며, 이 중 34개는 cognate antigen과 동등하게 또는 더 잘 자극했습니다. B. ovatus and Bacillus sp., 외에 다른 bacteria-derived and microbiota-derived peptides는 IPdBP와는 완전히 다른 bacteria 종에서 유래되었습니다 (확장 데이터 그림 9c 및 보충 표 15). 이러한 데이터는 tumour-infiltrating TCC가 단일 bacterial, viral or phage-derived peptides 뿐만 아니라 병원성 bacteria와 gut microbiota-derived commensals peptide의 많은 수의 peptide도 교차 인식할 수 있음을 보여줍니다. MS에서 자가 반응성 TCC를 사용한 이전 데이터와 유사하게, 광범위한 교차 반응성을 설명하기 위해 stimulatory 와 non-stimulatory peptides를 구별할 수 있는 아미노산 패턴이나 HLA-binding or TCR-binding motifs를 확인하지 못했습니다(확장 데이터 그림 5b). TCC88의 주요 HLA restriction element는 DRB3*02:02이며, 이전에 설명한 virus-specific and autoreactive TCCs와 유사하게, TCC88은 교차 제한, 즉 다른 자가 HLA-DR 분자의 맥락에서도 peptide를 인식할 수 있습니다. 그 기능과 관련하여 TCC88은 autologous glioblastoma cell line에 대한 in vitro cytotoxicity과 TNF, IFNγ, granzyme B, GM-CSF 등을 강하게 분비하는 multifunctional pro-inflammatory phenotype을 보여주었습니다. SIN3A*와 두 가지 bacterial peptides에 대한 반응은 거의 동일하지만, bacterial peptides 자극 시 IL-10의 상대적으로 높은 분비는 후자가 intratumoural T cell responses을 활성화할 뿐만 아니라 약화시킬 수 있음을 나타낼 수 있습니다.
