인간의 뇌 organoid에서 cell fate regulome 추론과 교란
Abstract
single-cell genomic technology과 결합된 pluripotent stem cell에서 성장된 Self-organizing neural organoid는 인간 뇌 발달의 기초가 되는 유전자 조절 네트워크를 조사할 수 있는 기회를 제공한다.
여기서 우리는 neuroepithelial formation, patterning, brain regionalization and neurogenesis을 다루는 human organoid의 밀도 높은 시간 과정에 걸쳐 single-cell transcriptome과 접근 가능한 chromatin data를 획득하고 temporally dynamic 및 brain-region-specific regulatory region을 식별한다. 우리는 유기체 발달을 설명하는 글로벌 유전자 조절 네트워크를 추론하기 위해 multi-omic data와 transcription-factor-binding site의 예측을 통합하는 유연한 프레임워크인 Pando를 개발했다. 우리는 single-cell transcriptome 판독과 함께 합동된 genetic perturbation를 사용하여 organoid의 cell fate과 상태 조절에 대한 transcription factor requirement를 평가한다. 우리는 특정 요인들이 cell fate의 abundance를 조절하는 반면, 다른 요인들은 분화 후 neuronal cell state에 영향을 미친다는 것을 발견했다. 우리는 transcription factor GLI3가 mammalian 모델 시스템에서 수행된 이전 연구를 요약하여 인간의 cortical fate 확립에 필요하다는 것을 보여준다. 우리는 정상 세포 또는 GLI3-perturbed cell에서 transcriptome 및 chromatin 접근성을 측정하고 telencephalic fate 결정의 핵심인 두 가지 구별되는 GLI3 regulome을 식별한다. 하나는 직접적인 GLI3 타겟으로 HES4/5를 사용하는 dorsoventral 패턴화, 다른 하나는 나중에 개발에서 ganglionic eminence diversification를 제어하는 것이다.
우리는 함께 인간 모델 시스템과 single-cell technology를 활용하여 인간의 발달 생물학을 재구성하는 방법에 대한 프레임워크를 제공한다.
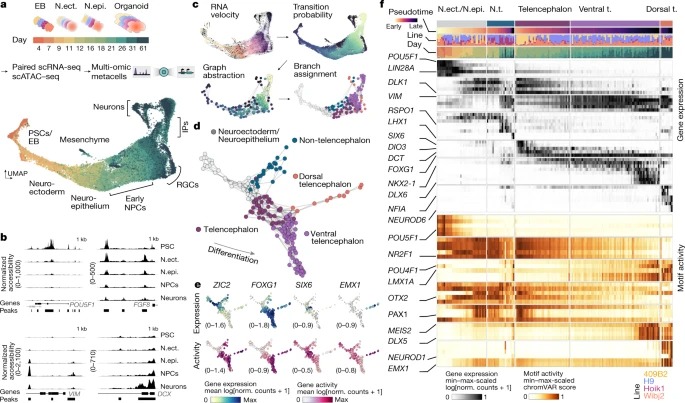
Fig. 1: brain organoid development의 multi-omic atlas는 발달 계층과 운명 결정의 중요한 단계를 보여준다.
a, integrated multi-omic metacell의 실험 설계 및 UMAP 임베딩의 개략도.
b, pluripotency 에서 organoid 발달 동안 차등 접근성을 갖는 loci의 예.
c, branch-inference strategy의 개략도.
d, force-directed layout의 branch visualization.
e, 마커 유전자의 평균 발현(log[transcript counts per 10,000 + 1])(상단) 및 gene activity (log[transcript counts per 10,000 + 1])(하단)으로 염색된 regional branch 그래프.
f, Stage- and branch-specific 유전자 발현 및 모티프 enrichment z-score.
Figure 2: Pando는 multimodal 측정을 활용하여 인간 뇌 organoid 발달의 기본이 되는 multiphasic GRN을 추론한다.
a, Pando GRN-inference 프레임워크의 개략도.
b, 다양한 transcription factor에 대한 다양한 branches 및 추론된 조절 영역에서 EMX1의 transcription start site에서 정규화된 접근성을 보여주는 신호 트랙.
c, TF 간의 co-expression 및 추론된 상호작용 강도를 기반으로 하는 추론된 TF 네트워크의 UMAP 임베딩.
d, 모듈 기능으로 음영 처리된 UMAP 임베딩
e, branch-specific TF에 대한 타겟 특이성.
f, branch-specific 타겟이 있는 TF 및 branch-specific 액세스 가능성과의 상호 작용을 강조하는 branch-specific TF 네트워크의 UMAP 임베딩.
g, dorsal (빨간색)과 ventral (보라색) telencephalon branch 사이에 차등 활성을 갖는 TF 그룹
Fig. 3: mosaic organoid의 TF perturbation은 neurodevelopmental fate decision에 critical regulator이다.
a, CRISPR droplet 시퀀싱(CROP-seq) 방법을 사용한 single-cell TF perturbation 실험의 개략도.
b, primary and organoid cortex의 NPC, IP 및 뉴런에서 표적 유전자의 최소-최대 규모 평균 발현(log[10,000 + 1당 전사 수]).
c, 각 실험에 대한 각 perturbation이 있는 세포의 비율.
d, 검출된 gRNA(좌측) 및 branch assignment (우측)에 의해 착색된 세포로 UMAP 임베딩.
e, gRNA의 Regional enrichment.
f, 운명 조절에 강한 영향을 미치는 선택된 유전자에 대해 일관된 gRNA로 염색된 UMAP 임베딩.
g, HES1-target (상단, n= 18 genes) 및 GLI3-target(하단, n=42 genes) 유전자의 dorsal branch로 이동 확률의spearman 상관 관계.
h, inducible CRISPR–Cas9 nickase 시스템을 사용한 GLI3 loss-of-function 실험의 개략도.
i, 6-week-old WT 및 GLI3-KO 뇌 organoid의 scRNA-seq 데이터의 UMAP 임베딩은 NPC에서 regional branch에 할당된 다른 클러스터로 색칠된 뉴런까지의 궤적을 보여준다.
j, 각 조건에 대한 organoid당 클러스터(색상) 할당의 분포를 보여주는 누적 막대 플롯.
k, GLI3-KO 데이터 및 GLI3 gRNA 를 포함하는 CROP-seq 데이터 에 대한 ventral telencephalic neuron에서의 차등 발현(DE).
Fig. 4: GLI3 loss of function의 single-cell multiome view는 dorsoventral telencephalon specification의 뚜렷한 regulomes and effector를 나타낸다
a, 뇌 organoid 발달 3주에 동일한 세포에서 transcriptome and chromatin 접근성을 측정하는 실험의 개략도.
b, 클러스터에 의해 착색되고 예상된 세포 운명에 의해 표지된 UMAP 임베딩.
c, 다른 클러스터에서 GLI3- KO 세포에 대한 대조군(WT)의 DEG 수.
d, GLI3 KO 후 telencephalic progenitor (클러스터 0 및 2)에서의 차등 발현 .
e, 초기 telencephalic progenitor (3주차), ventral telencephalic progenitor (6주) 및 뉴런(6주)에 대한 GLI3 KO 후 DEG 및 early telencephalic progenitor (3주차)에서 GLI3 KO후 차등 접근성.
f, g , 1차 및 2차 GLI3 타겟을 보여주는 early telencephalic ( f ) 및 ventral telencephalon ( g ) progenitor에 대한 GRN 하위 그래프.
h, WT organoid의 GLI3 결합 점수(유전자 본체에 대한 CUT&Tag 신호 강도의 합 + 2kb) 대 early telencephalic progenitor (3주차)에서의 차등 발현의 로그 변환 배수 변화.
i, 모듈 유전자 발현과 차등적으로 발현된 TF에 대한 분지 확률(분지 활성화 점수) 간의 z- scored 평균 상관관계.
j, SHH 대 GLI3 KO 처리 후 유전자의 로그 변환 배수 변화.
k, GLI3 및 SHH perturbation의 결과를 요약한 개략도.
Discussion
인간의 뇌는 다른 종들과 구별되는 독특한 특징을 가지고 있다. 최근의 cell atlas의 노력으로 생쥐와 인간이 발달하는 뇌세포 구성에 대한 high-resolution의 설명에도 불구하고, 뇌 패턴의 초기 단계에서 조직을 얻는 것이 어렵고, 유전자 기능을 체계적으로 조작하는 방법들이 부족하기 때문에 인간의 뇌 발달을 조절하는 메커니즘을 연구하는 것은 주요한 도전이었다. 여기서 우리는 transcriptome, chromatin accessibility and genetic perturbation dataset를 통합하여 인간의 뇌 regionalization의 기초가 되는 메커니즘에 대한 통찰력을 제공한다. 넓은 의미에서, 우리는 마우스와 다른 비인간 모델 시스템에서 확인된 프로그램이 인간에게 잘 보존되어 있다는 것을 발견했고, 줄기세포에서 파생된 뇌 조직이 이러한 프로그램을 재구성하는 정도가 눈에 띈다. 우리는 rodent telencephalon에서 dorsoventral fate specification을 제어하는 잘 연구된 transcription factor로 GLI3에 초점을 맞췄다. 우리는 이 같은 transcription 프로그램이 인간에게 잘 보존되어 있다는 명확한 증거를 발견했다. 중요한 것은, 이러한 데이터가 multiregion 인간 뇌 organoid는 예측 모델 시스템이 될 수 있다는 강력한 증거를 제공한다는 것이다. 유도되지 않은 neural organoid 프로토콜은 각 organoid 또는 batch에 표현되는 영역의 비율과 관련하여 stem line 간에 강한 변화를 초래한다는 점에 유의하십시오.
우리는 개발 프로그램의 글로벌 분석에 이전에 사용되지 않았던 regulatory genome의 특징을 통합한 Pando GRN 추론 프레임워크를 설정했다. Pando는 transcriptome, chromatin accessibility, an expanded TF family motif reference, 알려진 CRE 및 진화 보존을 유연한 프레임워크로 결합하여 multimodal dataset에 대한 regression based GRN 추론을 일반화한다. R 패키지는 candidate region selection, motif matching, model fitting and discovery of gene and regulatory modules을 포함한 전체 GRN 추론 전략을 구현한다. 또한 GRN 추론에 사용할 수 있는 광범위한 regression 모델을 제공한다. 우리는 pluripotency에서 neuroectoderm을 거쳐 neuroepithelium로 전환에 관련된 TF 모듈뿐만 아니라 regionalized된 뇌 상태와 관련된 하위 네트워크와 같은 네트워크의 흥미로운 측면을 강조했습니다. 이러한 네트워크 분석은 특정 뉴런 상태를 프로그래밍하도록 설계된 미래 실험을 안내할 수 있으며, human organoid의 gene perturbation을 해석하는 데 사용될 수 있다. 현재의 한계에는 organoid 발달 전반에 걸친 comprehensive active 및 repressive histone modification과 chromatin conformation status의 부족과 불완전한 TF 모티프 데이터베이스가 포함된다. 우리는 이러한 것들이 활발한 연구 영역이 될 것으로 예상하며, Pando는 그러한 이전을 GRN 추론 프레임워크에 포함할 수 있는 유연성을 가지고 있다.
우리는 인간에서 dorsal telencephalic cell-fate specification에서 GLI3의 중요한 역할을 검증했고, MGE 및 LGE/CGE 뉴런의 사양 중 GLI3의 기여를 추가로 확인했다. GLI3-KO organoid와 글로벌 GRN의 single-cell multiome 데이터의 통합은 neuroepithelial regionalization 동안 SH signalling을 통해 GLI3가 초기 뇌 NPC에서 유도되는 모델을 제안했다. 그런 다음 GLI3는 하류 표적을 조절하여 HES5, HES4 및 HES1의 differential activity를 통해 cortical fate acquisition을 활성화하고 BCL11A, LHX8, NKX2-1의 조절을 통해 MGE 유도 프로그램을 억제한다. 우리의 데이터는 또한 GLI3가 HES 유전자를 직접 조절할 수 있음을 시사하는데, 아마도 최근 마우스 limb development 중에 설명된 것과 유사한 NOTCH-independent 메커니즘을 통해서일 것이다. 더 넓게 말하면, 우리의 데이터는 인간 뇌 발달의 유전자 조절 프로그램을 이해하는 multimodal single-cell genomic and organoid technology의 놀라운 잠재력을 보여준다.