초기 중세 유럽의 고해상도 유전자 역사
Abstract
많은 알려진 및 알려지지 않은 역사적 사건들이 유전 연구의 탐지 한계 아래에 남아 있습니다. 이는 미세한 조상 변화가 재구성하기 어려워서 그렇습니다. 공유된 하플로타입과 희귀 변이를 기반으로 한 방법들은 유용성을 개선하지만, 시간적 요소를 명시적으로 다루지 않으며, 편향 없는 조상 모델에서는 적용하기 어려웠습니다.
여기에서는 최근 시기의 조합을 중심으로 하여 통계적 파워를 여러 배 향상시킬 수 있는 시간 계층화된 조상 분석 접근법인 Twigstats를 개발했습니다. 이 접근법은 인구 특유의 변동에 의해 편향되지 않으면서도 유전자 조합을 개선할 수 있습니다. 우리는 이 프레임워크를 유럽에서의 역사적 시기의 1,556개의 고대 전체 유전체에 적용했습니다. 이전의 유전체들을 사용하여 개인 수준에서 고해상도 조상을 모델링할 수 있었습니다.
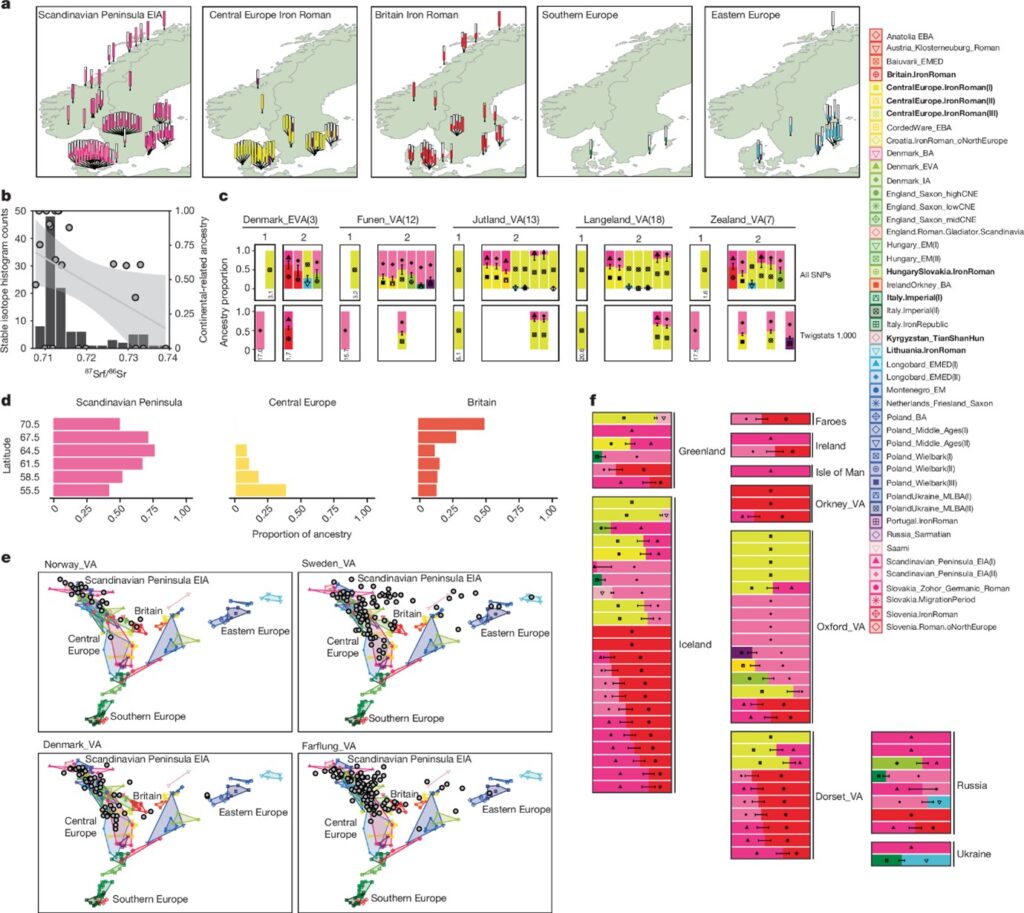
첫 번째 천년기의 전반부 동안, 우리는 최소 두 가지의 서로 다른 스칸디나비아 관련 조상 계통이 서유럽, 중앙유럽 및 동유럽 전역에 확장되는 모습을 관찰했습니다. 반대로, 첫 번째 천년기의 후반부 동안, 조상 패턴은 이러한 계통들의 지역적 소멸 또는 상당한 혼합을 시사합니다. 스칸디나비아에서는 약 800년 경에 중요한 조상 유입이 있음을 문서화했습니다. 이때, 대다수의 바이킹 시대 개인들이 초기 철기 시대의 개인들에서는 볼 수 없었던 중앙유럽과 관련된 집단의 조상을 지니고 있었습니다. 우리의 연구 결과는 시간 계층화된 조상 분석이 유전자 역사에 대한 고해상도 렌즈를 제공할 수 있음을 시사합니다.