FANCD2–FANCI는 DNA를 감시하고 이중 가닥에서 단일 가닥으로의 접합부를 인식합니다
Abstract
DNA 교차결합은 DNA 복제를 차단하며, 이는 Fanconi anaemia 경로에 의해 복구됩니다. FANCD2–FANCI (D2–I) 단백질 복합체는 이 과정에서 중심적인 역할을 하며, 병변 주변에서 DNA 절개를 조정하여 복구를 시작합니다. 그러나 D2–I는 DNA 복구에서 더 일반적인 역할을 하며, 비정상적인 분해로부터 멈춘 복제 포크를 보호하는 역할도 합니다. 현재로서는 DNA 교차결합이 어떻게 인식되며 D2–I가 복제 포크 보호에서 어떻게 기능하는지가 불분명합니다.
여기서 우리는 single-molecule imaging을 사용해 D2–I가 이중 가닥 DNA에 결합하여 확산하는 슬라이딩 클램프임을 보여줍니다. 특히, 슬라이딩하는 D2–I는 단일 가닥–이중 가닥 (ss–ds) DNA 접합부와 마주칠 때 멈추는데, 이러한 구조는 복제 포크가 DNA 병변에 멈췄을 때 생성됩니다. cryogenic electron microscopy을 사용해 DNA 위의 D2–I 구조를 분석한 결과, 멈춘 D2–I가 슬라이딩하는 D2–I와는 다른 방식으로 ss–dsDNA 접합부와 특정 상호작용을 한다는 것을 밝혀냈습니다. 따라서 D2–I는 이중 가닥 DNA를 감시하며, ssDNA 간격에 도달하면 ss–dsDNA 접합부에 특이적으로 결합합니다. ss–dsDNA 접합부는 멈춘 복제 포크에서 발견되므로, D2–I는 DNA 손상 부위를 식별할 수 있습니다. 따라서 우리의 데이터는 여러 DNA 복구 경로에서 D2–I가 멈춘 복제 포크를 인식하고 보호하는 역할을 통합적으로 설명하는 분자적 메커니즘을 제공합니다.
Figures
D2–I slides on DNA
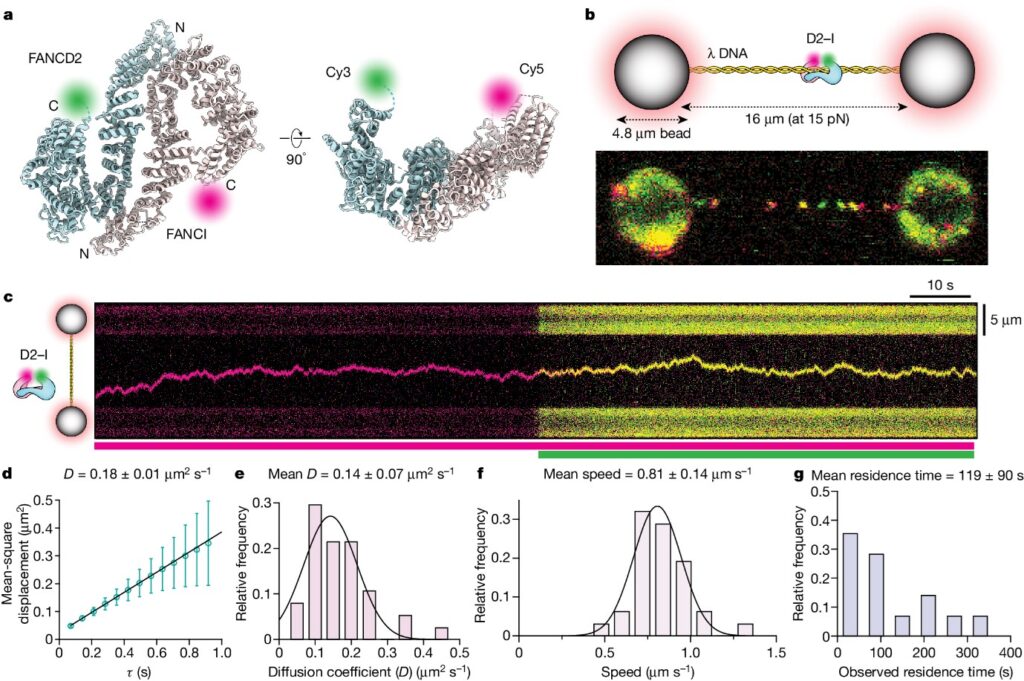
[Figure 1] DNA 위에서 D2–I 클램프의 single-molecule imaging
(A) 형광 염료 위치를 나타내는 열린 D2–I 클램프 모델의 직교 뷰 (PDB 6TNG).
(B) 상단: single-molecule 실험 장비의 개략적 설명. 하단: 라벨이 붙은 D2–I가 λ DNA에 결합한 상태를 보여주는 공초점 스캔.
(C) D2–I에 의한 일차원 랜덤 확산을 보여주는 대표적인 키모그래프.
-아래의 색 막대는 사용된 excitation laser를 나타냅니다 (자주색, Cy5; 녹색, Cy3).
(D) c의 키모그래프에서 추출한 단일 궤적의 평균 제곱 변위 (Mean square displacement : MSD)는 0.18 ± 0.01 μm²/s의 확산 계수 (D)를 나타냅니다.
(E) 개별 D2–I 입자의 계산된 D 분포와 가우스 적합.
-Mean D는 0.14 ± 0.07 μm²/s (n = 37)를 나타냅니다.
(F) λ DNA에서 개별 D2–I 분자의 속도 분포.
-Mean speed는 0.81 ± 0.14 μm/s (n = 31)를 나타냅니다.
(G) λ DNA에서 관찰된 D2–I 궤적의 체류 시간 분포 (n = 28).
D2–I bounces off non-specific obstacles
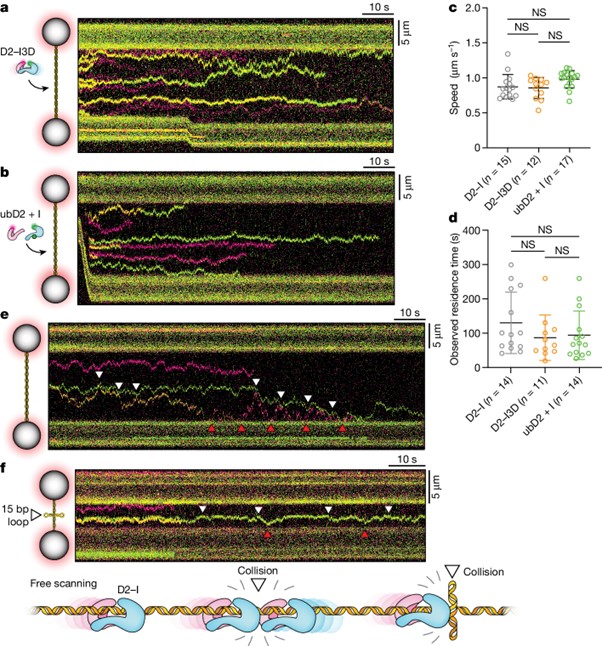
[Figure 2] DNA에서 D2–I 슬라이딩의 특성화
(A, B) 실험 장비의 개략적 설명 (왼쪽)과 15 pN에서 획득한 λ DNA 위의 phosphomimetic D2–I3D (a) 및 monoubiquitinated ubD2–I (b) 복합체의 대표적인 키모그래프 (오른쪽).
(C, D) D2–I, D2–I3D 및 ubD2–I의 속도 분포 (c)와 관찰된 체류 시간 (d)의 비교.
(E) 15 pN에서 λ DNA 위의 D2–I 키모그래프.
-다른 D2–I 분자와의 분자 충돌은 흰색 화살표로 표시되었으며, D2–I와 고정된 비드 사이의 충돌은 빨간색 화살표로 표시되었습니다.
(F) 중앙에 4방향 접합부를 포함하는 15 kb DNA에서 D2–I를 보여주는 키모그래프 (상단)와 개별 슬라이딩 D2–I 복합체와 DNA 루프가 있는 D2–I 사이의 만남을 나타내는 개략적 설명 (하단).
-D2–I와 15 bp DNA 루프 또는 D2–I와 비드 사이의 분자 충돌은 각각 흰색 및 빨간색 화살표로 표시되었습니다. 이 키모그래프는 10번의 독립적인 실험에서 관찰된 대표적인 결과입니다.
D2–I stalls at ss–dsDNA junctions
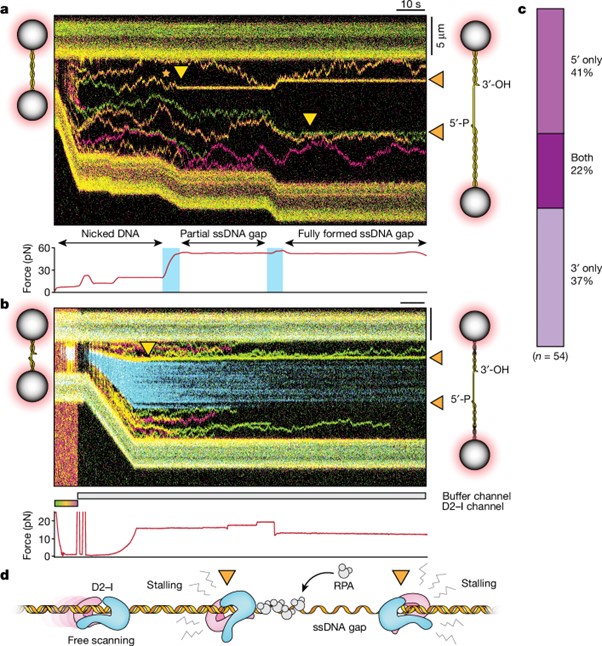
[Figure 3] ss-dsDNA 접합부에서 멈추는 슬라이딩 D2–I 복합체
(A) 특정 위치에 정의된 ssDNA 간극이 있는 λ DNA에 결합된 D2–I의 키모그래프.
(B) 파란색 eGFP-표지된 RPA가 ssDNA 간극을 덮고 있는 간극이 있는 λ DNA에서의 D2–I 키모그래프.
-노란색 화살표는 ss-dsDNA 접합부에서 정지된 D2–I의 위치를 나타냅니다. 키모그래프 아래의 색 막대는 채널 위치를 나타냅니다 (회색, 버퍼 채널; 녹색–노란색–빨간색, D2–I 채널). 힘 측정값은 키모그래프 아래에 표시됩니다.
(C) ss-dsDNA 접합부의 3′ 말단, 5′ 말단 또는 양쪽 말단에서 정지한 D2–I 복합체가 포함된 키모그래프의 정량화 결과.
(D) dsDNA에서 슬라이딩하고 ssDNA 간극을 둘러싼 RPA-bound ss-dsDNA 접합부 (주황색 화살표)에서 정지하는 D2–I의 개략적 설명.
[Fig. 3A] 키모그래프의 시작에서 λ DNA는 두 개의 특정 부위에서 절단됩니다. 두 개의 절단 부위 사이의 ssDNA 조각이 두 단계로 분리되어 17.8 knt의 ssDNA 간극이 생성되도록 힘을 가합니다. 키모그래프 아래에는 힘이 표시되며, 스트레칭 지점은 파란색으로 음영 처리되었습니다. 힘을 가하기 전과 후의 DNA 개략도가 각각 키모그래프의 왼쪽과 오른쪽에 표시되어 있습니다. 주황색 화살표는 ss-dsDNA 접합부를, 노란색 화살표는 ss-dsDNA 접합부에서 정지한 결합 이벤트를 나타냅니다. 주황색 별표는 ssDNA 간극이 생성되면서 정지하게 되는 이동 궤적을 나타냅니다.
Structure of a sliding D2–I on DNA
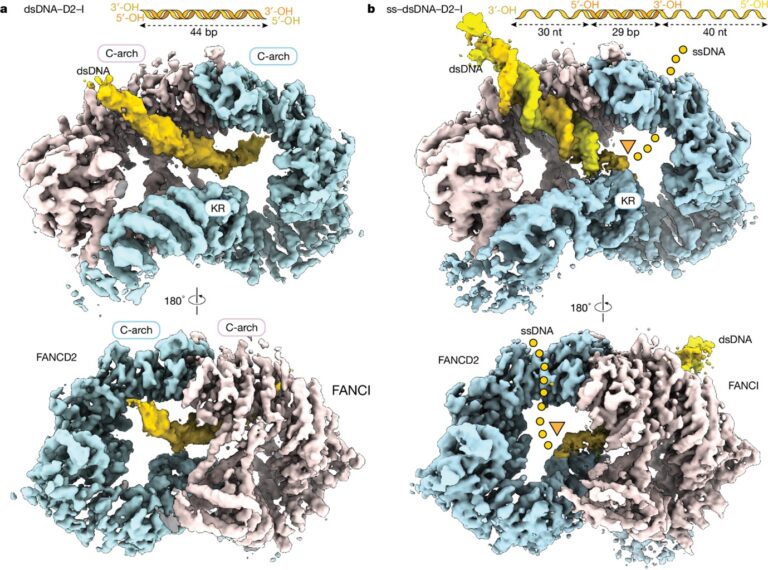
[Figure 4] dsDNA 또는 ss-dsDNA 접합부와 결합한 D2–I의 구조
(A) dsDNA–D2–I의 복합체 냉동 전자 현미경 (cryo-EM) 맵.
(B) ss-dsDNA–D2–I의 선명한 cryo-EM 맵.
ss–dsDNA junction recognition by D2–I
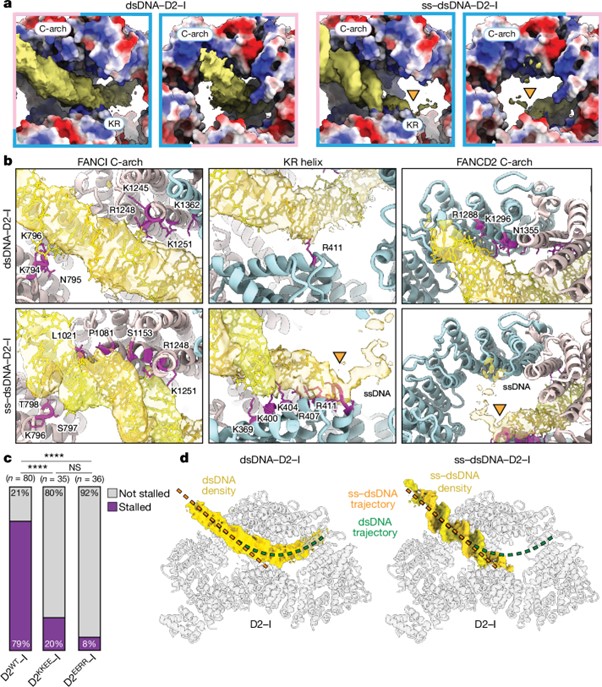
[Figure 5] dsDNA 또는 ss-dsDNA 접합부와 결합한 D2–I의 상호작용 세부 사항
(A) D2–I의 전기적 잠재력을 표면 표현으로 나타낸 그림.
-전기적 잠재력은 -10(kT) (빨간색)에서 +10(kT) (파란색)까지이며, 중성 순 전하를 가진 영역은 흰색으로 표시되었습니다. FANCD2의 C-아치(연한 파란색으로 라벨)와 FANCI (연한 분홍색으로 라벨), 그리고 FANCD2의 KR 나선이 라벨링 되어 있습니다.
(B) D2–I와 dsDNA (상단) 또는 ss-dsDNA (하단)의 상호작용의 차이.
-FANCD2는 연한 파란색, FANCI는 연한 분홍색, DNA는 노란색과 금색으로 표시되었습니다. DNA 모델에서 4.5 Å 거리 내에 있는 D2–I의 잔기 (residue)는 보라색으로 표시되며 ss-dsDNA 접합부는 주황색 삼각형으로 표시되었습니다.
(C) ss-dsDNA 접합부에서 멈춘 D2–I 궤적을 보여주는 키모그래프 수의 정량화.
(D) D2–I에서의 DNA 궤적은 dsDNA와 ss-dsDNA에 대해 다름을 보이는 모델.
[Fig 5D] 투명한 D2–I 모델 위에 세분화된 DNA 밀도를 표시하여 dsDNA (왼쪽)와 ss-dsDNA (오른쪽)가 따라가는 궤적을 보여줍니다. 각 상태의 중축을 추적하는 선이 비교를 위해 표시되었습니다 (dsDNA는 녹색, ss-dsDNA는 주황색).
The KR helix is important for DNA repair
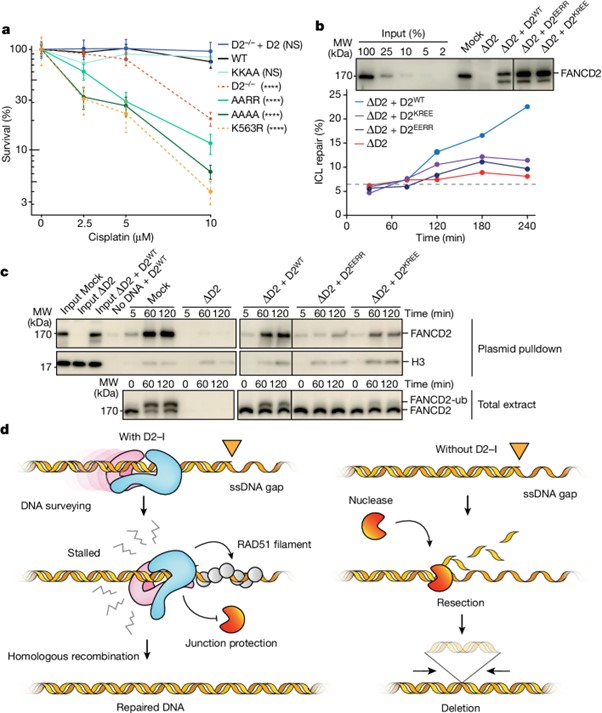
[Figure 6] DNA 복구에 중요한 역할을 하는 KR 나선
(A) DT40 FANCD2−/− 세포에서 clonogenic survival assay 를 통해 cisplatin 감수성을 평가했습니다.
-WT (Wild Type), FANCD2K563R (유비퀴틴화 결핍), 또는 KR 나선 변이체 (KKAA, AARR, AAAA)로 보완된 세포를 사용했습니다.
(B) KR 나선 돌연변이체는 ICL 복구가 손상되었습니다. Mock 처리한 (Mock) 및 FANCD2가 결핍된 (ΔD2) 핵질 Xenopus 난자 추출액을 야생형 (D2WT) 또는 돌연변이 FANCD2 (D2EERR 또는 D2KREE)로 보완했습니다. FANCD2 수준은 웨스턴 블롯 (상단 패널)으로 분석했습니다.
(C) ICL 복구 중 DNA 결합과 FANCD2 유비퀴틴화.
(D) D2–I가 dsDNA를 스캔하고 ss-dsDNA 접합부에서 멈추는 모델.
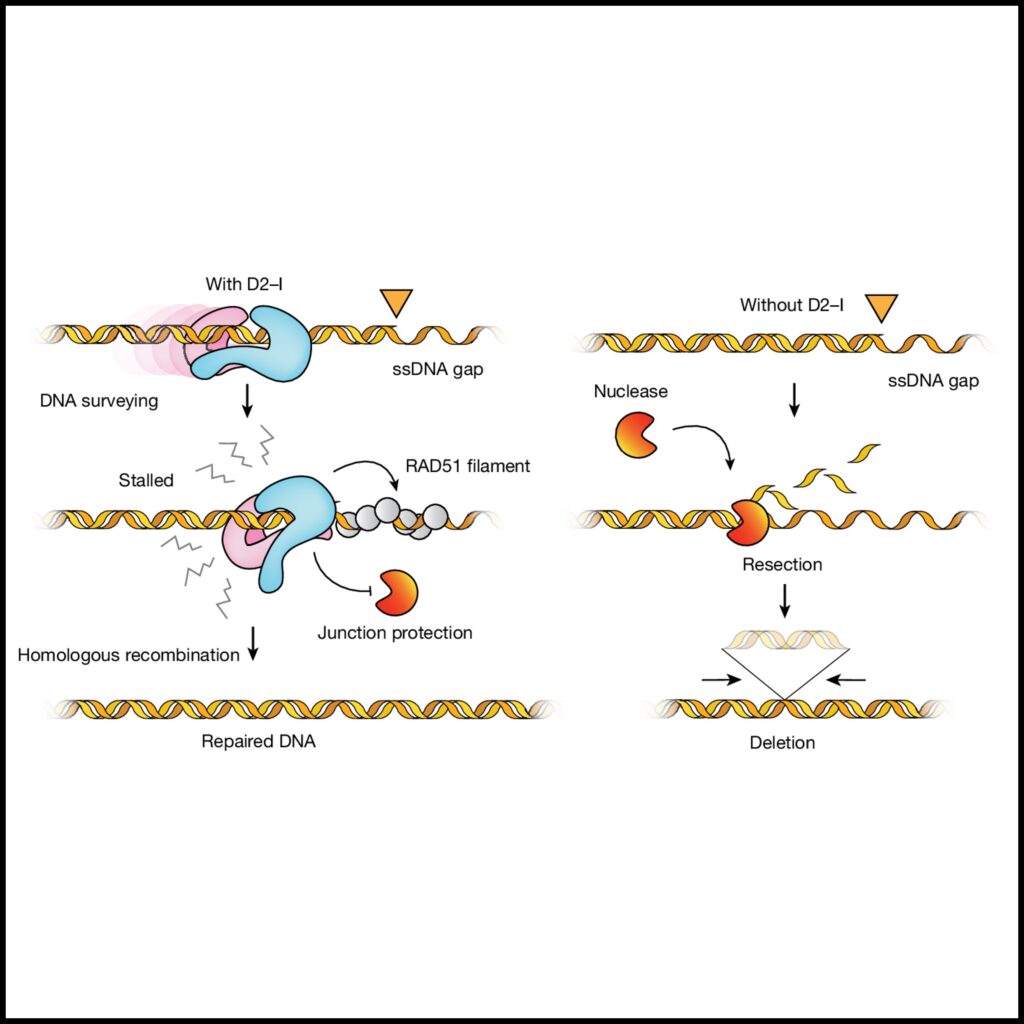
[Fig 6D] D2–I는 chromatin에 로딩되어 무작위 1차원 확산을 통해 dsDNA의 무결성을 검사합니다. 복제 포크가 멈출 때 발생하는 노출된 ssDNA 간극은 D2–I가 ss-dsDNA 접합부에서 멈추게 만듭니다. 닫힌 D2–I는 ss-dsDNA 접합부를 단단히 고정하여 추가 절단을 방지하고 DNA 손상을 최소화합니다. D2–I가 없을 경우 (오른쪽), 보호되지 않은 접합부는 더 광범위하게 절단되어 더 자주 발생하고 더 긴 염색체 삭제가 일어납니다.
Disscussion
이 연구에서 우리는 수정되지 않은 D2–I 클램프가 닫혀서 긴 dsDNA 구간을 스캔할 수 있음을 보여줍니다. D2–I는 다른 D2–I 클램프나 DNA 장애물을 우회할 수 없으므로 D2–I 스캔은 open chromatin에 제한됩니다. 이에 따라 우리는 D2–I가 복제 포크 주변에서 발생하는 nucleosome이 결핍된 영역을 조사한다고 제안합니다. 만약 복제체가 멈추면, 노출된 ss–dsDNA 접합부가 스캔 중인 D2–I에 의해 인식될 것입니다. ss–dsDNA 접합부에 위치한 닫힌 D2–I는 따라서 멈춘 복제 포크를 식별하게 되어, 여러 유형의 DNA 복구와 DNA 복제 스트레스에서 D2–I의 역할을 설명하는 통합된 모델을 제공합니다.
우리의 모델은 D2–I가 멈춘 복제 포크를 어떻게 보호할 수 있는지도 설명합니다. D2–I가 ss–dsDNA 접합부에서 이중 가닥을 물리적으로 고정함으로써, dsDNA 영역이 풀리거나 추가적인 절단이 발생하는 것을 막을 수 있습니다. 이 모델에 따르면, FANCD2를 결손시킨 쥐에서는 염색체 결손의 수와 크기가 증가한다는 결과와 일치합니다. 이러한 염색체 결손은 FANCD2가 없는 상태에서 ss–dsDNA 접합부가 nuclease에 민감해지면서 발생할 수 있습니다. FANCI 단독으로는 dsDNA에 결합할 수 있지만, 닫힌 DNA 클램프를 형성할 수 없기 때문에 FANCD2가 없는 상태에서는 복제 포크를 보호하지 못합니다. 이 가설에 대한 추가 검증은 앞으로의 연구 과제가 될 것입니다.
D2–I는 RAD51과 직접 상호작용하며, 멈춘 복제 포크에서 RAD51 필라멘트의 형성을 촉진합니다. DNA 교차 결합의 경우, D2–I의 유비퀴틴화가 ss–dsDNA 접합부에 이를 고정시켜 Fanconi anaemia 경로의 다른 하위 요소들을 모집하게 합니다. 이 데이터들은 D2–I가 ss–dsDNA 접합부를 식별하고 이를 보호하며, 하위 요소들을 모집하여 상동 재조합을 통한 DNA 수리를 시작하도록 돕는다는 것을 시사합니다. 다른 DNA 클램프들도 DNA 손상 부위로 수리 효소들을 모집하는 플랫폼 역할을 합니다 (예: 9-1-1 복합체와 PCNA). 이러한 클램프들은 ATP에 의해 구동되는 로더에 의존하여 DNA에 결합하지만, D2–I는 스스로 dsDNA에 결합하고 고정되며, 번역 후 수정에 의해 조절됩니다.
DNA 교차 결합을 인식하는 것이 무엇인지, 그리고 D2–I가 왜 여러 유형의 DNA 수리 및 복제 스트레스에 연관되어 있는지는 오랫동안 미스터리였습니다. 우리의 연구는 D2–I가 멈추거나 역전된 복제 포크 주변 구조에 특이적으로 위치하며, 이를 통해 교차 결합의 결과를 식별한다는 것을 제안합니다. ss–dsDNA 접합부와 멈춘 복제 포크는 복제 스트레스와 DNA 손상 반응에서 일반적으로 나타나는 특징이기 때문에, 이 메커니즘은 D2–I의 포괄적인 역할을 통합하여 DNA 수리에서의 그 역할을 설명하는 통합된 분자 기능을 제공합니다.

