유전체 및 질병 연관성을 통한 대규모 혈장 단백체의 비교
Abstract
High-throughput 단백체학 플랫폼은 혈장에서 수천 개의 단백질을 측정합니다. 이를 유전체 및 표현형 정보와 결합하면 유전체 정보와 질병 사이의 gap을 연결할 수 있는 힘을 가지고 있습니다. 우리는 유전형 및 표현형 데이터가 있는 50,000명 이상의 UK Biobank 참가자의 혈장 샘플에서 UK Biobank Pharma Proteomics Project에 의해 생성된 Olink Explore 3072 데이터의 연관성 분석을 수행하였습니다. 이때 영국 또는 아일랜드, 아프리카 및 남아시아의 조상에 따라 계층화하여 분석을 진행하였습니다. 우리는 이번 결과를 36,000명의 아이슬란드 사람들의 플라즈마에 대한 SomaScan v4 연구의 결과와 비교하였습니다. 3,6000명 중 1,514명의 경우는 Olink 데이터도 보유하고 있었습니다. 두 플랫폼 간에는 적은 상관관계를 발견하였습니다. 두 플랫폼에서 cis pTQL loci의 경우 유사한 개수가 감지되었지만 (Olink에서 2,101개 대 SomaScan에서 2,120개), 검사 성능을 support 하는 assay의 비율은 Olink 플랫폼에서 더 높았습니다(72% 대 43%). 상당한 수의 유전적 연관성을 가진 단백질이 플랫폼간 차이가 있었습니다. 우리는 단백질 수준의 통합 분석을 통한 질병 연구의 결론이 플랫폼 간의 차이로 인해 영향을 받는 예시를 제공합니다. 우리는 UK Biobank의 참가자들의 다양한 유전적 배경을 활용하여 새로운 연관성을 발견하고 유전적 위치를 더욱 정밀하게 지정하는 방법을 보여줍니다. 우리의 연구는 가장 흔히 사용되는 두 가지 high-throughput 단백체학 플랫폼에 의해 제공되는 정보의 가치와 차이점을 보여주며, 때로는 이를 상호보완적으로 사용하는 것이 유용할 수 있음을 보여줍니다.
Figure
정밀도의 비교
– 두 단백체 플랫폼 검사 결과의 반복성(재현성)과 상관관계의 평가
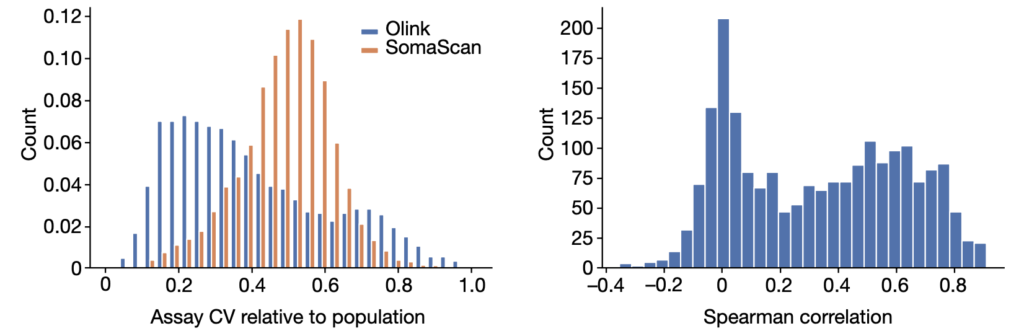
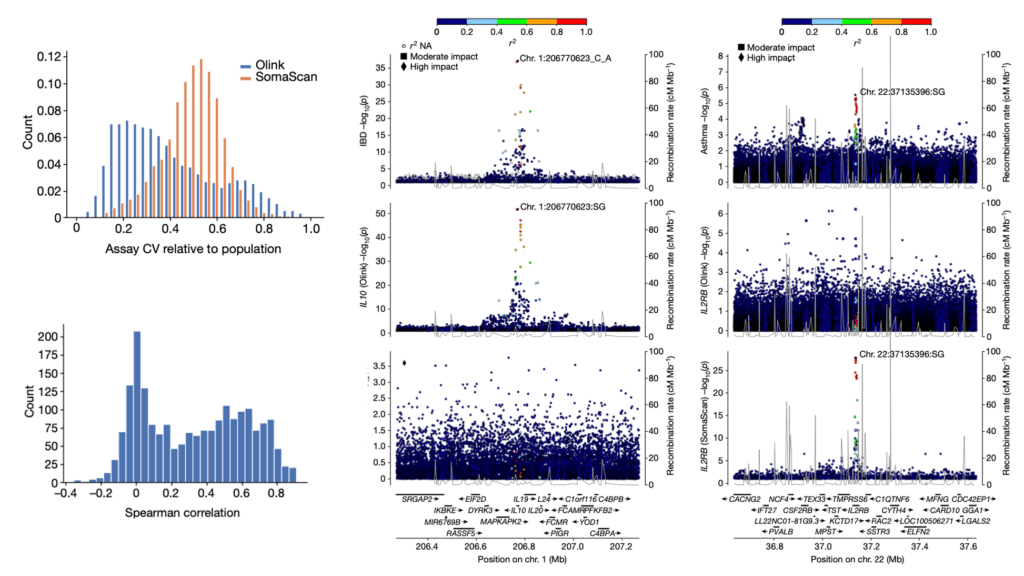
Fig. 1. 개별 검사로 측정된 단백질 레벨
– 좌측 그림은 플랫폼 별로 측정된 재현성을 의미합니다. 두 assay에서 반복 측정된 coefficient of variation (CV)를 무작위 측정의 CV로 나눈 비율인, assay CV 비율이 검사의 정밀도를 평가하는 데 사용되었습니다.
– 우측 그림은 Olink Explore 3072 및 SomaScan v4 플랫폼에서 측정된 단백질 수준의 측정값 간의 상관관계를 나타냅니다.
– 이 그림은 Olink와 SomaScan 플랫폼 간의 단백질 측정의 정밀도(반복성)와 상관관계를 보여줍니다. Olink는 더 나은 정밀도를 보여주며(CV 값이 낮다),두 플랫폼의 측정 값 사이에 moderate한 상관 관계가 있습니다.
– CV ratio가 0인 경우 반복 측정에서 항상 동일한 결과값을 주는 것을 의미하며 1일 경우 반복측정 결과의 변동성이 random하게 측정된 값들의 변동성과 동일함을 의미합니다.
– 좌측 그림의 경우 Olink Explore 3072 검사의 중앙값 CV 비율(파란색)은 SomaScan v4 검사(주황색)의 중앙값 CV 비율보다 낮았습니다. Olink Explore 검사는 UKB 47K 데이터 세트에서 1,474개의 반복 측정 데이터로 평가하였으며, SomaScan v4 검사의 경우 아이슬란드 36K 데이터 세트에서 419개의 반복 측정 데이터로 평가되었습니다.
– 우측 그림의 경우 Iceland 1K 데이터셋에서 (Spearman 상관관계를 사용하여) 평가되었으며, 두 플랫폼을 모두 사용한 1,514명의 개인의 혈장 샘플을 측정함으로써 평가되었습니다.
Ancestry group에 의한 pQTL 분석
– ancestry group기반 LD 정보를 활용하여 pQTL 분석을 했을 때의 결과
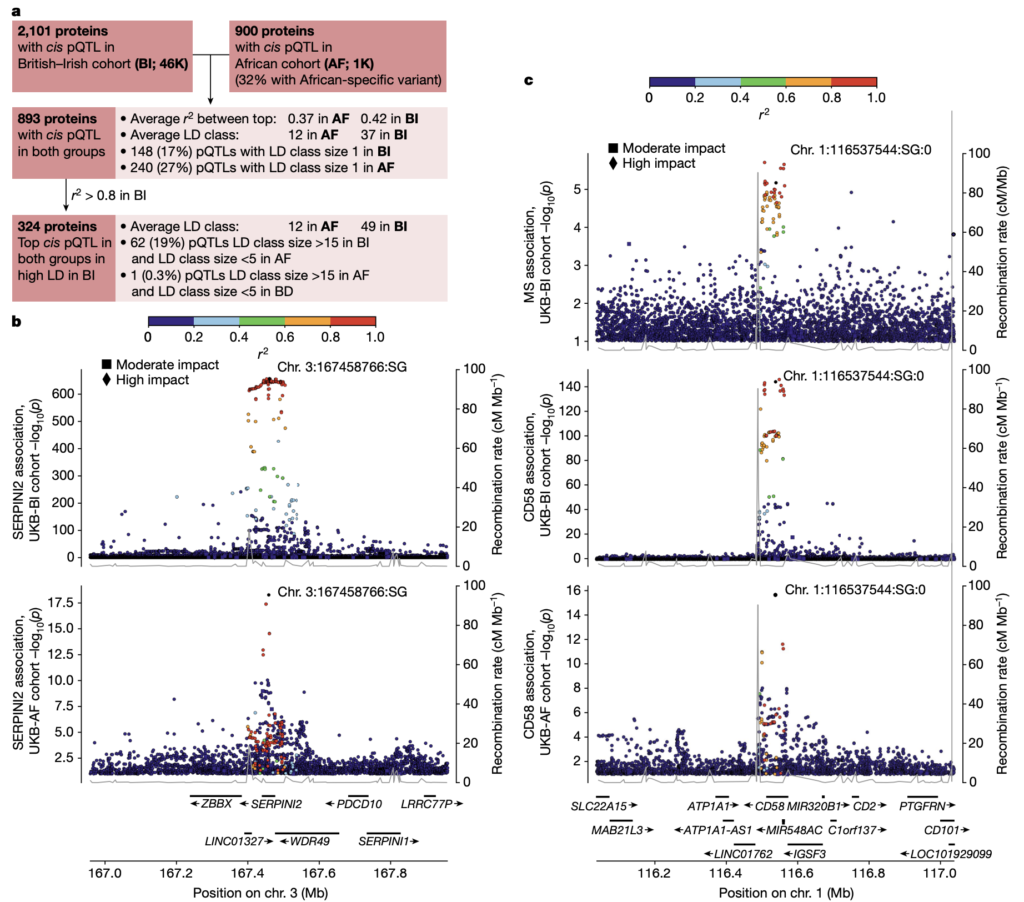
Fig. 2. 다른 ancestry 그룹을 사용하여 유전자 locus의 개선
(A) UKB-BI 데이터셋에서 찾아낸 pQTL의 locus를 개선하기 위해 UKB-AF pQTLs의 더 세밀한 LD 구조의 사용 과정
(B) UKB-BI (상단) 및 UKB-AF (하단) ancestry 그룹에 대한 sentinel cis pQTL의 locus plot입니다.
(C) CD58 위치에서의 다발성 경화증 (MS)과의 관련성에 대한 locus 그래프 (상단), 그리고 UKB-BI (중간) 및 UKB-AF (하단)에서의 CD58에 대한 sentinel cis pQTLs.
[Fig 2B] SERPINI2의 sentinel cis pQTL은 UKB-AF와 UKB-BI 그룹에서 동일하지만, 변이가 속한 LD 클래스는 UKB-AF 그룹에서 훨씬 작습니다. 이로 인해 어떤 변이가 실제로 단백질 수준에 영향을 미치는지 더 정확하게 결정할 수 있습니다.
[Fig 2C] UKB-AF 그룹에서 더 작은 LD class에 의한 locus 개선은 질병 관련성 또한 유사하게 개선될 수 있음을 보여줍니다.[Fig 2D] memory CG-DMR의 경우, primed reprogramming에서 minor transient demethylation (mCG/CG <0.1) 발생했으며, 이는 global CG methylation change (Fig.1b)와 일치.
- LD 구조를 사용하여 pQTL을 개선한다는 것은 넓었던 pQTL region을 좁게 만드는 것을 의미합니다. ancestry를 고려한 LD 구조를 사용했을 때 plot (b)와 (c)에서 locus가 좁아지는 것을 볼 수 있습니다.
플랫폼간의 pQTL 관계
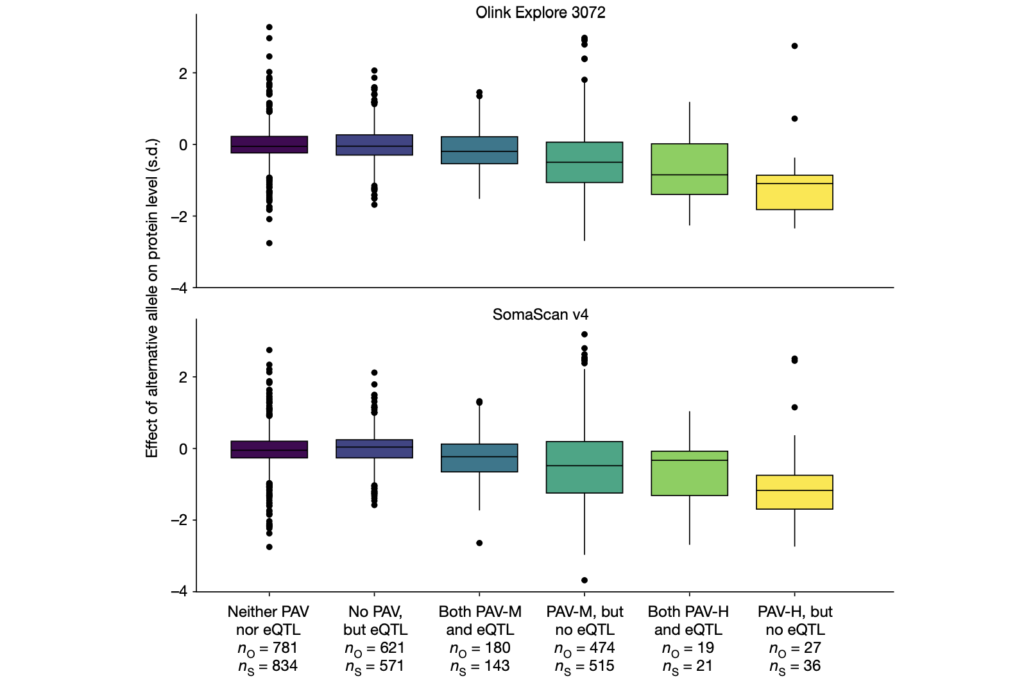
Fig. 3. 단백질 발현에 대립 유전자의 효과
- 플랫폼 별 단백질 발현에 대한 대립유전자의 효과를 높은 LD에서 Protein-Altering Variants(PAVs), cis eQTLs의 유무에 따라 나누었습니다.
- PAV-M은 중간 영향을 가진 PAV를, PAV-H는 높은 영향을 가진 PAV를 나타냅니다; nO는 Olink로 감지된 cis pQTLs의 수를, nS는 SomaScan으로 감지된 cis pQTLs의 수를 나타냅니다.
pQTLs 과 질병 연관 변이

Fig. 4. 한 플랫폼에서만 감지된 pQTLs와 질병 관련 변이와의 관계
– 좌측 그림, 시퀀스 변이와 IBD (상단) 그리고 Olink (중간)와 SomaScan (하단)을 사용하여 측정된 IL-10 레벨 간의 IL10 위치에서의 연관성.
– 우측 그림, 시퀀스 변이와 천식(상단) 그리고 Olink (중간)와 SomaScan (하단)을 사용하여 측정된 IL2RB 레벨 간의 IL2RB 위치에서의 연관성
- 두 플랫폼에서 검사 결과 IL-10 단백질의 경우 SomaScan 플랫폼에서는 cis pQTLs가 관찰되지 않았습니다. 반면에 IL2RB 단백질은 Olink 플랫폼에서는 cis pQTLs가 관찰되지 않았습니다.
Disscussion
이 연구는 두 개의 프로테오믹 플랫폼, 영국 바이오뱅크(UKB)에서 사용되는 Olink Explore 3072와 아이슬란드에서 사용되는 SomaScan v4를 비교합니다. 방대한 데이터셋에도 불구하고, 단백질 pQTLs 탐지에서 차이가 뚜렷하게 나타납니다. Olink는 더 많은 범위의 단백질에 대한 pQTLs를 식별하지만 혈장 측정에서의 어려움이 있습니다. 플랫폼 간 단백질 탐지 일관성을 기반으로 3단계 분류 체계가 개발되었습니다. 두 플랫폼에 공통으로 있는 단백질의 경우, Olink는 일반적으로 SomaScan보다 더 많은 cis pQTLs를 보여줍니다. 또한, 프로테오믹스에서의 ancestry의 영향은 중요하며, UKB 데이터를 통해 조상 특정 유전 변이와 단백질 수준 연관성을 밝혀냈습니다. 본 연구는 플랫폼 간의 불일치와 조상성과 같은 요인들이 고려한 상호 참조와 해석의 필요성을 강조합니다.
Olink와 SomaScan 플랫폼 모두 affinity-based 방식으로 작동하지만 Olink는 항체를 사용하고 SomaScan은 aptamers를 사용합니다. 이런 차이로 인해 혈장에서의 단백질 정량이 영향을 받을 수 있습니다. 이 연구는 주로 혈장 샘플에 중점을 둔 반면, 뇌척수액과 같은 다른 샘플 유형은 별도의 평가가 필요하며 다양한 샘플 유형에서 pQTLs를 탐색하면 귀중한 통찰력을 얻을 수 있습니다. 또한 비유럽계 인종의 샘플 크기가 제한적인 한계가 있으며, 이 크기를 늘리면 단백질 level과 유전 연관성에 대한 이해를 향상시킬 수 있다고 제안합니다. 플랫폼 간의 일부 불일치는 다른 프로테오폼에 대한 민감도로 인해 발생할 수 있지만 아직 연구되지 않았습니다. SomaScan 데이터의 SMP 정규화는 분석 결과에 큰 영향을 미치며, 이에 대한 추가 연구가 필요합니다. 또한 플랫폼은 에피톱 효과에 의해 다르게 영향을 받을 수 있으며, 결과가 일치하지 않을 때 직교 검증과 같은 추가 연구가 명확성을 위해 중요합니다.
두 플랫폼 모두 대상 단백질의 수를 확장하고 있으며, 한 플랫폼에서 혈장 내에서 cis pQTLs의 증거가 있는 단백질은 다른 플랫폼에서 측정 가능성이 가장 높은 단백질로 선택될 수 있음을 예측할 수 있습니다. 따라서 두 플랫폼 중 각각은 다른 플랫폼에서 문서화된 cis pQTLs를 가진 추가적인 1,000개의 단백질을 선별할 수 있습니다. 최대 500개의 단백질에 대해 두 플랫폼이 다른 프로테오폼을 측정할 수 있다는 증거 또한 있습니다. 우리는 미래의 프로테오믹스 검사 버전이 단백질의 단일 버전을 대상으로 하는 것이 아니라 주어진 유전자에 의해 코딩된 특정 프로테오폼을 대상으로 할 것으로 예측합니다.
