박테리아의 DNA 글리코실라아제인 Brig1이 변형된 바이러스 DNA 염기를 제거하여 박테리오파지 T4의 복제를 억제함으로써 바이러스에 대항하는 새로운 방어 기작을 발견하였습니다.
Abstract
박테리아는 다양한 방어 시스템을 진화시키며 파지 포식에 적응해왔습니다. 비록 생물정보학 도구를 사용해 항파지 면역 유전자를 식별할 수 있지만, 새로운 시스템의 발견은 사용 가능한 원핵 생물의 시퀀스 데이터에 제한되어있습니다. 이 제약을 극복하기 위해, 우리는 토양 메타게놈 DNA 라이브러리를 포함하는 대장균을 lytic coliphage (용해성 대장균 분해 바이러스) T4로 감염시켜 보호 유전자를 가진 클론을 분리했습니다. 이러한 방법을 통해, 우리는 Brig1이라는 DNA 글리코실라아제 (glycosylase)를 발견했는데, 이는 박테리오파지 T4의 게놈에서 α-글루코실-하이드록시메틸시토신 염기를 제거하여 무염기 부위를 생성하고 바이러스 복제를 억제합니다. T-even* 파지에 대한 면역을 제공하는 Brig1의 유사체들은 다양한 박테리아 분류군의 여러 파지 방어 지점에서 발견됩니다. 본 연구는 박테리아와 파지 사이의 상호 작용의 새로운 측면을 밝히기 위해 시퀀싱되지 않은 DNA를 탐색하는 것의 중요성을 강조하며, Brig1은 바이러스 포식에 대한 박테리아 방어에서 중요한 구성요소임을 시사하고 있습니다.
*T-even(짝수) phage: T2, T4 그리고 T6는 대장균 박테리아를 감염시키는 박테리오파지 그룹을 의미함.
Figures
Identification and functional analysis of Brig1 in E.coli conferring resistance against bacteriophage T4 infection through eDNA library
– eDNA library의 스크리닝은 바이러스 복제를 방지하여 파지 T4감염으로부터 대장균을 보호하는 알려지지 않은 유전자를 밝혀냅니다.
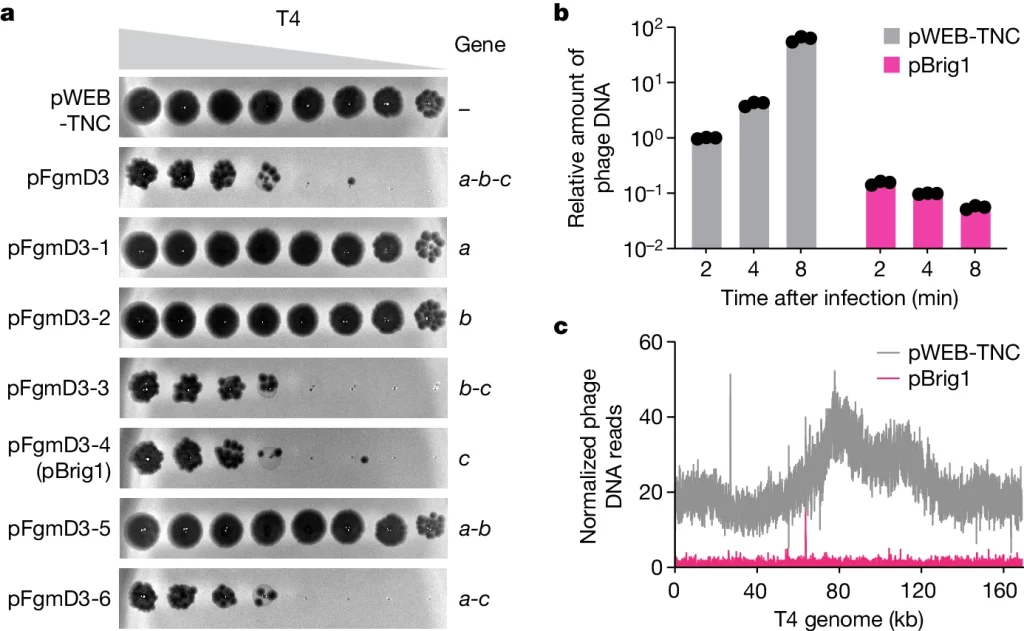
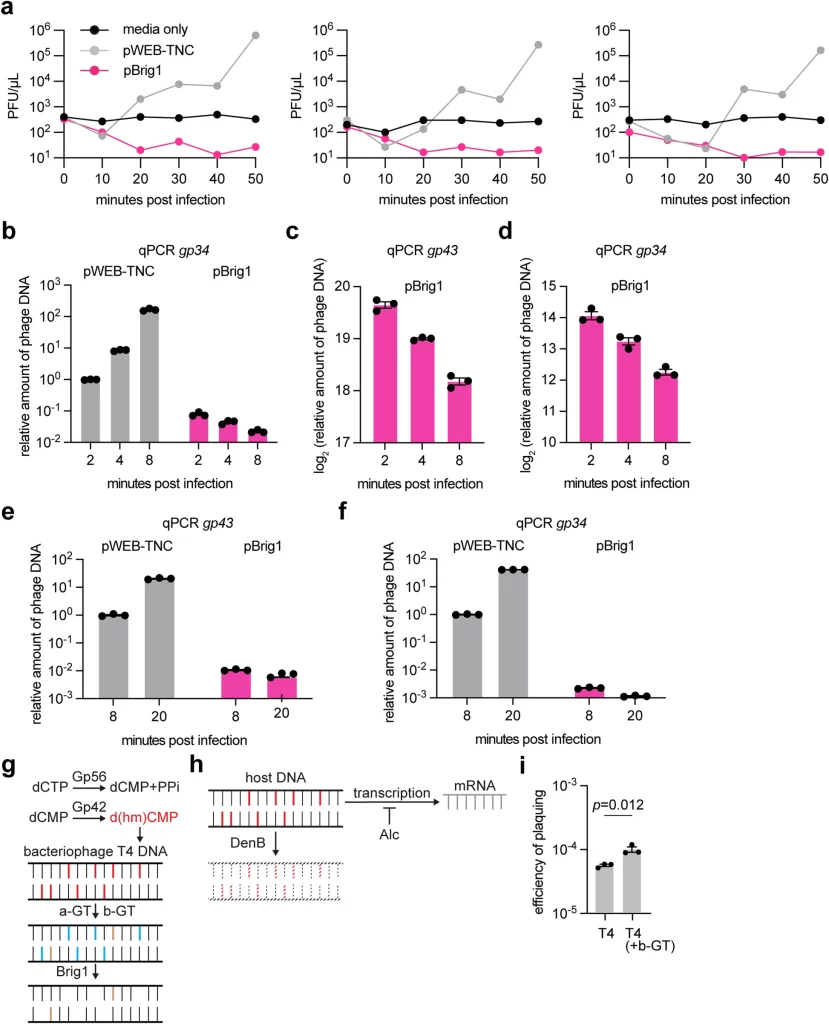
Fig. 1.eDNA library의 스크리닝을 활용한 유전자 investigation.
(A) 파지 T4를 이용한 eDNA library 스크리닝 후에 분리된 세 개의 유전자가 포함된 오페론이 삽입된 pWEB-TNC 코스미드를 함유한 대장균 EC100에서 T4 파지의 10배 희석 배양 결과. 여기서 유전자 c는 Brig1을 인코딩함. 이 데이터는 세 번의 독립적 실험 결과를 대표함.
(B) 감염 이후 2분, 4분, 8분에 pWEB-TNC 또는 pBrig1을 포함하는 대장균 EC100으로부터 추출된 바이러스DNA를 이용하여 gp43 유전자의 정량 PCR 분석 결과. 감염 다중도(MOI)가 1일 때, pWEB-TNC 2분 시점 대비 변화된 값을 계산하였음. 이는 세 번의 독립적 실험의 평균값 ± 표준오차를 나타냄.
(C) 감염 다중도(MOI) 5에서 pWEB-TNC 또는 pBrig1을 포함하는 대장균 EC100에 감염시킨 후 8분에 얻은DNA를 이용한 T4 DNA의 차세대 시퀀싱 리드를 바이러스 게놈에 매핑한 결과.
[Fig 1A] 각 행은eDNA library와 여러 클론들을 나타냅니다. 플라크로 알려진 투명한 반점의 존재는 박테리오파지가 박테리아를 죽인 영역을 나타냄. pBrig1로 표시된 클론 pFgmD3-4는 대조군 pWEB-TNC에 비해 플라크가 적거나 전혀 없음을 보여pBrig1 유전자가 파지 T4에 대한 보호를 제공하여 박테리아를 죽이고 플라크를 형성하는 것을 방지함.
[Fig 1B] 이 그래프는 gp43 유전자 (T4 유전자)를 표적으로 하는 정량적 PCR에 의해 측정된 감염 후 시간에 따른 파지 T4 DNA의 상대적인 양을 보여줌. 대조군 플라스미드 (pWEB-TNC)를 운반하는 대장균 세포의 파지 DNA의 양을 pBrig1 플라스미드를 운반하는 세포와 비교함. 감염 후 다른 시점에서 pBrig1 샘플의 파지 DNA 수준이 현저히 낮다는 것은 Brig1이 파지 복제를 방해한다는 것을 시사하고 있음.
[Fig 1C] 차세대 염기서열 분석 판독치: 이 차트는 대조군(pWEB-TNC)과 pBrig1을 비교하여 T4 게놈에 mapping된 T4 파지 DNA의 염기서열 판독치를 표시함. pBrig1 샘플의 감소된 피크는 박테리오파지 게놈 전체의 영역에서 파지 DNA 판독치가 더 적음을 나타내며, 이는 Brig1이 파지 게놈 무결성 또는 복제를 방해한다는 결론을 뒷받침함.
The antiviral effects of the DNA glycosylase Brig1 shows that it can target α-glucosyl-hydroxymethylcytosine (α-glc-hmC) modified bases within the T4 genome to hinder viral replication.
– 서로 다른 T4돌연변이에 대한 다양한 반응은 Brig1의 방어 메커니즘의 특이성과 잠재적 한계를 설명하는데 도움이 됨.
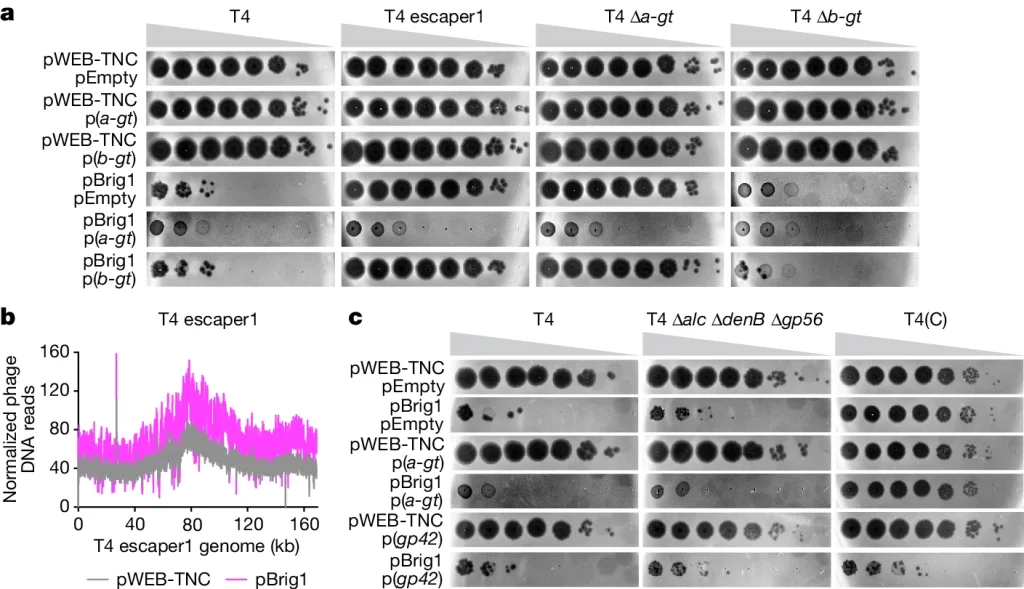
Fig. 2. Brig1의 방어 메커니즘.
(A) 각각 pWEB-TNC 또는 pBrig1 코스미드, 그리고 pEmpty, p(a-gt), p(b-gt) 플라스미드를 함유한 대장균EC100의 배양 평판에서 수행된 다양한 T4 파지의 10배 희석 연속 실험. 세 번의 독립적 실험 중 대표적인 한 실험의 플라크 형성 사진.
(B) 대장균 EC100이 pWEB-TNC 또는 pBrig1을 품고 있을 때, 감염 후 8분 만에 채취된 DNA로부터 얻은 T4 escaper1 DNA의 차세대 시퀀싱 리드가 바이러스 게놈에 정규화되어 매핑된 결과임. 이 실험은 감염의 다중도(MOI)가 5임을 확인함.
(C) 대장균 EC100의 평판에서 pWEB-TNC 또는 pBrig1 코스미드와 pEmpty, p(a-gt), 또는 p(gp42) 플라스미드를 갖는 각각의 평판에 대한 다른 T4 파지의 10배 희석한 연속실험. 세 개의 독립적인 실험에서 대표적인 한 실험의 플라크 이미지.
[Fig 2A] 행은 다른 플라스미드(pWEB-TNC, pEmpty)를 운반하는 대장균 (p(a-gt) 및 p(b-gt)와 같은 관심 유전자와 다른 파지 변종(T4, T4 escaper1, T4 δa-gt, T4 δb-gt)과의 상호작용을 포함하는 것을 나타냄. 플라크(맑은 원)는 파지가 박테리아를 용해시킨 영역을 나타냄. 결과는 야생형 T4에 의한 플라크 형성을 방지하는 pBrig1 유전자의 효과를 보여주지만 다른 파지 돌연변이에 대한 효과는 다양함을 보여줌.
[Fig 2B] 그래프는 파지 게놈에 매핑된 T4 escaper1 파지 DNA의 정규화된 시퀀싱 판독값을 보여줌. 대조군 플라스미드pWEB-TNC를 운반하는 대장균의 샘플과 pBrig1 플라스미드의 샘플을 비교하고 있음. 피크의 차이는 Brig1의 존재가 파지DNA의 복제 또는 안정성에 어떻게 영향을 미칠 수 있는지를 시사함.
[Fig 2C] a와 유사하게 다양한 플라스미드를 가진 E. coli EC100에 대한 T4 δalc, δdenB, δgp56 및 대조군(T4(C))을 포함한 다양한 파지 돌연변이체에 대한 플라크 분석을 보여줌. 이러한 결과는 pBrig1 유전자가 이러한 다양한 파지에 대한 E. coli의 민감도에 어떻게 영향을 미치는지 추가로 보여줌.
Brig1의 기능
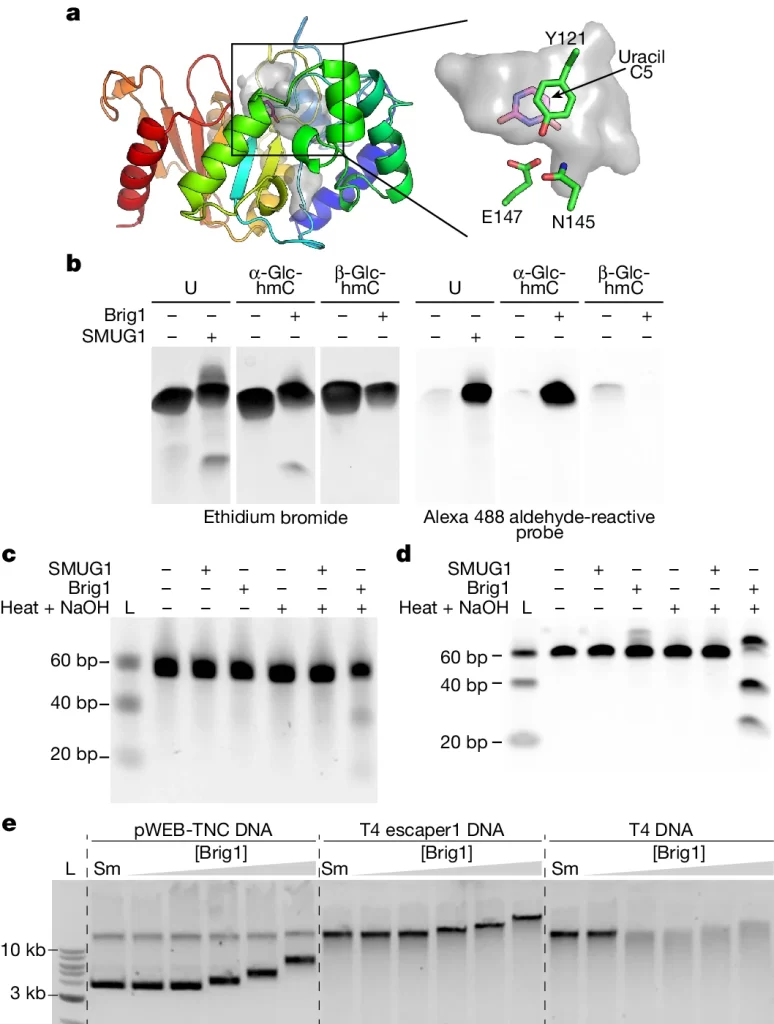
Fig. 3. Brig1은 α–글루코실-hmC 뉴클레오베이스를 절제합니다.
(A) Brig1의 AlphaFold2단백질 구조 시각화.
(B) 겔 전기영동 (Ethidium Bromide and Alexa 488염색)
(C) 이중가닥 DNA의 Urea-PAGE.
(D) 이중가닥 DNA의 Native PAGE.
(E) 아가로스 겔 전기영동.
[Fig 3A] N-말단(단백질의 시작)은 파란색으로 표시되고 C-말단(단백질의 끝)은 빨간색으로 표시. 단백질의 한 부분을 클로즈업하면 잠재적인 글리코실라제 포켓이 나타나는데, 여기서 DNA로부터 변형된 염기(α-글루코실-하이드록시메틸사이토신)가 적합하여 Brig1이 DNA와 어떻게 상호작용할 수 있는지 암시.
[Fig 3B] 이 겔은 변형된 염기(α-글루코실-hmC) 또는 규칙적인 염기(우라실, Uracil)가 있는 DNA에서 Brig1과 또 다른 글리코실라제인 SMUG1의 활성을 보여줌. 브롬산에티듐(Ethidium Bromide) 염색은 단일 가닥 DNA(ssDNA)의 존재를 드러내는 반면, 알렉사(Alexa) 488 알데히드 반응성 Probe는 염기를 DNA에서 제거하는 글리코실라제 활성의 결과인 염기 부위를 감지함.
[Fig 3C] 이 겔은 이중가닥 DNA로부터 변형된 염기를 절제하는 Brig1의 능력을 보여줌. DNA는 열과 수산화나트륨 처리 유무에 관계없이 Brig1 또는 SMUG1로 처리되어 염기성 부위에서 가닥이 끊어지는 것을 유도함.
[Fig 3D] 상부 및 하부 ssDNA 올리고뉴클레오티드가 모두 글리코실라제 활성을 받음을 확인한 후, 두 가닥 모두에 변형된 염기를 포함하는 DNA 이중체 분자를 테스트함. Brig1 처리는 β-제거 후 두 가닥 모두를 절단하여 비변성 native PAGE에 의해 분리될 수 있는 dsDNA break 및 절단 생성물을 생성함.
[Fig 3E] 이 겔은 파지 DNA (T4, T4 escaper1) 또는 대조군 DNA (pWEB-TNC)를 증가된 Brig1 농도로 처리한 결과를 보여줌. Brig1 농도가 더 높은 DNA 밴드의 도말 또는 강도 감소는 효소가 변형된 염기의 절제를 통해 파지 DNA에 손상을 입히고 있음을 시사함.
Experiments testing the immunity provided by the enzyme Brig1 against different bacteriophages and the effects on their DNA.
Brig1이 여러 박테리오파지에 대해 제공하는 면역력을 검증하고, 박테리오파지의 DNA에 끼치는 영향을 알아보기 위한 실험.
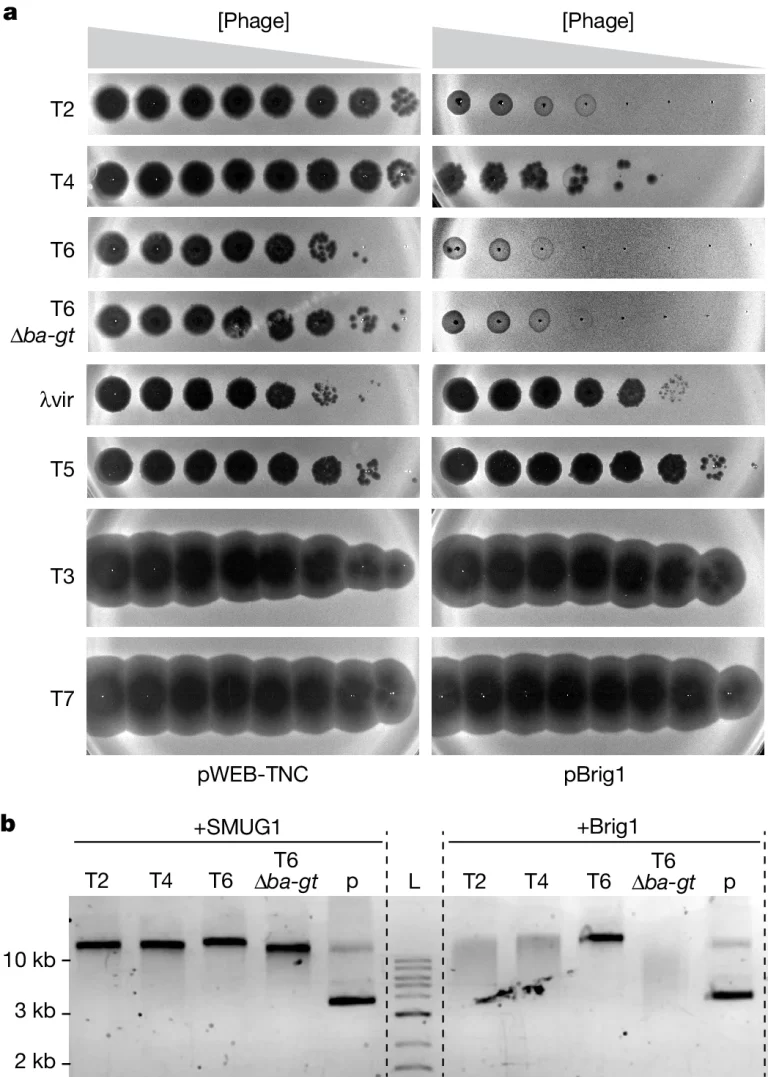
Fig. 4. Brig1은 α–글루코실-hmC 뉴클레오베이스를 운반하는 다양한 파지에 대한 면역을 제공합니다.
(A) Plaque Assays. 이미지는 E. coli EC100에 control cosmid pWEB-TNC 또는 pBrig1 cosmid를 위에뿌려진 다양한 박테리오파지의 10배 희석액을 보여줌.
(B) 아가로스 겔 전기영동. SMUG1또는 Brig1효소로 파지 DNA를 처리한 결과. DNA단편은 크기별로 분리되어 있으며 왼쪽의 size ladder는 DNA단편의 길이를 나타냄.
[Fig 4A] 각 박테리오파지 유형에 대해 왼쪽과 오른쪽 패널 간의 차이는 Brig1에 의해 제공된 보호 수준을 나타냄. 예를 들어, T4의 경우, Plaque가 오른쪽에서 적거나 없어서 Brig1이 T4에 대한 면역을 제공한다는 것을 시사함. 그러나 T3와 T7과 같은 박테리오파지의 경우, Brig1에 의해 제공되는 면역이 없음을 나타내 대조군과 뚜렷한 차이가 없음을 알 수 있음.
[Fig 4B] +SMUG1 처리와 +Brig1 처리 간의 밴드 패턴의 차이는 파지 DNA에 대한 이러한 효소의 다른 효과를 나타냅니다.예를 들어, T6 δ Ba-gt 밴드는 Brig1 처리 후 감소 또는 변경된 것으로 나타나, 이는 Brig1이 α-글루코실-하이드록시메틸사이토신(α-Glc-hmC) 뉴클레오베이스를 제거하여 DNA를 효소적으로 변형시켰음을 나타내며, 이는 Brig1의 활성과 일치함.
The antiviral effects of the DNA glycosylase Brig1 shows that it can target α-glucosyl-hydroxymethylcytosine (α-glc-hmC) modified bases within the T4 genome to hinder viral replication.
서로 다른 T4돌연변이에 대한 다양한 반응은 Brig1의 방어 메커니즘의 특이성과 잠재적 한계를 설명하는데 도움이 됩니다.
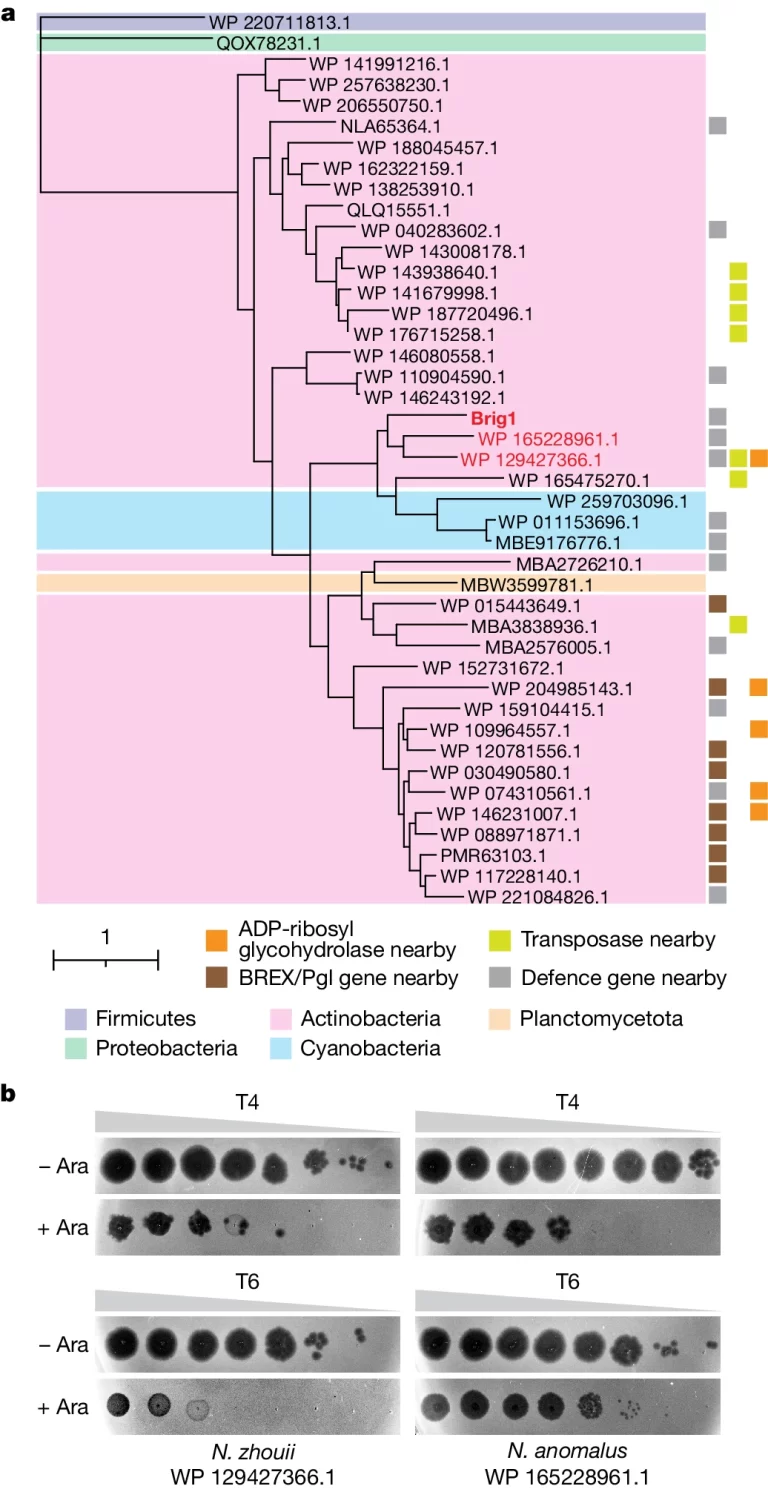
Fig. 5. Brig1 유사체는 항파지 방어기재를 지니고 있습니다 .
(A) Brig1유사체의 계통수. 다양한 세균 문(phylum)에 걸쳐 발견된 Brig1 유사체의 최대 우도(maximum likelihood) 계통 발생 트리를 보여줍니다. 각 세균 문은 색상 코드로 표시된 배경으로 나타냅니다 (예: 보라색으로 표시된 피르미쿠테스, 연두색으로 표시된 프로테오박테리아 등). T4 및 T6에 대한 항–박테리오파지 방어 기능을 제공하는 유사체는 빨간색으로 표시. 이 유사체들 근처에 방어 유전자, BREX/Pgl 유전자, 전위효소, ADP-리보실 글리코히드롤라제의 존재는 색상이 있는 사각형(각각 회색, 갈색, 노란색, 주황색으로)으로 표시. 이는 이들 유전자와 항–박테리오파지 방어 간의 가능한 연관성을 시사함.
(B) Plaque 분석결과: N. zhouii WP_129427366.1 또는 N. anomalus WP_165228961.1 Brig1 상동체를 포함하는 E. coli군에서 T4 및 T6 박테리오파지를 10배 연속 희석한 플라크 분석 결과. 이러한 분석은 아라비노스 유도(+Ara 및 -Ara) 유무에 따라 수행되었음. 아라비노스 유도(+Ara)가 있는 플라크가 적거나 없다는 것은 Brig1 유사체의 발현이 파지에 대한 방어기재를 부여한다는 것을 나타냄.
Disscussion
본 연구에서는 eDNA library를 사용하여 세균성 항박테리오파지 방어 체계를 찾기 위한 방법을 개발하였고, 유전체 데이터베이스에서 제공하는 정보를 넘어서 연구 범위를 넓혔습니다. 새로운 효소인 Brig1이 발견되었는데, 이는 특정한 DNA modification을 가진 박테리오파지에 대해 E. coli를 보호하는 역할을 합니다. Brig1은 DNA backbones을 절단하지 않고, 박테리오파지의 생명 주기를 방해하는 무염기 부위를 생성하는 특수한 DNA 글리코실라제로 보입니다. 또한, 다른 DNA modification을 가진 박테리오파지나 박테리아의 DNA 복구에는 영향을미치지 않음으로써, 특정한 항바이러스 도구로서의 역할을 합니다. Brig1은 BREX 및 CRISPR과 같은 유전자를 포함하는 더 큰 방어 체계의 일부로서, 박테리오파지에 대한 다층적인 방어를 제공합니다. 연구는 박테리오파지가 Brig1을 우회하여 진화할 가능성이 있으며, 이로 인해 지속적인 숙주–박테리오파지 간의 진화적 갈등이 발생할 수 있음을 시사합니다.