Rpd3S에 의한 H3K36me3-guided nucleosomal deacetylation의 diverse modes.
Abstract
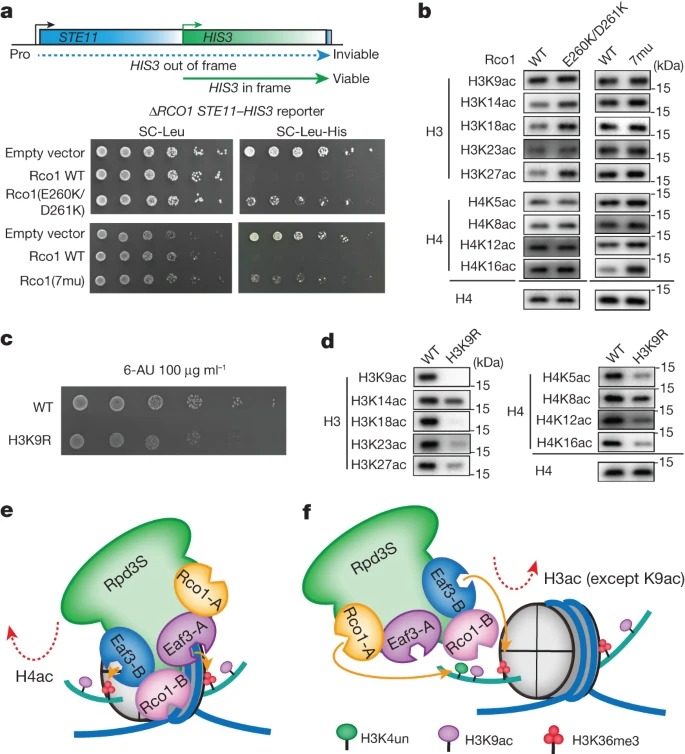
Context-dependent dynamic histone modifications은 gene regulation의 key epigenetic mechanism을 구성합니다. Rpd3 small (Rpd3S) complex는 lysine 36 (H3K36me3)의 histone H3 trimethylation를 인식하고 transcribed regions의 multiple sites에서 histone H3 및 H4를 deacetylates합니다.
여기서 우리는 Saccharomyces cerevisiae Rpd3S의 free 상태와 H3K36me3 nucleosome-bound states의 cryo-electron microscopy structures를 해결했습니다. 우리는 two copies의 Eaf3-Rco1 heterodimers가 Rpd3 및 Sin3와 asymmetrically 조립되어 catalytic core complex를 형성하는 Rpd3S의 unique architecture를 보여주었습니다. 두 개의 H3K36me3 marks, nucleosomal DNA 및 linker DNA를 Eaf3, Sin3 및 Rco1이 Multivalent recognition하면 Rpd3의 catalytic centre이 histone H4 N-terminal tail 옆에 위치하여 deacetylation이 이루어집니다. alternative catalytic mode에서, Rco1과 Eaf3에 의한 unmethylated histone H3 lysine 4 및 H3K36me3의 combinatorial readout는 등록된 histone H3 acetylated lysine 9를 제외한 histone H3-specific deacetylation을 지시합니다.
종합적으로, 우리의 연구는 multivalent nucleosomal engagement의 dynamic and diverse modes와 Rpd3S에 의한 methylation-guided deacetylation을 보여 주며, 전사 및 그 이상에서 섬세하게 설계된 multi-subunit enzymatic machineries를 통한 epigenetic regulation의 exquisite complexity를 강조합니다.