바이러스 capsid protein에 의한 박테리아 선천 면역계의 직접적인 활성화 과정
박테리아는 박테리오파지로부터 오는 지속적인 공격으로부터 스스로를 보호하기 위해 다양한 면역 메커니즘을 진화시켰습니다. 진핵생물의 선천 면역체계가 pathogen-associated molecular patterns(PAMPs)를 통해 외부로부터의 침입자를 감지하는 것과 유사하게, 박테리오파지 감염에 반응하는 세균의 수많은 면역체계는 파지에 대한 특이적인 트리거가 활성화되어야 합니다. 그러나 이러한 트리거와 감지 메커니즘의 정체는 여전히 거의 알려지지 않은 상태로 남아 있습니다.
이 논문에서는 다양한 파지로부터 Escherichia coli을 보호하는 융합 toxin–antitoxin system인 CapRelSJ46의 파지 대항 기능을 식별하고 조사합니다. 유전학적, 생화학적, 구조적 분석을 사용하여 CapRelSJ46의 C-terminal 도메인이 독성인 N-termial 영역을 침범하여 항독소 및 파지 감염 센서 역할을 한다는 것을 입증합니다. 특정 파지에 의한 감염 후, 새로 합성된 주요 capsid protein은 CapRelSJ46의 C 말단 도메인에 직접 결합하여 자가 억제를 완화하고 독소 도메인이 pyrophosphorylate tRNAs로 전환되도록 하여 바이러스 감염을 제한합니다. 종합적으로, 이들의 결과는 박테리아 면역 시스템이 보존된 파지의 필수 구성 요소를 직접 감지하는 분자 메커니즘을 보여주며, 박테리아 내의 toxin–antitoxin-mediated innate immunity에 대한 PAMP-like sensing model을 제시합니다. 이들은 CapRels 와 그들의 파지 방출 트리거가 ‘Red Queen conflict’에 관여하고 있다는 증거를 제공하여, 파지와 박테리아 간의 치열한 공진화(coevolutionary) 전투에서 새로운 전선을 드러냅니다. 일부 진핵생물 바이러스의 capsid protein이 포유류 숙주에서 선천적인 면역 신호 전달을 자극하는 것으로 알려져 있다는 점을 고려할 때, 이들의 결과는 깊숙이 보존된 면역의 측면을 보여줍니다.
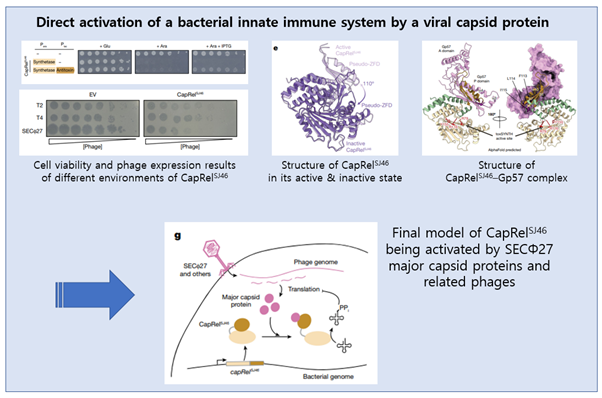
[Figure 1] 융합된 CapRel homologues는 E. coli에게 파지에 대한 강력한 방어를 제공할 수 있는 toxin–antitoxin 시스템입니다.
(a) long RelA-SpoT homologues (RSH), SAS, toxSAS 및 CapRelSJ46를 포함한 toxSAS toxin–antitoxin system의 융합된 하위 클래스의 구성입니다.
(b) CapRelSJ46 단독 또는 C-terminal antitoxin domain으로 N-terminal toxin domain을 발현하는 균주에 대한 연속 희석 실험에 의해 평가된 세포 생존성을 보여줍니다. (Ara, arabinose; glu, glucose; Para, arabinose-inducible, glucose-repressible promoter; Plac, IPTG-inducible promoter)
(c) CapRelSJ46, CapRelEbc 또는 CapRelKp을 생성하는 세포를 감염시킬 때 표시된 파지가 있는지에 대한 EOP data입니다.
(d) CapRelSJ46 또는 빈 벡터(EV)를 발현하는 플라스미드가 있는 세포에서 발견된 표시 파지의 연속적인 10 fold 희석 결과입니다. 상대적인 파지 농도는 쐐기의 높이로 표시되었습니다.
(e,f) 1차 감염 동안의 plaque-forming units(PFU)을 측정하는 1단계 성장 곡선입니다. 감염은 T4를 기본으로 (e) SECΦ27에 의한 감염, (f) CapRelSJ46 또는 빈 벡터 기반의 플라스미드를 보유한 세포 사이의 경향을 비교했습니다.
(g) CapRelEbc, CapRelEbc(Y153A) 또는 빈 벡터를 발현하는 플라스미드를 수용하는 세포들에서 발견된 T7 phage의 연속 희석 결과입니다.
(h) CapRelEbc, CapRelEbc(Y153A) 를 생산하거나 빈 벡터를 보유하는 세포들의 성장 데이터입니다. MOI = 10 & 0.001에서 T4 감염을 진행했습니다(데이터는 평균 ± s.d입니다. 8개의 플레이트 반복실험과 3개의 독립적인 실험을 대표합니다).
[Figure 2] CapRel의 유사 ZFD(pseudo-ZFD) 영역은 파지의 특이성의 원인입니다.
(a) CapRelSJ46 및 CapRelEbc의 sequence alignment 결과입니다. Alignment 결과에 차이가 가장 많은 영역인 pseudo-ZFD 영역이 표시되어 있습니다.
(b) 그림 상 표시된 CapRel constructs를 가지고 있는 세포들에서 지정된 파지들이 발견된 결과입니다. 연속 희석 실험 결과를 보이고 있습니다. (왼) CapRel을 어떻게 설계했는지의 모식도입니다.
(c) CapRelSJ46 의 3D 단백질 구조 그림입니다. 활성 영역인 G-loop Y155 및 ATP-coordination residues R79 & R116을 붉게 표시했습니다. 전체적인 구조의 요소들은 (a)에서의 sequence alignment를 따릅니다(toxSYNTH, pseudo-ZFD 및 anchors).
(d) CapRelSJ46 의 닫힌 형태를 AlphaFold를 통해 예측하였으며, (c)에서 표시된 색깔과 동일하게 영역을 표시했습니다.
(e) CapRelSJ46 의 활성화 지역(open, 연보라) 및 비활성화 지역(closed, 어두운 보라)을 단백질 결정 구조로 관찰 및 AlphaFold로 예측한 결과입니다. Superposition을 함께 제시합니다.
(f) 닫힌 상태의 CapRelSJ46 에서의 활성화 지역 중 자동으로 억제된 영역에 대한 세부 구조입니다. 이 구조에서는, YXXY neutralization motif가 adenine coordination site를 막으며, 효소 반응을 억제합니다.
[Figure 3] CapRelSJ46 은 SECΦ27의 major capsid protein으로 활성화되어, tRNA를 pyrophosphorylation하고 translation을 막습니다.
(a) SECΦ27로 감염된 His6–CapRelSJ46 의 immunoblot 결과입니다. Control은 감염되지 않은 세포입니다. Biological replicate를 대표하는 결과입니다.
(b) SECΦ27 escape mutant를 탐지하기 위한 실험에서의 진화적 접근을 모식도로 그렸습니다. 이러한 종류의 SECΦ27는 CapRelSJ46 를 보유하는 세포를 감염시킬 수 있습니다.
(c) 서로 독립적으로 진화시킨 5 종류의 SECΦ27 및 대조군의 연속적 희석 실험 결과입니다. 사용한 세포는 공벡터 보유군 및 CapRelSJ46 표현 벡터 보유군입니다.
(d) 확인된 escape mutants의 요약입니다. 모두 SECΦ27 위에 있는 gene 57의 hypothetical protein을 매핑하는 지역에 변이가 확인되었습니다. 각 escape mutant의 비율이 함께 표시되었습니다.
(e) Gp57의 AlphaFold 예측 구조이며, phage Hk97로부터 온 major capsid protein Gp5와 비교되었습니다.
(f) SECΦ27 phage lysate의 mass spectrometry 분석 결과입니다(wild type 및 Gp57(L114P)를 보유하는 변이종). 스펙트럼은 분자 질량을 기준으로 표준화되었으며, 개별 파지 단백질로 분류되어 보여졌습니다.
(g) CapRelSJ46를 표현하는 세포들 및 arabinose-inducible promoter에서 온 Gp57의 지정된 변이를 보유하는 세포들의 연속적 희석 결과입니다. 배지는 glucose 및 arabinose를 보유하는 환경을 가집니다.
(h) arabinose-inducible promoter 혹은 공벡터를 보유하는 CapRelSJ46 및 Gp57 (wild type (WT) 혹은 L114P)를 표현하는 세포들이 arabinose 첨가 후 35S-cysteine 및 35S-methionine으로 pulse-label되었습니다.
(i) (h)와 마찬가지 세팅이지만, CapRelSJ46 표현 벡터 혹은 공벡터를 보유하며, (위) SECΦ27 감염을 진행한 후의 결과, (아래) Gp57(L114P)를 표현하는 escape mutant의 결과를 보여줍니다. 별 표시는 P=0.022(45분) 혹은 P=0.004(60분)을 의미합니다. (unpaired two-tailed t-test 결과)
(j) DNA template 상 DHFR 생산물을 이용한 in vitro transcription-translation assay 결과입니다. Gp57 생산에 사용되는 template와 함께 정제된 CapRelSJ46를 추가했으며, biological replicate를 대표하는 결과입니다.
(k) CapRelSJ46를 정화한 반응의 autoradiography를 [γ-32P]ATP, bulk E. coli tRNAs 및 Gp57로 배양했습니다. Bult tRNA에 대한 SYBR Gold 염색을 loading control로 사용했습니다. Biological replicate를 대표하는 결과입니다.
[Figure 4] SECΦ27 major capsid protein Gp57는 CapRelSJ46의 pseudo-ZFD에 직접적으로 결합합니다.
(a) CapRelSJ46–Flag는 CapRelSJ46–Flag 및 GP57—HA를 표현하는 세포들로부터 immunoprecipitation(IP)되었습니다. 이후 지정된 Gp57 변이의 존재를 추적하기 위하여 HA tag를 사용하였습니다. IP에 이용한 용해물은 대조군으로 사용되어 발현량을 비교했습니다. 보여지는 그림들은 3가지의 biological replicate의 대표격입니다.
(b) CapRelSJ46 및 Gp57의 결합을 ITC로 관찰했습니다. (Kd, binding affinity; N, stoichiometry.)
(c) CapRelSJ46–Gp57 complex의 구조적 모델을 AlphaFold로 예측한 결과입니다. 모델에 따르면 Gp57의 P-domain(분홍)이 CapRelSJ46의 pseudo-ZFD(주황) 및 anchor region(초록)을 인식하는 것으로 파악됩니다. 이러한 상호작용은 pseudo-ZFD가 활성영역으로 꼬이는 것을 방지하고 효소를 활성화하는 데 기여합니다.
(d) CapRelSJ46 및 CapRelSJ46–Gp57 사이의 HDX 차이(ΔHDX)입니다. Heatmap으로 차이를 표현했습니다. 빨간색은 CapRelSJ46이 Gp57과 함께 존재할 때 중수소화의 비중이 증가하는 것을 나타내고, 파란색은 그 반대를 의미합니다. 회색 막대는 mass spectrometry 분석에서 파악된 펩타이드를 나타냅니다.
(e) CapRelSJ46의 위상학적인 표현이며, ΔHDX에 의해 색칠되었습니다. 효소의 활성 영역은 검정색 빗금으로 표현되었으며, catalytic toxSYNTH domain 및 phage-recognition pseudo-ZFD는 각각 연노랑 및 연주황으로 배경 처리 되었습니다.
(f) Glucose 혹은 arabinose를 보유하는 배지 상에서 CapRelSJ46의 지정된 변이를 보유하는 각 세포들의 연속적 희석 결과로, native promoter 유래군, arabinose-inducible promoter으로부터 온 wild-type Gp57 유래군으로 나뉩니다.
(g) 지정된 변이의 CapRelSJ46를 보유하거나 빈 벡터를 발현하는 세포들에서 발견된 SECΦ27 phage의 연속적 희석 결과입니다.
(h) (a)에서의 결과와 비슷한 선상으로, 지정된 CapRelSJ46–Flag의 변이를 포함한 결과입니다. 보여지는 그림들은 3가지의 biological replicate의 대표격입니다.
[Figure 5] CapRelSJ46, SECΦ27의 major capsid protein 및 관련 파지들의 공동 진화에 대한 증거
(a) Glucose 혹은 arabinose를 보유하는 배지에서 CapRelSJ46를 포함하는 세포들의 연속적 희석 결과입니다. 세포의 CapRelSJ46는 native promoter 혹은 arabinose-inducible promoter에 지정된 파지 유래 major capsid protein homologue에서 유래합니다.
(b) CapRelSJ46 혹은 공벡터를 표현하는 세포에서 발견된 파지들의 연속적 희석 결과입니다.
(c) wild-type Bas8 phage, 혹은 CapRelSJ46를 숨기고 있는 major capsid mutation의 escape mutants, 혹은 공벡터의 연속적 희석 결과입니다.
(d) SECΦ27의 major capsid protein을 코딩하는 시퀀스의 alignment 결과입니다. Bas5 및 Bas8은 CapRelSJ46를 트리거하며, 함께 있는 Bas4는 113번 자리에 phenylalanine 대신 tyrosine을 가집니다.
(e) Glucose 혹은 arabinose를 보유하는 배지에서 CapRelSJ46를 표현하는 세포들 중 native promoter 및 Bas4 혹은 arabinose-inducible promoter에서 지정된 SECΦ27 major capsid protein 변이의 연속적 희석 결과입니다.
(f) Wild-type Bas4 혹은 major capsid protein Gp8에 Y113F를 보유하는 두 가지의 변이 클론을 보유하는 세포의 CapRelSJ46 혹은 공벡터의 연속적 희석 결과입니다.
(g) CapRelSJ46이 SECΦ27의 major capsid protein 및 관련된 파지들로 인해 직접적인 활성화를 겪는 모델입니다. 게놈 주입 후, major capsid protein의 생산으로 인해 CapRelSJ46의 C-terminal antitoxin으로 인한 자동 억제 효과가 완화되었으며, 활성화된 CapRelSJ46을 통해 tRNA의 pyrophosphorylation(PPi)가 발생하며, translation 및 바이러스 감염을 억제합니다.
Conclusion
해당 연구는 SECΦ27에 의한 CapRelSJ46 활성화에 대한 모델을 제안합니다. 파지 감염이 없는 CapRelSJ46은 C-말단 항독소 도메인이 N-말단 독소 도메인을 자동으로 억제하는 세포에서 비활성적이고 폐쇄적인 형태를 가집니다. 반대로 바이러스 감염 시, 파지의 주요 capsid protein이 생성되고 활성화된 개방 상태를 안정화하기 위해 CapRelSJ46에 직접 결합합니다. 이러한 개방 상태는 CapRelSJ46이 tRNA를 pyrophosphorylation하고 translation을 억제할 수 있게 하며, 이는 mature virion 생성을 방해하여 세포 집단을 통한 파지의 전파를 막는 감염 감소 현상으로 이어질 가능성이 높습니다. 특히, 이들의 결과는 단백질 항독소를 특징으로 하는 type II toxin–antitoxin systems이 종종 주요 활성화 수단으로 주장되는 항독소의 단백질 분해 없이 활성화될 수 있음을 암시합니다. 이들의 연구는 융합된 type II toxin–antitoxin systems에 초점을 맞췄지만, 파지 단백질의 직접적인 결합에 의한 활성화는 표준적이고 비융합된 형태의 type II toxin–antitoxin systems에 대한 일반적인 메커니즘일 수도 있습니다. 위에서 언급한 바와 같이 항독소는 동족 독소보다 안정성이 떨어지지만, 회전율은 파지 감염에 반응할 만큼 충분히 빠르게 발생하지 않을 수 있는 반면, 파지 인코딩 트리거에 직접 결합하면 신속한 활성화가 가능할 수 있습니다.
SECΦ27의 Gp57과 같은 주요 capsid protein들은 toxin-antitoxin 시스템 및 기타 안티파지 방어 시스템의 일반적인 트리거가 될 수 있습니다. 선행 연구들은 T4의 주요 capsid protein Gp23 내에서 Gol이라는 짧은 펩타이드가 Gol과 Lit 단백질 가수분해효소가 모두 과잉 생산될 경우 EF-Tu를 절단하는 대장균의 Lit 단백질 가수분해효소를 활성화할 수 있다는 것을 발견했습니다. F 플라스미드가 T7을 배제할 수 있게 하는 PifA의 경우 주요 캡시드 단백질에 매핑된 돌연변이를 탈출시키지만, 이러한 상호작용은 생화학적으로 연구되지 않았습니다. 최근 연구에 따르면 T5의 주요 캡시드 단백질의 돌연변이는 Pycsar 매개 방어를 극복할 수 있게 하지만 capsid protein만으로는 Pycsar를 활성화하기에 충분하지 않습니다. 마지막으로, Pseudomonas aeruginosa의 주요 capsid protein을 코딩하는 유전자의 돌연변이는 CBASS 매개 방어로부터 탈출을 가능하게 하지만, capsid protein이 CBASS의 활성화제인지는 아직 알려지지 않았다. 그럼에도 불구하고, 우리는 주요 capsid protein이 다양한 범위의 안티파지 방어 시스템에 대한 일반적인 직접 트리거일 수 있다고 예상합니다. 예를 들어, 항바이러스성 STAND 방어 시스템은 최근 터미네이스 또는 포털 단백질에 의해 직접적으로 유발되는 것으로 밝혀졌습니다. 구조 단백질이 효과적인 트리거가 될 수 있지만, 파지와 박테리아의 강력한 공진화는 방어 시스템이 다른 트리거에 의존하도록 할 수도 있습니다.
진핵생물의 PAMP와 마찬가지로 활성화를 위해 필수적이고 풍부한 파지의 구성 요소에 의존하는 것은 감염 후에만 면역 반응이 장착되도록 하는 데 도움이 될 수 있습니다. 특히, 일부 진핵생물 바이러스의 capsid protein은 포유류의 선천적인 면역 경로를 자극합니다. 예를 들어, HIV capsid protein은 숙주 세포질과 핵에서 각각 TRIM5와 NONO에 의해 직접 검출되어 선천적인 면역 활성화를 유발합니다. 따라서, 이들의 결과는 병원체 탐지의 유사한 원칙이 박테리아와 진핵생물 모두에서 선천 면역의 기능과 분자 기반에 기초하고 있음을 시사합니다.