딥러닝을 이용한 luciferases의 De novo 디자인.
Abstract
De novo enzyme의 설계는 기하학적으로 호환되는 native scaffold에 관심의 반응을 촉매할 것으로 예측되는 active site와 substrate-binding pocket을 도입하고자 했지만, 적절한 단백질 구조의 부족과 native protein sequence–structure relationship의 complexity로 인해 제한되었다.
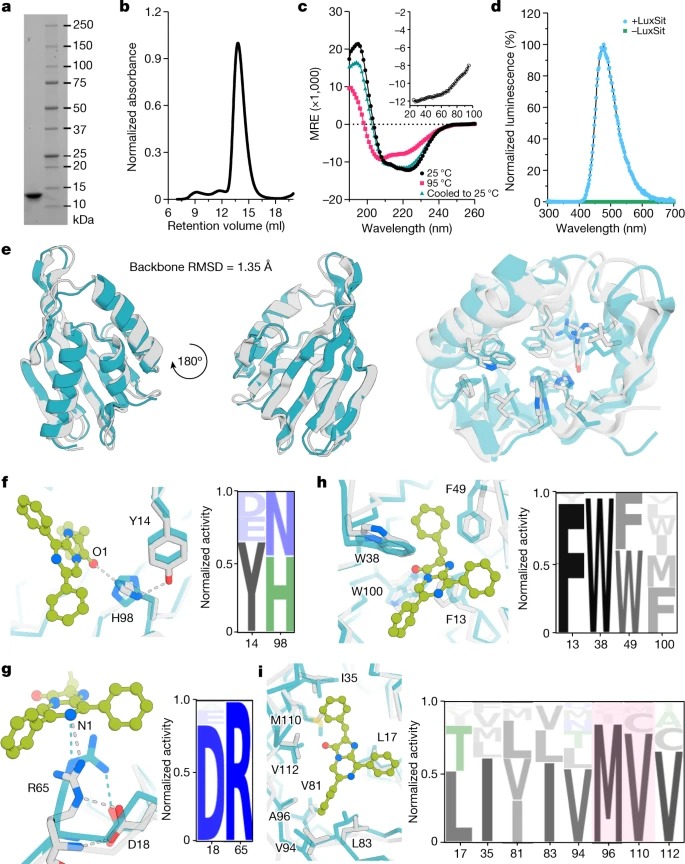
여기서는 다양한 주머니 모양과 이를 인코딩하는 설계된 시퀀스를 포함하는 idealized protein structure를 다수 생성하는 deep-learning-based ‘family-wide hallucination’ 접근 방식을 설명한다. 우리는 synthetic luciferin substrate인 diphenylterazine과 2-deoxycoelenterazine의 oxidative chemiluminescence을 선택적으로 촉매하는 artificial luciferase를 설계하기 위해 이러한 scaffold를 사용한다. 설계된 active sites는 반응 중에 발생하는 음이온에 인접한 arginine guanidinium group을 높은 형태의 상보성을 갖는 결합 포켓에 위치시킨다. 두 luciferin substrate 모두에서, 우리는 high selectivity을 가진 설계된 luciferases를 얻는다; 이들 중 가장 활동적인 것은 작고(13.9kDa) thermostable (녹는 온도가 95°C보다 높음) enzyme로 native luciferases와 비슷한 diphenylterazine에 대한 촉매 효율을 갖지만 substrate specificity은 훨씬 더 높다. 생물 의학에서 광범위한 응용을 통해 처음부터 매우 활성적이고 특정한 biocatalysts를 만드는 것은 계산 효소 설계의 핵심 이정표이며, 우리의 접근 방식은 광범위한 luciferases 및 다른 효소의 생성을 가능하게 해야 한다.
