Cooperative assembly로 regulatory specificity와 long-term genetic circuit stability 제공
Abstract
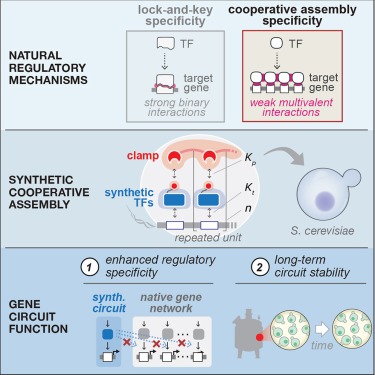
eukaryotic transcriptional regulation의 보편적인 특징은 transcription factors (TF)와 DNA cis-regulatory motifs 간의 cooperative self-assembly입니다. 이 전략은 상호 작용이 약하고 특이성이 낮은 분자 구성 요소 간의 유전자 네트워크에서 specific regulatory connections이 형성되도록 하는 것으로 생각됩니다.
여기에서는 yeast에 구축된 synthetic gene circuits을 사용하여 artificial zinc-finger-based TFs간의 cooperative, multivalent interactions에서 high regulatory specificity가 나타날 수 있음을 발견했습니다. 우리는 cooperative TF assembly strategy를 사용하여 “wired”된 circuits이 host cell genome의 aberrant misregulation으로부터 효과적으로 격리된다는 것을 보여줍니다. 실험과 수학적 모델에서 입증한 바와 같이, 이 메커니즘은 circuit-driven fitness defects을 복구하기에 충분하여 long-term continuous culture에서 circuits의 genetic and functional stability를 유지합니다.
자연에서 영감을 얻은 우리의 접근 방식은 다양한 host organisms과 applications로 확장할 수 있는 high-fidelity, evolutionarily robust gene circuits을 구축할 수 있는 간단하고 일반화 가능한 수단을 제공합니다.
Figure
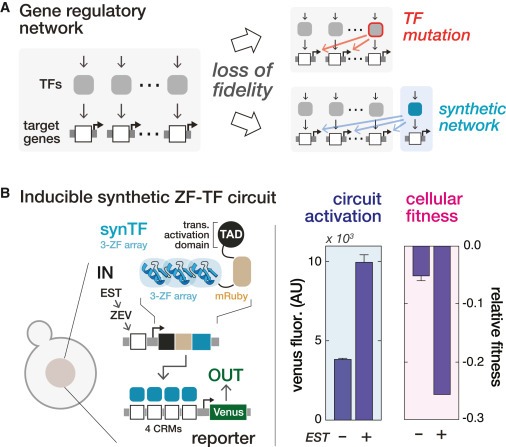
Figure 1. 유전자 네트워크는 적절한 cellular function을 위해 transcription factors와 target genes 간의 specific interactions에 의존합니다.
(A) transcription factor (TF) mutations 또는 synthetic circuits의 도입으로 인해 발생하는 Cross-reactivity는 genome-wide interaction fidelity를 떨어뜨리고 cellular function and fitness를 방해할 수 있습니다.
(B) 일반적인 종류의 artificial zinc-finger (ZF)-based synthetic transcription factors (synTF)로 구성된 synthetic gene circuits을 활성화하면 yeast에서 관찰 가능한 fitness defects가 발생합니다.
[Figure 1] TF는 aberrant misregulation을 초래할 수 있는 off-target interactions을 피하면서 높은 specificity를 가진 gene-associated CRM을 인식해야 함.
Cooperative TF assemblies reduce fitness cost burden while maintaining circuit output
– TF는 우리의 synTF와 유사한 molecular characteristics을 가지고 있기 때문에, 우리는 cooperative assembly에 의한 조절을 통합하는 circuits이 잠재적으로 fidelity가 향상되고 fitness defects가 감소된 synthetic circuits을 설계하는 데 사용될 수 있다는 가설 도출.
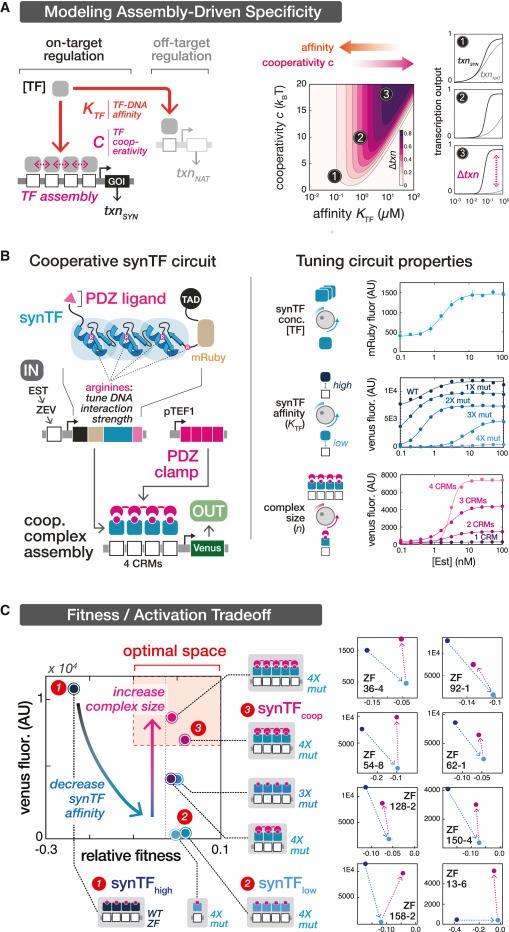
Figure 2. cooperative assembly를 사용하여 gene circuits을 구성하면 강력한 circuit output을 유지하면서 fitness defects를 최소화할 수 있습니다.
(A) 간단한 모델은 regulatory specificity를 설계하기 위해 cooperative assemblies를 사용하는 molecular strategy를 보여줍니다.
왼쪽: transcription factor (TF)가 on-target synthetic circuit (SYN) locus와 ff-target native (NAT) locus 모두에서 DNA와 상호작용할 수 있는 thermodynamic-based model입니다.
오른쪽: SYN 대 NAT locus의 transcriptional output으로 정의되는 regulatory specificity는 DNA에 대한 TF affinity가 낮아지고 TF cooperativity가 증가함에 따라 개선됩니다.
(B) cooperative synthetic transcription factor (synTF) assemblies로 구성된 circuits을 구성하기 위한 Experimental platform.
왼쪽: Inducible circuit architecture.
오른쪽: Tunable circuit properties.
(C) low-affinity synTF의 cooperative regulatory assemblies를 활용하는 circuits은 high circuit output을 유지하면서 fitness costs를 최소화하여 fitness-activation tradeoff를 최적화합니다.
왼쪽: 다양한 non-clamp and clamp circuit configurations에 대한 cellular fitness와 circuit activation 비교.
오른쪽: 서로 다른 결합 서열을 가진 서로 다른 ZF species로 구성된 circuit configurations의 fitness-activation measurements는 유사한 패턴을 보입니다.
[Figure 2A] cooperative synTF assemblies를 사용하여 매우 specific circuit connections을 구성하는 molecular strategies에 대한 통찰력을 얻기 위해 이전 연구를 확장하는 간단한 thermodynamic-based model을 구축.
[Figure 2A right] regulatory specificity score를 SYN(txnSYN) 및 NAT(txnNAT) loci에서의 transcriptional output 차이로 정의한 다음 이 점수를 [TF], KTF, c의 함수로 플롯(그림 2A 오른쪽).
[Figure 2B left] multiple covalently linked PDZ domains으로 구성된 synthetic protein으로, synTF의 peptide ligands와 상호작용하여 DNA에 결합하는 multivalent coordination을 가능하게 함.
[Figure 2B right] synTF expression level ([TF])은 EST를 추가하여 조정할 수 있는 반면, synTF에 대한 KTF는 DNA phosphate backbone과 non-specific interactions을 하는 ZF array의 conserved arginine residues 세트에 alanine mutations (WT-4X mut)를 도입하여 조정할 수 있음.
[Figure 2B] varying complex valency (n)를 변화시킴으로써 c를 조정할 수 있으며, 그 결과 dose response steepness의 가파른 정도를 변경할 수 있음.
[Figure 2C] mean fluorescence와 relative fitness를 2차원 “fitness-activation” phenotypic space에 플롯.
[Figure 2C] 그림 1B에서 테스트한 circuit configuration (high-affinity synTF를 포함하는 circuit, synTFhigh circuit이라고 함)은 reporter activation는 높았지만 cellular fitness는 낮아 공간의 왼쪽 상단 영역에 배치.
[Figure 2C] 우리 모델의 예측과 일치하게, 우리는 low-affinity synTF와 n = 4(synTFcoop circuit) 또는 5 CRM sites를 가진 clamp의 발현을 통해 circuit activation이 회복될 수 있으며, fitness의 명백한 손실은 거의 없다는 것을 발견.
- cooperative assemblies를 사용하여 synTF circuits을 배선하면 circuit function and host fitness를 모두 최적화할 수 있는 간단하고 확장 가능한 전략을 제공한다는 것을 보여줌.
Cooperative assembly is sufficient to rescue aberrant gene expression caused by synthetic circuits
– synTF circuit-imposed fitness costs의 차이가 실제로 host cell network misregulation의 결과인지 확인하기 위해, 우리는 RNA sequencing (RNA-seq)을 수행하여 synTFhigh, synTFflow 및 synTFcoop circuits를 유도한 후 host cell transcriptomics를 평가.
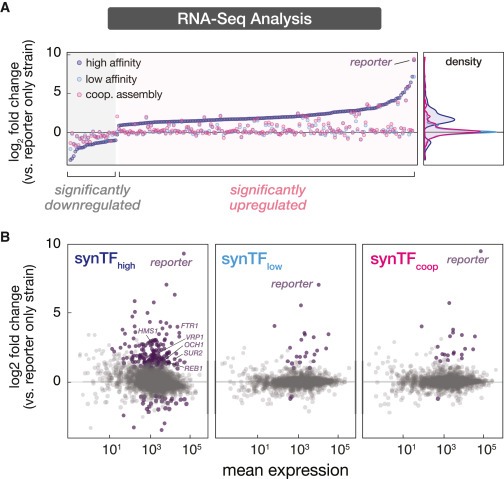
Figure 3. synthetic circuits로 인한 aberrant gene expression을 구출하는 Cooperative assembly.
(A) synTFhigh, synTFlow 및 synTFcoop circuits를 유도한 후 RNA-seq 측정의 Differential gene expression analysis.
(B) 모든 유전자에 대해 plotted된 synTFhigh, synTFlow 및 synTFcoop strains의 Differential gene expression profiles.
[Figure 3A] synTFhigh expression이 reporter control strain에 비해 host transcriptom을 광범위하게 잘못 조절하는 것을 발견.
[Figure 3B] synTF에 의한 TAD-dependent off-target gene activation의 일반적인 모델과 일치하여, 대부분의 misregulated genes은 upregulated되었으며 (182/211), 이러한 유전자는 transcription start site (TSS)의 upstream 300 bp window 내에 잠재적 인 synTF binding sites (CRM과 8/9 bp homology)를 보유 할 가능성이 더 큼.
[Figure 3A,B] synTFlow 및 synTFcoop 회로를 보유한 세포의 transcriptomes은 서로 유사하고 strain background과 유사한 expression profiles을 보여 native transcription에 대한 영향이 미미함을 보임.
[Figure 3B] 예상대로, clamp의 추가는 synTFlow의 경우와 비교하여 endogenous gene misregulation에 영향을 미치지 않는 것으로 나타남.
- 이러한 결과는 전체적으로 synTFhigh 회로에서 관찰된 성장 결함의 기초로서 transcriptional network misregulation을 암시하며, 이러한 defect는 synTFlow 및 synTFcoop 회로에서 tuned-down synTF-CRM interaction affinity를 조정하여 복구할 수 있음을 보여줌.
Synthetic cooperative assembly reduces off-target binding in the genome
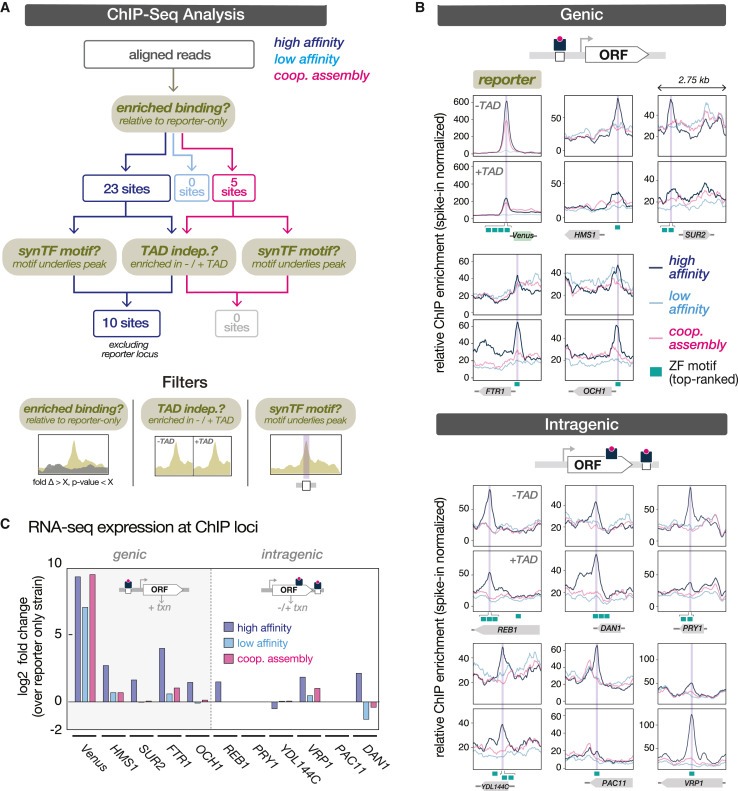
Figure 4. Synthetic cooperative assembly는 host genome에서 off-target binding을 줄입니다.
(A) synTF에 의한 genome-wide binding events를 식별하기 위한 ChIP-seq analysis pipeline.
(B) 10개의 nominated binding sites에서 synTF ChIP enrichment patterns, gene TSS의 upstream에 위치한 경우 genic, gene body 내에 위치한 경우 intragenic으로 분류.
(C) synTF ChIP binding event와 관련된 유전자의 RNA-seq differential expression.
[Figure 4A] synTFhigh의 23개 부위, synTFcoop의 5개 부위, synTFlow에서는 없는 부위에서 유의미한 synTF binding의 enrichment를 관찰.
[Figure 4A] 이 28개 부위가 잠재적인 off-target synTF misregulation을 매개할 수 있는지 평가하기 위해 두 가지 기준에 따라 부위를 필터링했습니다: (1) strains both with and without the TAD fusion 모두에서 해당 부위가 견고하고 잠재적인 pull-down artifact가 아닌지 여부, (2) quantitated 42-10 nuclease DNA cleavage sequence specificities에 대한 독립적인 데이터 세트에 의해 결정된 alignment peak (700 bp 이내)와 putative synTF CRM의 근접성. 동일한 10개의 부위가 두 기준 모두에 의해 단독으로 독립적으로 분리되었으며 synTFhigh strain에서 나온 것으로 나타났습니다
[Figure 4B] 대부분의 경우, 이러한 부위에는 ChIP peak maxima 바로 아래에 있는 모티프가 포함되어 있었으며, 일부 부위는 피크 아래에 여러 모티프가 모여있음.
[Figure 4B] 세 가지 circuit strains (positive, negative, no effect) 각각에 대해 reporter와 ChIP-nominated genes에 대한 RNA-seq-measured expression changes를 플롯한 결과, synTFhigh strain에서 genic binding events 와 관련된 모든 유전자의 transcription이 대조군에 비해 상향 조절된 반면, genic binding events와 관련된 유전자는 다양한 조절을 보이는 것을 발견.
- 전체적으로 이러한 결과는 synTFhigh 회로를 보유한 strains에서 host cell transcriptional misregulation의 원인으로 off-target synTF binding을 강력하게 암시하며, 이 효과는 synTFcoop 회로에서 cooperative synTF assembly에 의해 최소화.
Cooperative synTF regulatory linkages enhance long-term genetic circuit stability
– cooperative synTF assemblies를 사용하여 transcriptional fidelity의 손실과 circuit expression과 관련된 fitness cost를 완화할 수 있다는 사실에 착안하여, 이 전략이 continuously growing cultures에서도 long-term circuit stability를 부여할 수 있는지 조사.
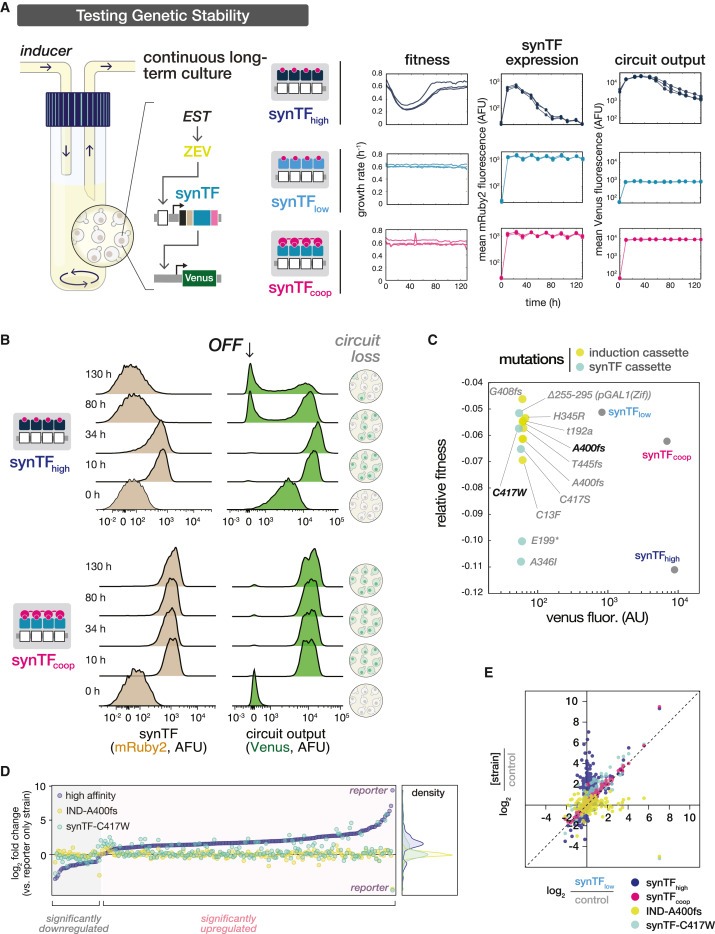
Figure 5[A,B]. Cooperative regulatory linkages로 synthetic circuits의 long-term genetic stability 향상.
(A) cellular fitness를 실시간으로 측정하는 automated continuous culture system인 eVOLVER에서 synTF circuits의 long-term stability 테스트.
(B) continuous culture experiment의 시간 경과에 따른 synTF 및 circuit reporter expression의 Single-cell flow cytometry distributions.
[Figure 5A] 최근 개발한 customizable, automated bioreactor platform인 eVOLVER를 활용하여 reporter-only control과 함께 synTFhigh, synTFlow, synTFcoop 회로 설계를 발현하는 strains를 5-day continuous culture
[Figure 5A] 회로 유도 후, synTFhigh 균주의 성장 속도가 급격히 감소한 후 ∼40시간에 걸쳐 느린 회복 단계가 나타나는 것을 관찰.
[Figure 5B top] 시간 경과에 따른 Single-cell fluorescence-activated cell sorting (FACS) 분포를 보면 점차적으로 증가하는 “circuit-off” subpopulation이 나타나며, 이는 집단 전체에서 synTF-mRuby 발현의 손실과 동시에 나타남.
[Figure 5B bottom] synTFflow 및 synTFcoop 회로를 접종한 cultures는 유도 후 및 실험 기간 내내 control strain과 유사한 성장률을 유지했으며, 유도 후 정상 상태에 도달한 후에도 synTF 농도와 reporter activation이 변하지 않고 뚜렷하게 정의된 “circuit-on” 집단을 유지.
Adaptive circuit-breaking mutations target synTF expression and function
– synTFhigh 및 synTFcoop cultures에서 추출한 50개의 개별 콜로니의 endpoint genotypes을 분석하여 loss of circuit function에 대한 mutational paths를 실험적으로 평가.

Figure 5[C,D,E]. Cooperative regulatory linkages로 synthetic circuits의 long-term genetic stability 향상.
(C) eVOLVER continuous culture experiment에서 선택된 circuit genotype mutations Characterizing.
(D) Adaptive circuit-breaking mutations는 synTFhigh circuit에 의해 유도된 gene misregulation pattern rescue. synTFhigh circuit와 두 가지 mutant genotypes에 대한 RNA-seq differential gene expression analysis: IND-A400fs(induction cassette) 및 synTF-C417W(synTF cassette).
(E) 다양한 circuit genotypes에 대한 transcriptomes와 synTFlow circuit genotype의 상관관계.
[Figure 5C] synTFcoop-derived colonies의 circuit genotype에서는 돌연변이가 관찰되지 않았지만, induction cassette (10개)와 synTF cassette (5개) 내에서 돌연변이가 발생한 총 27개의 돌연변이 synTFhigh-derived colonies가 발견.
[Figure 5C] 모든 돌연변이는 circuit output을 비활성화하는 것으로 나타났으며, 두 개를 제외한 모든 돌연변이는 체력을 대조군 수준으로 회복.
[Figure 5D] 두 돌연변이 [induction cassette (IND-A400fs)의 A400fs와 synTF cassette (synTF-C417W)의 C417W] 중 하나만으로도 synTFhigh에 의해 유도된 gene misregulation 패턴을 대부분 복구할 수 있음을 발견.
[Figure 5E] 흥미롭게도 IND-A400fs 돌연변이의 transcriptomic profile은 reporter-only control에 비해 유의미한 gene misregulation이 나타나지 않은 반면, synTF-C417W mutant circuit의 프로파일은 synTFlow 및 synTFcoop strain과 유사성을 보임.
- 이러한 결과는 synTFhigh circuit instability의 기초로서 functional synTF expression을 강화하고, 나아가 synTF affinity를 낮춤으로써 이러한 fitness defect를 구제하는 우리의 엔지니어링 전략이 adaptive circuit-breaking mutations의 growth phenotype을 요약한다는 것을 나타냄
Cooperative assembly mediated positive feedback circuits for stable long-term activation memory in continuous culture
– positive feedback circuits을 설계하기 위해 기존의 2-node inducible circuit 설계를 3 node로 확장
– 이 circuits의 두 synTF species는 모두 affinity가 낮으며, autoregulatory and reporter nodes의 complexes는 모두 clamp binding에 의해 매개되며, 이는 genetic stability를 가능하게 할 것이라고 가설을 세움.
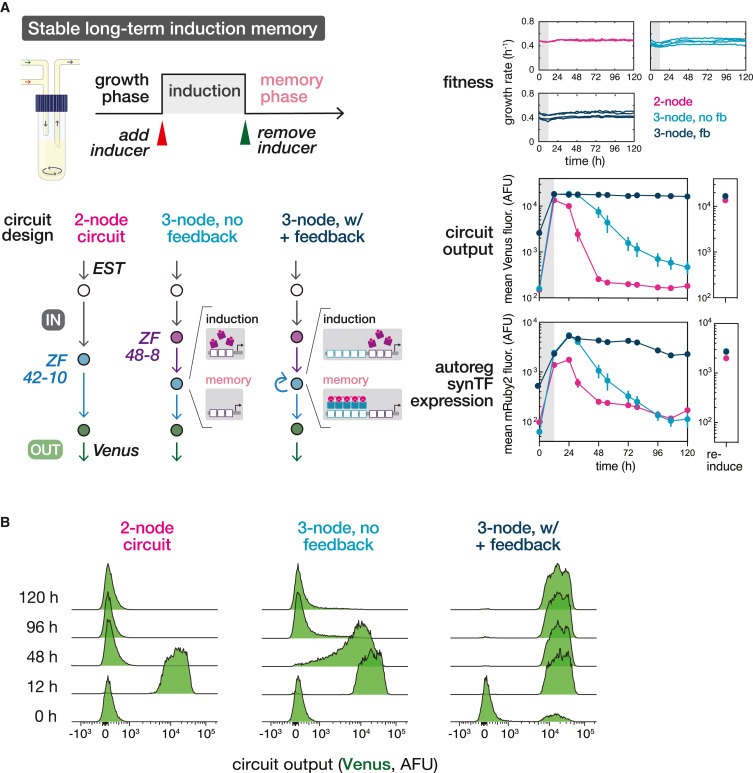
Figure 6. long-term continuous culture에서 안정적인 activation memory를 위한 engineer positive feedback circuits를 위한 cooperative assembly 사용.
(A) transient induction 후 eVOLVER에서 synTF circuits의 activation memory 테스트.
(B) continuous culture experiment의 시간 경과에 따른 circuit reporter expression의 Single-cell flow cytometry distributions.
[Figure 6A] 3-node positive feedback circuit를 no-feedback control 및 기존 two-node circuit과 비교하여 continuously growing strains에 12시간 동안 inducer (100nm β-estradiol)를 추가한 다음 나머지 시간 동안 uninduced growth media로 다시 전환.
[Figure 6A] 중요한 것은 모든 3-node circuit 설계(feedback and non-feedback controls 모두)가 시간 경과 전반에 걸쳐 일관되고 높은 성장률을 유지하여 genetic stability가 유지되는 것을 관찰.
[Figure 6A,B] 예상대로 positive feedback circuit은 non-feedback circuits에 비해 강력한 activation memory를 보였으며, 이는 inducer removal 후 빠르게 감소.
– 전체적으로 이러한 결과는 cooperative assembly mediated feedback circuits이 바이오 생산 및 기타 생명 공학 요구 사항을 해결할 수 있는 circuit function에 대한 강력하고 조정 가능한 활성화를 가능하게 한다는 것을 보여줍니다. 또한, high regulatory specificity, fitness, and long-term genetic stability를 갖춘 more complex circuits를 생성할 수 있는 프레임워크의 확장성을 검증.
Disscussion
이 연구에서는 놀랍도록 low-information content를 가진 natural TF CRM이 널리 퍼져 있음에도 불구하고 gene regulatory networks에서 어떻게 정교한 regulatory specificity가 달성되는지에 대한 근본적인 질문을 조사했습니다. individually weakly and non-specifically interact하는 TF regulatory proteins 간의 cooperative interactions을 통해 high specificity가 나타난다는 것을 발견했습니다. 또한, cooperative TF assemblies가 gene circuits에서 specific regulatory connections을 설계하는 데 사용될 수 있으며, 이는 circuit performance를 향상시키고 eukaryotic cells에서 circuit-imposed fitness costs를 최소화할 수 있는 수단을 제공한다는 것을 보여줍니다. 종합적으로, 이 연구는 자연에서 영감을 얻은 전략을 활용하여 synthetic circuits를 host regulatory networks와의 cross-talk로부터 효과적으로 차단함으로써 evolutionary pressures에 대해 향상된 안정성을 갖춘 circuits를 빠르게 개발할 수 있다는 것을 보여줍니다(그림 7).
이 연구에서 우리는 off-target genomic binding으로 인한 transcriptional misregulation이 또 다른 종류의 fitness-reducing circuit-host interaction을 구성하며, 이는 genomic complexity로 인해 eukaryotic host cells에서 circuit function을 방해하는 주요 원인이 될 수 있다는 증거를 제시합니다(그림 7B). RNA-seq 및 ChIP-seq 실험의 결과는 synTF circuit으로 인한 host transcription의 잘못된 조절이 primarily, but not exclusively, native gene transcriptional initiation에 인접한 게놈 CRM의 일부 하위 집합과의 상호 작용의 결과일 가능성이 높다는 증거를 제시합니다.
Limitations of the study
cooperative assembly scheme의 일반화 가능성에도 불구하고, 이 전략을 다른 subcellular 또는 organismal settings에서 확장하거나 배포할 때 직면할 수 있는 여러 가지 문제가 있을 것으로 예상됩니다. multi-node orthogonal circuit function의 demonstration은 우리 접근 방식의 확장성에 대한 강력한 초기 증거를 제공하지만, 여러 clamp across multiple regulatory complexes의 stoichiometric distribution에 미치는 영향을 포함하여 higher-order networks를 구성할 때 제한이 발생할 수 있습니다. Clamp concentration은 특히 시스템을 다른 organismal settings으로 이식할 때 시스템 튜닝에 중요한 요소가 될 수 있습니다. 이 연구에서 clamp는 complex assembly에 허용되는 농도로 발현되었지만, clamp의 lower expression은 assembly를 유도하기에 충분하지 않을 수 있으며, clamp concentration이 지나치게 높으면 synTF squelching으로 인해 활성화가 제한될 수 있습니다. 이 문제를 극복하려면 complex assembly를 매개하기 위해 clamp species를 조정하거나 additional clamp species를 개발해야 할 수도 있습니다. 위에서 설명한 것처럼, 다른 유형의 molecular networks (예: intracellular signaling or cell-cell communication)에서 특이성을 설계하기 위해 cooperative assembly strategy를 사용할 수 있습니다. 하지만 저희의 synTF toolkit은 synthetic part sets에 비해 상대적으로 성숙되어 있으며, 잘 이해되고 정량적으로 예측 가능한 biophysical properties를 가지고 있습니다. 다른 유형의 네트워크에 대한 Molecular assembly strategies는 동일한 수준의 프로그래밍 가능성에 도달하기 위해 extensive component set validation이 필요할 수 있습니다. 마지막으로, 다른 organismal host, 특히 signaling pathways에서 PDZ 도메인의 광범위한 사용이 현재의 clamp 설계를 방해할 수 있는 인간 및 기타 포유류 세포에 전략을 이식하는 데 어려움을 겪을 수 있습니다. 또한, 인접한 genomically integrated CRM에 대한 synTF의 mutually reinforced binding이라는 단순한 전략을 사용하여 cooperative assembly를 프로그래밍하는 것은 mammalian transcriptional regulation의 대부분을 차지하는 chromatin regulatory mechanisms을 설명하지 못하기 때문에 강력한 cooperativity or activation를 달성하기에 충분하지 않을 수 있습니다. 이는 transgene silencing이 특히 심각한 primary cells에서 특히 어려울 수 있습니다.