인간과 침팬지 stem cells의 genetic dependencies 비교 환경
Abstract
유인원에 대한 비교 연구는 우리의 진화를 들여다볼 수 있는 창을 제공하지만, hominin evolution 과정에서 나타난 세포 차이의 범위와 정체는 아직 밝혀지지 않은 부분이 많습니다. 우리는 인간 세포가 뚜렷한 genetic dependencies를 보이는지 평가하기 위해 comparative loss-of-function approach를 확립했습니다. 인간과 침팬지 pluripotent stem cells에서 genome-wide CRISPR interference screening을 수행하여 세포 증식에 species-specific effects를 미치는 75개의 유전자를 확인했습니다. 이 유전자들은 cell-cycle progression과 lysosomal signaling을 포함한 일관된 프로세스로 구성되어 있으며, 오랑우탄 세포와 비교하여 human-derived로 확인되었습니다. neural progenitor cells와 cerebral organoids에서 CDK2 및 CCNE1 depletion에 대한 Human-specific robustness가 지속되어, human brain expansion의 potential evolutionary mechanism으로서 G1-phase length hypothesis를 뒷받침했습니다. 우리의 연구 결과는 인간 세포의 진화적 변화가 essential genes의 landscape를 재편하고 종 간의 latent cellular 및 molecular differences를 체계적으로 밝혀낼 수 있는 플랫폼을 구축했음을 보여줍니다.
Figure
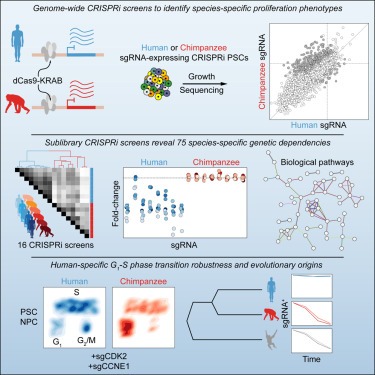
Figure 1. 인간과 침팬지 줄기세포에서 Genome-wide CRISPRi screens을 통해 candidate species-specific genetic dependencies 확인.
(A) 인간과 침팬지의 종 차이를 예술적으로 표현하기 위해 제인 구달의 원본 초상화와 키발레 침팬지 프로젝트의 성인 암컷 침팬지가 함께 있는 CRISPRi screening approach의 모식도.
(B) WTC11 및 C3649 technical replicates에 대한 sgRNA log2 fold change의 Scatterplots 및 4개 화면 모두에서 essential genes의 intersection을 보여주는 UpSet plot.
(C) 각 화면에 대한 Precision-recall analysis (왼쪽 상단). DepMap essential and non-essential genes을 사용하여 Precision and recall을 측정했습니다.
(D) genome-wide CRISPRi screens에서 Species-level gene log2 fold change.
Figure 2. 5개의 인간 개체와 6개의 침팬지 개체를 대상으로 검증된 Species-specific genetic dependencies.
(A) validation sgRNA library design 및 CRISPRi screening approach의 모식도.
(B) 16개의 validation CRISPRi screens에서 sgRNA 프로파일에 대한 Pearson correlations 및 hierarchical clustering의 Heatmap.
(C) t0(검은색 원) 및 tfinal(빨간색 및 파란색 원)에서의 sgRNA 수에 대한 Principal-component analysis.
(D) DESeq2로 모델링한 sgRNA 수의 log2 fold change의 Scatterplot.
(E) validation CRISPRi screens에서 Species-level gene log2 fold change.
(F) individual, species, and time point (t0 대 tfinal)에 따른 sgRNA 수의 sources of variation을 정량화하기 위한 Dream-variancePartition analysis.
(G) validation CRISPRi screens에서 cellular proliferation에 species-specific effects를 미치는 75개 유전자에 대한 gene log2 fold change 및 hierarchical clustering Heatmap (1% FDR).
Figure 3. Core species-specific genetic dependencies.
(A) STRING protein-protein associations을 가진 Species-specific genetic dependencies.
(B) ATP6AP1, ATP6AP2 및 ATP6V0C를 표적으로 하는 sgRNA에 대한 log2 fold change의 Strip plots.
(C) CDK2, CCNE1 및 CDK4를 표적으로 하는 sgRNA에 대한 log2 fold change의 Strip plots.
(D) 인간(28126B, 파란색, n = 3) 및 침팬지(40280L, 빨간색, n = 3) PSC의 knockdown efficiency에 대한 qRT-PCR 측정 결과.
(E) 세 가지 wild-type 인간(H1, 21792A, 28126B) 및 세 가지 wild-type 침팬지(3624K, 40280L, 8861G) cell lines, ATP6AP1 또는 ATP6AP2가 depleted cell lines(28126B 및 40280L)에 대한 phospho-S6 (pS6) 발현 및 GAPDH loading control Western blot
Figure 4. 인간과 침팬지 세포의 cell-cycle progression에 대한 Divergent regulation.
(A) CDK1/CDK2 regulatory network의 개략도.
(B) co-culture에서 성장한 침팬지 wild-type cells (GFP-)와 sgRNA-containing cells (GFP+)의 Cell-cycle proportions.
(C) CDK2, cyclin E1, RBL1, FAM122A를 knockdown 시켰을 때 인간(28126B, 파란색) 및 침팬지(40280L, 빨간색) 세포의 G1 phase 비율 변화(two-tailed t test로 계산).
(D) core cell-cycle regulators에 대한 인간과 침팬지 PSC의 Comparative gene expression data.
(E) CDK1 overexpression degree에 대한 qRT-PCR 측정.
(F) CDK2 또는 cyclin E1 knockdown과 함께 CDK1의 overexpression에 따른 G1 phase의 cells의 비율 변화.
(G) no drug, Chk1 inhibitor prexasertib (CHK1i), or WEE1 inhibitor adavosertib (WEE1i) 투여 시 FAM122A sgRNA-containing cells의 비율 변화.
(H) no drug, CHK1i, or WEE1i 투여 상태에서 co-culture하여 성장시킨 wild-type 인간(파란색, 21792A) 대 wild-type 침팬지(빨간색, 40280L) 세포의 비율.
Figure 5. cell-cycle perturbations에 대한 Human-specific robustness는 neural progenitor cells에서 지속됩니다.
(A) PSC를 neural progenitor cells (NPC)로 분화시키는 모식도.
(B) co-culture에서 성장한 wild-type neural progenitor cells (GFP-)와 sgRNA-containing cells (GFP+)의 Cell-cycle proportions.
(C) CDK2, cyclin E1 또는 RBL1의 depletion 시 G1 phase에서 인간(28126B, 파란색) 및 침팬지(40280L, 빨간색) NPC의 비율 변화(two-tailed t test로 계산).
(D) FAM122A depletion 시 G2 phase에서 NPC의 비율 변화(two-tailed t test로 계산).
Figure 6. cerebral organoids의 cell-cycle perturbations에 대한 Human robustness.
(A) 18일째에 bright-field microscopy로 측정한 인간(H1) 및 침팬지(8861G) cerebral organoids의 Organoid size measurements.
(B) 각 sgRNA condition의 organoids Representative images와 corresponding image segmentation.
(C) 9일차 인간(H1, 20961B, 23555A, 파란색) 및 침팬지(8861G, 40280L, 빨간색) organoids의 Cell-cycle measurements.
Figure 7. 오랑우탄 PSC는 species-specific genetic dependencies의 evolutionary origin을 시사합니다.
(A) 인간(파란색, 28126B), 침팬지(빨간색, 40280L), 오랑우탄 PSC(보라색)에서 시간에 따른 CDK2 sgRNA-containing cells의 상대적 비율 변화(two-tailed t test로 계산).
(B-E) 시간 경과에 따른 Relative sgRNA fraction 및 (B) CDK4, (C) ATP6AP1, (D) KAT6A 및 (E) UFL1을 표적으로 하는 sgRNA에 대한 qRT-PCR 측정값.
Disscussion
Loss-of-function은 model organism의 발달을 조절하는 유전자에 대한 근본적인 통찰력을 제공했습니다. 여기서는 인간과 침팬지 PSC에 genetic screens을 적용하여 essential genes의 요구 사항이 밀접하게 관련된 종에 따라 다를 수 있는지 조사했습니다. paired genome-wide CRISPRi screens을 수행하여 서로 다른 genetic dependencie의 풍경을 발견했습니다. 인간과 침팬지 PSC는 cellular morphology, response to in vitro differentiation protocols, and core set of essential genes이 유사하지만, cellular proliferation을 제어하는 데 서로 다른 역할을 하는 75개의 유전자를 확인했습니다. 이러한 유전자 중 다수가 일관된 protein complexes와 biochemical pathways로 구성되어 있음을 관찰했습니다. 이와 대조적으로, RNA-seq 및 chromatin state profiling을 포함한 existing state-of-the-art comparative approaches은 인간과 비인간 영장류 간에 차별적으로 발현되고 접근 가능한 수천 개의 유전자를 확인했지만 survival, proliferation, and differentiation과 같은 key cellular processes에서 각 유전자의 역할을 직접 평가할 수는 없습니다. 또한, 다르게 발현되는 유전자 간의 일관성이 제한적이기 때문에 서로 다른 경로나 protein complexes를 정확히 찾아내기가 어렵습니다. 따라서 우리의 데이터는 gene regulation and chromatin states에 대한 기존 연구와 연결되는 풍부한 리소스로 구성되어 있으며, 미래의 candidate gene approaches을 위한 기능적 유전체학 가이드를 제공합니다.
PSC에서 관찰한 genetic dependencies는 발달 과정에서 나타나는 organismal differences와 어떤 관련이 있을까요? 흥미롭게도, unbiased genome-wide screening approach에서 나타난 가장 강력한 관찰 중 하나는 NPC에서 지속되는 cell-cycle factor의 depletion에 대한 human-specific robustness이었습니다. 이 발견은 cell-cycle regulation의 변화가 human-specific cortical expansion에 영향을 미칠 수 있다는 오랜 가설과 일치합니다. G1-phase length hypothesis은 NPC에서 factors lengthening G1 duration이 non-proliferative neuronal fates으로 분화할 확률을 높이는 반면, factors reducing G1 length는 NPC의 proliferative self-renewal을 촉진한다고 제안합니다. 실제로 마우스 NPC에서 CDK2 또는 CDK4의 loss는 G1 length를 연장하고 expense of self-renewal를 통해 premature neuronal differentiation을 유발합니다. 반대로, 마우스와 흰 족제비에서 CDK4와 cyclin D1의 exogenous overexpressio은 G1 length를 줄이고 basal progenitor cells의 self-renewing divisions을 촉진하며 구조적으로 정상적인 six-layered cortex를 보존하면서 brain size and cortical area를 증가시킵니다. 인간의 경우 cyclin D2 안정성을 촉진하는 mutations 은 megalencephaly를 유발합니다. 이러한 연구는 발달 중 brain expansion에 대한 G1/S transition inputs의 영향을 강조합니다. 그러나 최근 인간 진화에서이 developmental mechanism이 구체적으로 변경되었는지 여부는 여전히 탐구되지 않은 상태로 남아있었습니다. 인간 NPC가 침팬지 NPC보다 CDK2 또는 cyclin E1을 동등하게 억제했을 때 cycling을 계속할 가능성이 더 높다는 사실을 입증함으로써 제안된 developmental mechanism을 인간 진화에서 발생한 molecular changes와 연결할 수 있습니다. 발달 과정에서 neural progenitor pool의 크기에 영향을 미치는 physiological stressors이 발생하지만, external environmental stimuli or the intrinsic tempo of differentiation이 인간과 침팬지 사이에서 어떻게 달라지고 우리가 관찰한 G1 regulation의 변화와 어떻게 상호작용하는지는 아직 알려지지 않았습니다. Human-specific genetic dependencies는 cell behavior의 evolutionary changes 또는 developmental systems drift (a process in which cell behaviors are conserved)로 인해 발생할 수 있습니다. cell behavior에 미치는 영향과 관계없이, altered genetic dependencies 또는 drug sensitivities는 disease vulnerabilities과 추가적인 evolutionary changes에 대한 최근 evolved substrates를 나타냅니다. 우리는 인간과 침팬지 NPC의 반응을 더 넓은 범위의 genetic and physiological perturbations에 연결하는 향후 연구가 human lineage를 따라 NPC의 proliferative capacity이 증가한 evolutionary mechanisms에 대한 추가 통찰력을 제공할 것으로 기대합니다.
human-specific genetic variants and gene expression changes는 다양하고 대부분 중립적이기 때문에 인간 진화의 molecular basis를 연구하는 노력은 건초 더미에서 바늘 찾기와 비교되어 왔습니다. 대조적으로, 인간과 침팬지 PSC가 명확한 발현이나 protein-coding sequence divergence가 없는 유전자에 대해서도 distinct genetic dependencies를 나타낸다는 우리의 발견은 protein-coding sequence divergence에서 recently evolved functional changes를 분리하는 데 complementary approach를 제공합니다. 이 전략은 TCGA과 같은 sequencing and transcriptomics efforts에서 DepMap과 같은 functional genetics-based efforts에 이르기까지 암 유전학 연구의 발전을 반영합니다. 또한 종양에서 independent recurrence를 기반으로 driver mutations을 식별할 수 있는 반면, 인간의 진화는 단 한 번만 일어났기 때문에 functional genomics platform의 부가가치를 강조할 수 있습니다. 우리는 loss-of-function profiling을 인간 내 genetic variation and disease risk에 대한 cellular models로 확장하여 shared vulnerabilities and convergent pathway level differences를 식별할 수 있을 것으로 기대합니다. 마지막으로, 우리의 접근 방식은 분화된 세포 유형에 쉽게 적용될 수 있으며 cell phenotypes의 higher dimensional measurements과 연결될 수 있으며, 발달 단계에 따른 종 차이의 분자 제어를 이해하기 위한 향후 노력의 문을 열어줍니다.