uncultured bacterium의 antibiotic의 immutable target에 결합합니다.
Abstract
Antimicrobial resistance는 전 세계적으로 주요 사망 요인중 하나입니다.
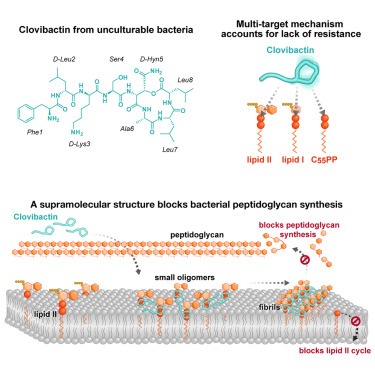
여기에서는 uncultured soil bacteria에서 분리한 antibiotic인 clovibactin의 발견을 보고합니다. clovibactin은 drug-resistant Gram-positive bacterial pathogens을 detectable resistance 없이 효율적으로 죽입니다. biochemical assays, solid-state nuclear magnetic resonance, and atomic force microscopy를 사용하여 clovibactin의 작용 방식을 분석합니다. clovibactin은 여러 essential peptidoglycan precursors (C55PP, lipid II, and lipid IIIWTA)의 pyrophosphate를 표적으로 삼아 cell wall synthesis를 차단합니다. clovibactin은 특이한 hydrophobic interface를 사용하여 pyrophosphate를 단단히 감싸지만 precursors의 다양한 구조적 요소를 우회하여 저항성이 부족합니다. 선택적이고 효율적인 target binding은 precursors를 lipid-anchored pyrophosphate groups을 포함하는 bacterial membranes에만 형성되는 supramolecular fibrils로 격리함으로써 달성됩니다.
이 potent antibiotic은 resistance development 없이 bacterial pathogens을 죽이는 개선된 치료제를 설계할 수 있는 잠재력을 가지고 있습니다.
Figure
Discovery of clovibactin
Figure 1. Biosynthetic gene cluster와 clovibactin의 biosynthesis 제안
whole-genome sequencing을 통해 clovibactin biosynthesis와 관련된 gene cluster (cloABCD)를 확인했습니다.
[Figure 1] clovibactin은 linear N terminus에 두 개의 D-amino acids가 있고, depsi-cycle에 uncommon residue D-3-hydroxyasparagine이 존재. depsi-peptide인 teixobactin과 어느 정도 유사함.
clovibactin은 화합물에 존재하는 두 개의 양전하를 품고 있는 linear N terminus가 상당히 짧음(clovibactin은 4개의 residues, teixobactin은 7개의 residues). 또한, teixobactin에는 clovibactin에는 없는 nusual enduracididine residue가 포함된 macrolactone에 양전하를 띤 두 개의 amino acids 중 하나가 포함되어 있음.
[Figure 1] 제안된 clovibactin의 biosynthetic pathway (Figure 1)는 dual-function condensation domains에 의해 3개의 epimerizations이 수행되는 8개의 amino acids의 assembly line condensation과 TauD/TfdA dioxygenase CloD에 의한 Asn5의 β-hydroxylation을 통해 진행. 이 hydroxylation은 NRPS에서 방출되어 macrocyclic lactone을 형성하기 위한 cyclization point를 제공. clovibactin과 teixobactin BGC를 비교했을 때, BLASTN alignment에 의한 동일성은 72%.
– time-dependent killing 조사
– Clovibactin의 강력한 antimicrobial activity와 낮은 cytotoxicity를 감안하여, 이 antibiotic의 in vivo 작용을 조사
– Antibiotic이 immune system의 도움 없이도 감염을 제어할 수 있는지 평가
Figure 2. clovibactin은 in vitro and in vivo에서 S. aureus를 사멸시킵니다.
(A) S. aureus의 Time-dependent killing.
(B 및 C) S. aureus에서 Clovibactin-induced lysis. Cells of S. aureus SA113 and a ΔaltA mutant를 표시된 대로 각 화합물과 함께 2 × MIC(B) 및 5 × MIC(C)에서 24시간 동안 배양했습니다.
(D) S. aureus 및 S. aureus ΔatlA(clovibactin, teixobactin, and vancomycin [각각 2 × MIC])의 Time-dependent killing.
(E) Watson LIMS software를 사용하여 측정한 마우스 모델에서 clovibactin의 Pharmacokinetic parameters.
(F) 투약 전과 치료 후 24시간 동안 허벅지 감염 모델에서 bacterial load.
[Figure 2A] clovibactin은 1차 defense antibiotic인 vancomycin에 비해 S. aureus을 죽이는 데 더 효과적임.
[Figure 2B, C] 우리는 clovibactin이 세포 배양을 비정상적으로 strong lysis시키는 것을 발견하고 이 효과를 정량화.
clovibactin(2 × MIC)은 teixobactin보다 더 뚜렷한 strong lysis를 유도함. 놀랍게도 ΔatlA mutant에서는 lytic events가 약간만 영향을 받음.
- clovibactin-induced lysis가 주로 AtlA activity에 의존하지 않는다는 것을 시사
[Figure 2D] clovibactin에 의한 S. aureus 사멸은 wild type과 ΔatlA mutant 모두에서 거의 영향을 받지 않았으며, 이는 teixobactin과 대조적임ㅇ.
[Figure 2E] clovibactin plasma levels을 LC-MSMS로 측정하고 software package Watson LIMS를 사용하여 PK parameters를 측정.
[Figure 2F] clovibactin은 bacterial burden을 줄이는 데 있어 vancomycin과 비슷.
Target identification
– structure의 novelty와 이 화합물의 유망한 개발 가능성을 고려하여 clovibactin의 molecular target을 규명
– clovibactin이 억제할 수 있는 biosynthetic pathway를 확인
Figure 3. clovibactin은 cell wall biosynthesis를 target으로 합니다.
(A) clovibactin이 S. aureus의 macromolecular biosyntheses에 미치는 영향.
(B) major biosynthesis pathways에 대한 간섭을 확인하기 위해 selected promotor-lacZ fusions을 가진 B. subtilis bioreporter strains를 사용했습니다.
(C) clovibactin treatment는 B. subtilis의 phase-contrast microscopy로 관찰한 cell-shape deformations and characteristic blebbing을 초래합니다.
(D) clovibactin(1 × MIC, 파란색)은 B. subtilis PliaI-lux에서 lux operon의 발현으로 관찰된 바와 같이 Plial을 강력하게 유도했습니다.
(E) 다양한 농도의 clovibactin으로 S. aureus을 처리한 후 soluble cell wall precursor UDP-MurNAc-pp의 Intracellular accumulation.
(F) clovibactin은 in vitro에서 peptidoglycan (PGN) 및 wall teichoic acid (WTA) synthesis의 membrane-associated steps를 억제합니다.
(G) cell wall precursors에 의한 clovibactin의 antimicrobial activity의 Antagonization.
[Figure 3A] clovibactin은 radiolabeled N-acetylglucosamine (GlcNAc)이 cell wall로 통합되는 것을 특이적으로 방해한 반면, DNA, RNA 및 protein biosynthesis는 영향을 받지 않았음.
[Figure 3B] clovibactin에 의해 B. subtilis PypuA-lacZ에서 LacZ의 발현이 특이적으로 유도되었으며, 이는 cell wall biosynthesis에 간섭이 있음을 나타냄.
[Figure 3C] clovibactin으로 B. subtilis를 처리하면 phase-contrast microscopy로 시각화된 것처럼 cell-shape deformations이 유도됨. 이러한 blebbing phenotype은 많은 cell-wall-acting antibiotics에 의해 특징적으로 유도되며, 대조군으로 사용된 protein synthesis inhibitor clindamycin은 아니지만 teixobactin, hypeptin, or vancomycin에서도 유사하게 관찰되어 cell wall biosynthesis에 대한 직접적인 간섭을 뒷받침함.
[Figure 3D] clovibactin은 PliaI-lux를 강력하게 유도하여 lipid II biosynthesis pathway와 직접적으로 상호작용할 수 있음을 시사.
[Figure 3E] vancomycin에서 관찰된 바와 같이, clovibactin으로 S. aureus을 농도를 증가시켜 처리하면 cytoplasm에 UDP-MurNAc-pp가 축적됨.
[Figure 3F] clovibactin은 dose-dependent fashion으로 lipid I, lipid II, lipid IIIWTA, or undecaprenyl-pyrophosphate (C55PP)을 substrate로 사용하는 모든 cell wall biosynthesis를 억제했으며, 이는 enzyme function을 억제하기보다는 이러한 lipid intermediates에 결합하는 것으로 나타남.
주목할 점은 clovibactin은 C55PP와 lipid II를 substrate로 사용하는 반응도 동일하게 억제함.
[Figure 3G] purified cell wall lipid intermediates를 추가하면 clovibactin의 antimicrobial activity가 antagonized되고 S. aureus의 성장이 회복됨.
- C55PP가 clovibactin의 주요 표적임을 시사.
Oligomerization upon target binding
– solid-state NMR(ssNMR)을 사용하여 lipid bilayers에서 clovibactin과 lipid II 사이의 상호작용을 연구
– near-native conditions에서 membrane targets에 결합하는 antibiotics의 molecular mechanism을 조사
Figure 4. 막에서 clovibactin/lipid II complex의 4ssNMR 구조 모델 및 oligomerization
(A) aqueous solution (장미색)의 free clovibactin에 겹쳐진 membranes (청록색) 내 lipid-II-bound clovibactin의 2D NH ssNMR 스펙트럼.
(B) 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) membranes에서 lipid-II-bound teixobactin and clovibactin의 Site-resolved 15N R1rho dynamics.
(C) NMR-derived dynamics 그림.
(D 및 E) complex의 2D CC 스펙트럼을 확대하면 clovibactin의 head-to-tail contacts하는 것을 볼 수 있으며, 이는 complex에서 clovibactin의 dimeric (supramolecular) arrangement를 시사합니다.
(F) (D) 및 (E)에서 볼 수 있는 Phe1-Hyn5와 Phe1-Leu8 사이의 head-to-tail contacts를 개략적으로 나타낸 그림.
(G) Atto-labeled lipid II로 도핑되고 clovibactin으로 처리된 DOPC GUV의 domain/cluster formation을 보여주는 Confocal microscopy.
[Figure 4A] backbone amide protons의 Large signal shifts는 clovibactin이 lipid II binding 시 주요한 형태 변화를 겪는다는 것을 보여줌.
[Figure 4B, C] 전체적으로 clovibactin은 complex 내에서 고정되어 있으며, 이는 더 큰 supramolecular structure가 형성되었음을 나타냄.
N terminus (Leu2 및 Lys3)은 complex 형성 시 강하게 rigidifies되는데, 이는 target binding 시 N terminus가 self-assembly를 large clusters로 유도하는 teixobactin의 antimicrobial action을 연상시킴.
[Figure 4D, E] supramolecular structure의 형성과 일치하여, 이는 clovibactin 사이의 intermolecular contacts일 가능성이 높음. N-terminal과 C-terminal residues 사이의 intramolecular distances가 약 8Å의 distance threshold로 CC magnetization transfer를 하기에는 너무 멀어 보임.
[Figure 4F] 관찰한 contact는 clovibactin molecules의 antiparallel dimeric organization과 일치.
- suprastructure의 intermolecular clovibactin-clovibactin contacts가 주로 backbone-backbone mediated이며 주로 N-terminal residues를 포함한다는 것을 보여줌.
[Figure 4G] clovibactin이 없으면 large clovibactin-lipid II patches가 형성되는 것을 명확하게 보여줌.
Flexible fibrils
– clovibactin-지질 II 복합체의 초분자적 특성과 막 상호작용을 더 자세히 조사
(H) 10분간의 interactions 후 clovibactin-lipid II supramolecular fibrils의 형성을 보여주는 3D 렌더링된 HS-AFM 이미지.
(I) 아래쪽 패널: mobility-edited ssNMR 실험에 따르면 clovibactin은 lipid II-bound state에서 teixobactin보다 훨씬 더 water-accessible한 것으로 나타났습니다.
상단 패널: clovibactin과 teixobactin의 ssNMR-derived topology 및 membrane insertion.
(J) clovibactin-lipid II fibrils 조립 후 time-lapse HS-AFM video (Video S1)의 스냅샷.
(K) clovibactin의 작용 방식 모델.
[Figure 4H, J] lipid II가 도핑된 membranes에 5 μM의 clovibactin을 첨가한 후 몇 분 이내에 membrane surfac에 fibrils이 형성되는 것을 보여줌.
[Figure 4I] teixobactin-lipid II fibrils을 사용한 이전의 ssNMR 연구에서는 phospholipid tails과 뚜렷한 상호 작용을 보였는데, 이는 AFM 및 전산 연구에서 관찰된 teixobactin fibrils의 membrane내 깊게 insertion되는 것과 잘 일치.
[Figure 4K] 반면에 clovibactin-lipid II supra-structures는 lipid tails과 약하게만 상호작용하는데, 이는 clovibactin으로 형성된 supra-structures가 훨씬 덜 깊이 insertion되고 아마도 membrane surface 위에 놓여 있음을 의미.
- 이러한 membrane insertion의 차이는 teixobactin의 N terminus에 3개의 long hydrophobic anchors (Ile2, Ile5, Ile6)가 존재하는 반면, clovibactin의 훨씬 짧은 N terminus에는 단 하나의 hydrophobic anchor (Leu2)만 존재하기 때문에 합리화할 수 있음.
The complex interface
– clovibactin이 PPi 그룹을 어떻게 표적으로 삼는지 조사
Figure 5. 인터페이스와 supramolecular structure
(A) lipid II의 Chemical structure.
(B) clovibactin 첨가 시 liposomes의 1D 31P ssNMR 데이터는 lipid II PPi signals의 현저한 변화를 보여줍니다.
(C) liposomes 내 13C,15N-clovibactin-13C,15N-lipid II complex의 2D CC ssNMR 데이터는 MurNAc 당 및 depsi-cycle의 hydrophobic side chains과의 interfacial contacts를 보여줍니다.
(D) clovibactin과 lipid II sugars의 interfacial contacts의 합계.
(E) interface Illustration: PPi와 MurNAc는 직접 근접해 있고, GlcNAc는 멀리 떨어져 있으며, pentapeptide는 유연하고 인터페이스에 관여하지 않습니다.
[Figure 5A] clovibactin이 보존된 pyrophosphate (PPi) 그룹, sugars인 MurNAc 및 GlcNAc로 구성된 headgroup, vancomycin과 같은 antibiotics에 대한 내성을 부여하는 variation이 있는 pentapeptide를 가진 complex lipid인 lipid II를 정확히 표적으로 삼는 방법을 알아보고자 함.
[Figure 5B] clovibactin을 첨가하자 1D 31P ssNMR 데이터는 지질 II의 pyrophosphate 신호에 현저한 변화를 보여 direct coordination을 시사
lipid I (GlcNAc sugar가 없는 lipid)에서도 유사한 신호 변화가 관찰됨.
- 이러한 데이터를 종합하면 clovibactin은 cell wall biosynthesis를 여러 단계에 걸쳐 차단하는 multi-targeting antibiotic임을 확인할 수 있음.
또한 clovibactin의 backbone amino protons이 PPi 그룹을 depsi-cycle의 amino protons (Hyn5, Ala6, Leu8)와 directly coordinate 하는 것을 보여줌. 또한, 약하지만 명확하게 식별할 수 있는 신호는 N terminus-depsi-cycle junction의 Ser4가 PPi 그룹에 가깝게 근접해 있음을 보여줌.
[Figure 5C] target binding에 대한 lipid II sugars와 pentapeptide의 역할을 확인하기 위해 13C,15N-clovibactin과 13C,15N-lipid II의 complex를 준비하여 950 및 1,200MHz의 ultra-high magnetic fields에서 일련의 2D PARISxy32 13C13C 스펙트럼을 측정.
[Figure 5D] clovibactin과 lipid II 당 사이에 총 12개의 모호하지 않은 interfacial contacts이 관찰되었으며, 이 중 10개는 Ala6 및 Leu8과 관련됨.
- 이는 뎁시 사이클이 지질 II 헤드그룹과 직접 상호작용한다는 것을 확인시켜 줍니다.
- 이는 또한 Leu7과 당류 사이에서 관찰된 추가적인 모호한 계면 접촉과도 일치
[Figure 5E] pentapeptide의 마지막 네 가지 residues (γGlu∗2, Lys∗3, Ala∗4 및 Ala∗5)를 독점적으로 검출
Structural model of the complex
– complex의 구조 모델을 계산
(F) clovibactin-lipid II complex의 ssNMR-derived structural model.
(G) 계산된 ssNMR 구조 모델의 인터페이스는 잘 중첩되어 있으며, hydrophobic depsi-cycle side chains (Ala6, Leu7, Leu8)이 lipid II PPi 그룹을 장갑처럼 감싸고, MurNAc의 hydrophobic side와 상호작용하는 것을 보여줍니다.
(H) cationic K3 및 polar S4 side chains은 lipid II PPi 그룹과 유리하게 상호 작용합니다.
(I) clovibactin의 Hydrophobic residues는 장갑처럼 PPi 그룹을 감싸고 있으며, 이는 boundary water release에 의해 엔트로피적으로 유리하게 나타납니다.
[Figure 5F] 얻어진 구조는 잘 중첩되었으며, HS-AFM으로 관찰된 supramolecular structures와 일치하는 fiber-like supramolecular structures로 늘어날 수 있는 clovibactin의 antiparallel dimeric units을 보여줌.
[Figure 5G] despi-cycle의 hydrophobic residues (Ala6, Leu7, Leu8)는 hydrophobic glove structure를 연상시키는 PPi moiety를 깔끔하게 감싸고 있으며, 이는 ssNMR distance measurements와 일치.
[Figure 5H] anionic lipid II PPi group과 N terminus의 cationic (Lys3) 및 polar (Ser4) side chains 사이의 유리한 closer-distance interactions을 일관되게 보여줌.
[Figure 5I] clovibactin과 sugars 사이의 specific interactions이 없는 것은 hydrophobic cage의 유리한 엔트로피 기여에 의해 보상될 수 있는데, hydrophobic cage가 유연하게 유지되지만 PPi 그룹을 조정하는 boundary water molecules를 제거하기 때문.
- 이는 membrane surface에 정착하고 안정적인 supramolecular structures를 형성하는 것이 precursors의 효과적인 결합에 기여한다는 것을 시사
Disscussion
clovibactin은 다양한 세포벽 생합성 경로에서 multiple essential cell wall precursors C55PP, lipid II 및 lipid IIIWTA의 pyrophosphate moiety에 결합합니다. clovibactin은 이러한 precursors의 PPi moiety에 결합합니다. 일반적으로 PPi는 PPi-containing nucleoside phosphates와 함께 죽은 세포에서 방출되고 환경에 일반적으로 존재하기 때문에 antibiotic의 표적으로 부적합한 것으로 보입니다. 동시에 pyrophosphate는 cell wall lipid intermediates의 필수적이고 immutable한 분자이며, 예를 들어 lipid II의 pentapeptide나 sugars와 같은 분자의 다른 부분과 달리 mycobacteria에서 변형되거나 돌연변이를 일으킬 수 있습니다. 또한, lipid II pentapeptide의 D-Ala-D-Lac substitution은 terminal D-Ala-D-Ala motif에 결합하는 vancomycin에 대한 일반적인 resistance mechanism입니다. immutable PPi에 결합하면 이 화합물에 대한 내성이 검출되지 않는 것을 설명할 수 있습니다. clovibactin이 어떻게 central peptidoglycan precursor lipid II의 PPi에 단단하고 선택적으로 결합하는지는 이 연구에서 상세한 구조 분석을 수행하여 해결하고자 하는 흥미로운 질문입니다.
clovibactin은 external medium에서 cell wall precursors와 결합하는 membrane surface에 침전됩니다. N terminus의 leucine side chain은 clovibactin을 targets sites의 membrane에서 분할하는 데 도움을 줍니다. clovibactin의 depsi-cycle의 Backbone amino protons은 PPi를 직접 조정하는 반면, hydrophobic depsi-cycle side chains (Ala6, Leu7, Leu8)은 adjustable glove처럼 PPi 그룹을 둘러싸는 특이한 상호작용으로, 이는 아마도 boundary water를 대체하여 엔트로피적으로 유리하고 별개의 indispensable cell wall precursors와 효율적으로 결합할 수 있게 하는 것으로 추정됩니다. hydrophobic warhead로 highly polar target (PPi)을 공격하는 것은 놀랍고 직관적이지 않은 행동 방식입니다. clovibactin은 용액에서는 PPi에 눈에 띄게 결합하지 않는 반면, membranes에서 수집한 ssNMR 데이터는 이것이 clovibactin이 lipid II target에 결합하는 principle moiety임을 보여줍니다. lipid precursors의 PPi moiety에 대한 selective binding은 supramolecular complex의 형성과 target에 결합된 상호 작용하는 clovibactin 분자의 안정적인 higher-order fibrillar assembly의 올 oligomerization에 의해 달성되는 것으로 보입니다. 이는 아마도 short N terminus가 oligomerization domain으로 작용하는 clovibactin 분자의 antiparallel arrangement에 의해 가능한 것으로 추정됩니다. antibacterial action과 clovibactin fibrils 형성에 대한 비슷한 농도는 supra-structures가 killing mechanism의 중요하거나 필수적인 부분임을 시사합니다. bacterial membranes에 쉽게 접근할 수 있는 PPi-carrying molecules이 존재하기 때문에 이러한 구조가 형성될 수 있으며 포유류 세포에 대한 clovibactin의 독성이 없는 것으로 설명할 수 있습니다. clovibactin의 놀라운 특징은 cell lysis를 일으키는 탁월한 능력으로, 이는 teixobactin과 기계적으로 구별되며 S. aureus의 주요 autolysin인 AtlA와는 무관합니다. 이는 WalRK TCS에 의해 제어되는 유전자의 differential cellular response 및 expression pattern과 잘 일치합니다. WalRK는 S. aureus의 유일한 필수 TCS이며 주로 cell wall lytic enzymes의 transcription을 조절하여 cell wall metabolism을 제어하는 데 중요한 역할을 합니다. 안정적인 supramolecular structure의 형성은 이 화합물의 in vivo activity에 유리한 결과를 가져와, 이 화합물이 속한 bacterial surfaces에 집중되고 soluble compound가 체내에서 제거된 후에도 오랫동안 계속 작용할 수 있습니다. 클로비박틴은 단순한 immutable target인 cell wall precursors의 PPi에 대한 작용을 통해 내성을 피하기 위해 진화한 antibiotics에 대한 이해를 넓히고 임상적으로 유용한 수명이 긴 화합물을 합리적으로 설계할 수 있는 길을 제시합니다.
Limitations of the study
clovibactin-lipid II suprastructure의 배열을 결정적으로 확립하기 위해서는 추가 연구가 필요합니다. clovibactin-lipid II interface는 모호하지 않은 ssNMR distance restraints로 잘 설명되지만, suprastructure에서 다른 clovibactin-clovibactin arrangements를 단정적으로 배제할 수는 없습니다. 정확한 supramolecular arrangement를 확립하려면 clovibactin에 synthetic routes를 포함한 selectively isotope-label clovibactin하는 새로운 ssNMR 방법이 필요할 것입니다. 이 연구의 또 다른 한계는 현재로서는 clovibactin에 의해 유도되는 강력한 lytic effect를 담당하는 특정 hydrolase 또는 glucosaminidase를 확인하지 못했다는 점입니다.