후행 리보솜은 힘과 알로스테리를 통해 전사 충실도를 희생시키면서 RNA 중합효소 속도를 높입니다.
Abstract
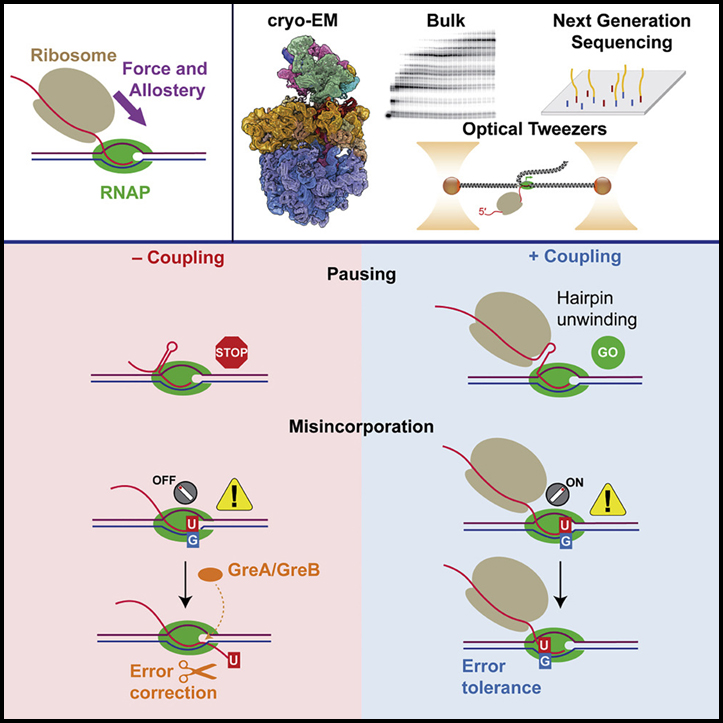
원핵생물에서 번역은 커플링이라고 하는 과정에서 전사되는 mRNA에서 발생할 수 있습니다. 결합하는 동안 리보솜이 RNA 중합효소 (RNAP)에 어떻게 영향을 미치는지는 잘 알려져 있지 않습니다.
이 연구에서는 대장균 커플링 시스템을 재구성하고 리보솜이 RNAP의 일시 중지 및 종료를 방지하고 충실도를 희생시키면서 전체 전사 속도를 두 배로 늘릴 수 있음을 입증했습니다. 또한, 우리는 리보솜에 결합된 단일 RNAP를 모니터링하고 결합이 중합효소의 정지 없는 속도를 증가시키고 기계적 보조력이 결합 효과의 대부분을 설명하기에 충분하다는 것을 보여줍니다. 또한, cryo-EM에 의해 우리는 말단 불일치가 있는 RNAP가 역추적 형태를 채택하는 반면, 결합된 리보솜은 알로스테릭하게 이러한 중합효소를 촉매 활성 반회전 상태로 유도함을 관찰했습니다.
마지막으로, 우리는 연장된 RNAP 일시 중지가 결합된 리보솜을 통한 중합효소 재활성화에 의해 예방될 수 있는 세포 생존에 해롭다는 것을 입증합니다.
Figure
[Figure 1] 전사-번역 커플링의 시험관 내 재구성
(A) pyr 오페론을 묘사한 개략도.
(B) TTC (transcription-translation complex)의 단계별 조립.
(C) -커플링 및 +커플링 반응에 대한 실험 계획.
(D) 변성 우레아 겔에서 32P-α-ATP-표지된 전사물로 모니터링된 -커플링 및 +커플링 반응에서의 전사.
[Figure 2] 커플링 중 pyrL을 따라 증가하는 번역 속도
(A) 전사를 위한 단순화된 동역학 체계.
(B와 D) -커플링 (회색) 및 +커플링 반응 (검정색)에서 P1 (B)과 HP 사이트 (D)에서 RNAP의 동역학의 백분율.
(C) HP 사이트와 -커플링 및 +커플링 반응에서 RNAP의 도달률.
(E) -커플링 및 +커플링 반응에서 종료되는 RNAP의 백분율.
(F) -커플링 및 +커플링 반응에서 종료를 우회하는 RNAP의 백분율.
[Figure 3] 커플링은 RNAP의 정지 없는 속도와 이동 거리 증가
(A) 전사를 모니터링하기 위한 광학 핀셋 설정.
(B) rNTP를 포화시키는 반대 힘 및 rUTP (5μM) 제한 하에서 리보솜이 있거나 없는 반대 또는 보조 힘 하에서 RNAP에 의한 대표적인 전사 흔적.
(C) (B)의 5가지 조건에 대한 RNAP (상단 패널)과 횡단 거리 (하단 패널)의 평균 일시 중지 없는 속도.
(D와 E) (B)의 5가지 조건에 대한 일시 중지 P1 (D)과 HP (E)에서의 교차 시간.
[Figure 4] 커플링에 의한 전사 충실도 감소
(A) 조립된 RNAP의 템플릿 DNA (파란색)와 비템플릿 DNA (보라색)에 어닐링된 RNA (빨간색)를 보여주는 회로도.
(B) 충실도 분석의 실험 계획.
(C) -커플링과 +커플링 반응 ±2 μM GreA에 대한 시간 경과에 따른 전사 진행을 보여주는 변성 우레아 겔.
(D) (B)와 (C)에 묘사된 충실도 분석의 정량화.
(E) 전사 오류를 식별하기 위해 시퀀싱 라이브러리를 준비하는 워크플로.
(F) 상단 패널: -커플링 및 +커플링 반응의 전사 오류 백분율. 중간 패널: -커플링 반응에 대한 +커플링의 상대적 백분율 오차. 하단 패널: 각 위치에서 확인된 잘못 통합된 리보뉴클레오티드를 나타내는 서열 로고.
(G) 커플링 중에 RNAP가 잘못 통합되는 경향이 증가했음을 보이는 볼케이노 플롯.
[Figure 5] RNAP에 의한 지원력 하에서 불일치로 인한 일시 중지 극복
(A) RNAP는 그림의 위치에서 59-nt RNA로 조립되고 1-mM rGTP와 잘못 통합된 +커플링 반응과 -커플링 반응.
(B) +커플링/-커플링에서 디-, 트리-뉴클레오티드 밴드 강도의 비율로서 (A)에서 겔의 정량화.
(C) 말단 rU-dG 불일치가 있는 RNAP는 보조력 기하학을 보이는 개략도.
(D) ~15pN의 평균 일정한 힘으로 유지되는 RNAP의 전사 재시작에 대한 대표적인 추적.
(E) 각 힘 범위에서 다시 시작하는 시간.
(F) 재시작 동역학은 힘 (F)에 대한 재시작 시간 (t)의 지수 의존성을 제안하는 Arrhenius 방정식 모델링.
[Figure 6] RNAP_Free, TTC 및 RNAP_TTC의 Cryo-EM 구조
(A) 말단 불일치 (RNAP_Free)를 품고 있는 RNAP의 왼쪽, cryo-EM 구조. 오른쪽, EM 재구성의 단면.
(B) RNAP_Free 활성 사이트의 확대 이미지.
(C) TTC의 결합된 cryo-EM 구조.
(D) RNAP-리보솜 상호작용 표면.
(E) RNAP_TTC 활성 부위에서 P 부위 tRNA로의 mRNA 경로를 보여주는 TTC 모델의 내부.
(F) RNAP_TTC 활성 사이트의 확대 이미지.
(G) RNAP_TTC 대 RNAP_Free의 회전.
(H) RNAP_Free (상단) 및 RNAP_TTC (하단) 모두에서 β’SI3 도메인 “내부” 역학의 다체 분석.
Disscussion
후행 리보솜은 기계적 및 알로스테릭 수단으로 전사 속도를 높입니다.
RNAP에 의한 전사에는 일시 중지로 강조된 지속적인 rNTP 추가가 포함됩니다. rNTP 추가에는 rNTP 결합, 인산이에스테르 결합 형성 및 RNAP에 의한 전위가 포함되어 주기를 다시 시작합니다. 간단하게 하기 위해 이 세 단계를 하나의 속도 kn으로 축소했습니다. 이 활성 전사 속도 kn은 일시 중지 진입 속도 kp와 역학적으로 경쟁하며 후행 리보솜이 이 경쟁을 왜곡하는 것을 볼 수 있습니다. RNAP에 전방 방향의 힘을 가함으로써 리보솜은 RNAP를 전위된 레지스터로 편향시켜 kn을 증가시킬 수 있습니다. 또한 이 힘은 백트래킹을 방지하고 헤어핀을 펴서 kp를 감소시킬 수 있습니다. 알로스테릭 방식으로 RNAP가 회전 방지 구조를 채택하도록 유도하는 능력에서 리보솜은 아마도 경로를 벗어난 전사 일시 중지 (kp 감소)를 막고 심지어 kn에도 영향을 미칠 수 있습니다. 종합하면, 우리의 결과는 기계적 및 알로스테릭 효과를 통해 리보솜이 kn 및 kp를 조절하여 결합하는 동안 전체 전사율을 향상시킴을 나타냅니다.
리보솜은 RNAP의 충실도를 감소시킵니다.
아이러니하게도, 전사 속도를 증가시키는 리보솜의 동일한 기계적 및 알로스테릭 효과가 전사 충실도를 감소시킬 수도 있습니다. 기계적 힘은 우리의 구조에서 볼 수 있듯이 불일치 베어링 RNAP의 전위된 레지스터로의 순방향 전좌를 촉진하여 광학 핀셋 실험에서 볼 수 있듯이 지속적인 전사를 허용하고 오류에 대한 내성을 증가시킵니다. 또한 인접한 리보솜은 RNAP 역추적을 방해하고 문제가 되는 rNTP의 편집을 방지합니다. 리보솜에 의한 RNAP의 알로스테릭 활성화는 잘못된 혼입에서 일시 중지에 대한 선천적 반응을 방해하고 중합효소에 의한 선회를 억제합니다. 또한, 우리의 결과는 리보솜이 RNAP를 회전 방지 상태로 유지하여 β’SI3 도메인이 촉매 작용을 위해 TL이 TH로 접히는 데 도움이 되는 촉매 부위에 닫히도록 합니다. 특히, 마찬가지로 β’SI3를 편향시켜 촉매 부위에 가까운 위치 (TH 형성에 유리함)를 가정하는 β’F1199 돌연변이도 전사 오입을 증가시킨다. 잘못된 혼입은 정상적인 조건에서는 해롭지만 스트레스 조건에서는 표현형 가변성을 유발하여 박테리아 개체군의 생존 가능성을 높일 수 있다는 이론이 있습니다.
TTC는 거대 분자 충돌을 방지하여 RNA 및 DNA 무결성을 보호합니다.
고정된 RNAP를 구출할 수 없는 리보솜은 또한 초기 전사체에서 리보솜-리보솜 충돌로 번역 “교통 체증”을 유발할 수 있습니다. 최근 SmrB 단백질에 의한 리보솜 구조 경로가 충돌한 리보솜 사이의 독특한 경계면을 인식하는 것으로 밝혀졌습니다. SmrB는 충돌된 리보솜에서 이 기본 mRNA를 절단하여 SsrA 시스템에 의한 단백질 분해 및 리보솜 재활용을 유도합니다. 장기간의 RNAP 실속이 리보솜 충돌을 일으키고 세포 적합성에 영향을 미칠 수 있는지 궁금했습니다. 이를 위해 우리는 리보솜 구조 (ssrA, smrB 및 smrB paralog smrA)에 중요한 유전자의 다양한 결실이 있는 세포에 항생제 슈도우리디마이신 (PUM)을 첨가하여 RNAP를 정지시키는 일련의 세포 생존능 분석을 수행했습니다. 우리는 ΔssrA 균주가 WT 균주에 비해 심각한 성장 결함을 보이는 것을 관찰했으며, 이는 ssrA가 정체된 RNAP에 의해 유도된 리보솜 구조의 핵심 역할임을 시사합니다. 대조적으로, ΔsmrA 및 ΔsmrB 균주는 PUM 처리로 인해 mRNA가 적은 세포에서 더 적은 mRNA가 절단되고 분해되기 때문에 WT 및 ΔssrA 균주보다 더 잘 성장했습니다. 이러한 실험은 리보솜 충돌이 해결되는 RNA 분해 메커니즘으로 인해 RNAP의 긴 일시 중지가 세포에 매우 비용이 많이 들 수 있음을 시사합니다. 우리는 결합이 충실도를 희생하더라도 RNAP에서 일시 중지 기간을 줄임으로써 리보솜-리보솜 충돌을 방지함으로써 적합성 이점을 부여할 수 있다는 가설을 세웁니다.
정지되거나 역추적된 RNAP와 같은 방향으로 충돌하는 DNA 중합효소가 DNA 손상을 유발한다는 것도 밝혀졌습니다. 잘못된 통합으로 인해 RNAP가 중단될 수 있습니다. 세포에서 GreA 및 GreB와 같은 오류 감시 전사 인자는 문제가 되는 rNTP를 즉시 제거하기 위해 RNAP를 지원하는 역할을 합니다. 또는 Mfd 및 Rho ATPase가 정지된 RNAP를 대체할 수 있습니다. 우리는 RNAP가 실수를 간과하고 계속 늘어나도록 함으로써 심각한 DNA 손상을 초래할 수 있는 DNA 중합효소의 움직임을 방해하는 RNAP 장애물을 방지하기 위해 리보솜이 추가적인 방어선 역할을 한다고 제안합니다. 표면적으로는 영구적이고 사소하지 않은 게놈 DNA의 변경과 달리 일시적이고 중요하지 않을 가능성이 가장 높은 RNA의 실수는 두 가지 중 작은 것으로 보입니다.