complete human centromeres의 변화와 진화
Abstract
Human centromeres는 repetitive nature와 large size로 인해 염기서열을 분석하고 조립하기가 매우 어려웠습니다. 그 결과, human centromeric variation의 패턴과 진화와 기능에 대한 모델은 여전히 불완전하지만, centromeres는 가장 빠르게 mutating regions입니다.
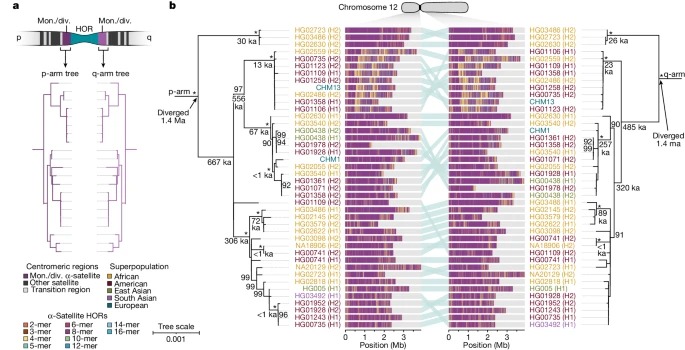
여기에서는 long-read sequencing을 사용하여 second human genome의 모든 centromeres를 완전히 시퀀싱하고 조립하여 finished reference genome과 비교했습니다. 그 결과 두 세트의 centromeres는 고유한 측면과 비교했을 때 single-nucleotide variation이 최소 4.1배 이상 증가했으며 크기가 최대 3배까지 다양하다는 것을 발견했습니다. 또한 새로운 α-satellite higher-order repeats (HOR)의 발견으로 인해 centromeric sequence의 45.8%가 표준 방법으로는 안정적으로 정렬할 수 없다는 사실을 발견했습니다. DNA methylation 및 CENP-A chromatin immunoprecipitation experiments에 따르면 26%의 centromeres가 kinetochore 위치에서 500kb 이상 차이가 나는 것으로 나타났습니다. 진화적 변화를 이해하기 위해 침팬지, 오랑우탄, 원숭이 게놈에서 6개의 염색체를 선택하고 31개의 orthologous centromeres를 시퀀싱하여 조립했습니다. 비교 분석 결과, 각 종마다 α-satellite HOR의 특징적인 변화와 함께 거의 완전한 α-satellite HOR의 idiosyncratic changes가 나타났습니다.
인간 haplotypes의 계통학적 재구성은 중심부에 걸쳐 short (p) arms과 long (q) arms 사이의 재조합이 제한적이거나 전혀 없음을 뒷받침하며, 새로운 α- satellite HOR이 monophyletic origin을 공유한다는 사실을 밝혀 human centromeric DNA의 saltatory amplification 및 mutation rate을 추정할 수 있는 전략을 제공합니다.