합성 역방향 서열이 기본 유전체 상태를 드러낸다
Abstract
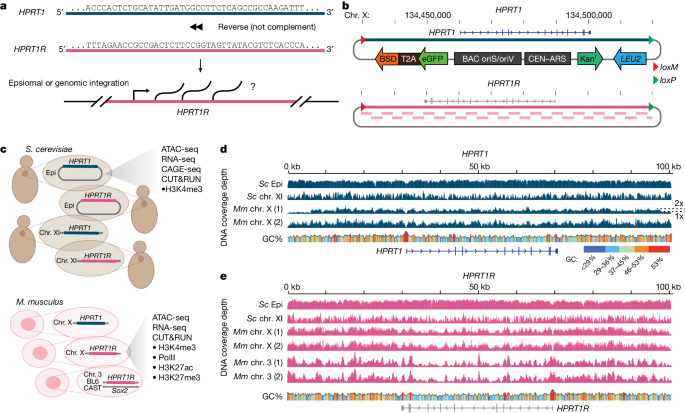
광범위한 전사 활동이 다양한 종에서 관찰됩니다. 현존하는 생물체의 게놈은 수십억 년에 걸친 진화를 겪었으며, 이러한 게놈 활동이 선택의 결과인지 아니면 ‘잡음’인지 분명하지 않습니다. 기본 게놈 상태를 특성화하는 것은 광범위한 전사 활동이 생물학적 의미를 가지는지 이해하는 데 도움이 될 수 있습니다. 여기서 우리는 Saccharomyces cerevisiae와 Mus musculus의 게놈에 합성 101-kb 위치를 도입하고 게놈 활동을 특성화함으로써 이 질문에 접근했습니다. 위치는 인간 HPRT1과 그 주변 영역을 역전시키되 보완하지 않음으로써 자연 순서의 기본 특성은 유지하되 진화된 코딩 또는 조절 정보를 없앴습니다. 우리는 진화된 효모 프로모터가 없음에도 불구하고 효모에서 역전된 HPRT1 위치와 원래 위치의 광범위한 활동을 관찰했습니다. 반면에, 역전된 위치는 마우스 배아 줄기세포에서 전혀 활동하지 않았으며 대신 억제적인 크로마틴 특징을 보였습니다. CpG 다이뉴클레오타이드가 없는 위치 변이체에서는 억제적 특징이 완화되었지만, 이 변이체 또한 전사적으로 비활성이었습니다. 이러한 결과는 코딩 정보가 없는 합성 게놈 시퀀스가 효모에서는 활성화되지만 마우스 배아 줄기세포에서는 비활성화된다는 것을 보여주며, 이는 이 두 다양한 진핵 세포 유형 간의 ‘기본 게놈 상태’의 주요 차이와 광범위한 전사, 유전 정보의 수평적 전달 및 새로운 유전자의 탄생을 이해하는 데 시사하는 바가 큽니다.
