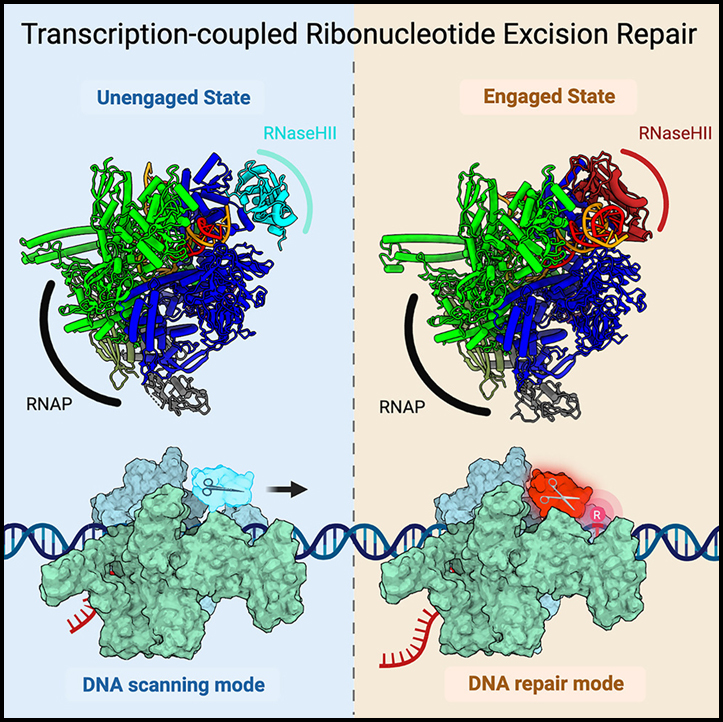
대장균에서 RNA 중합효소의 ribonucleotide 절제 DNA 복구 유도
Abstract
Ribonuclease HII (RNaseHII)는 게놈 DNA에서 잘못 통합된 ribonucleoside monophosphate (rNMP)를 제거하는 주요 효소입니다. 이 연구에서는 ribonucleotide 절제 복구 (RER)가 전사와 직접적으로 연결되어 있음을 보여주는 구조적, 생화학적 및 유전적 증거를 제시합니다.
Affinity pull-down 및 in cellulo에서 단백질 가교의 질량 분석 보조 매핑은 대장균에서 RNA polymerase (RNAP)와 상호 작용하는 대부분의 RNaseHII 분자를 나타냅니다. 신장 과정 동안 RNAP에 결합된 RNaseHII의 저온 전자 현미경 구조는 표적 rNMP 기질 유무에 관계없이 결합 및 결합되지 않은 상태에서 전사 결합 RER (TC-RER) 복합체를 정의하는 특정 단백질-단백질 상호작용을 보여줍니다.
RNAP-RNaseHII 상호 작용의 약화는 생체 내에서 RER을 손상시킵니다. 구조-기능 데이터는 RNaseHII가 RNAP를 “타고” 있는 동안 rNMP를 찾기 위해 1차원에서 DNA를 스캔하는 모델을 지원합니다. 우리는 또한 TC-RER이 복구 이벤트의 상당 부분을 차지하므로 RNAP를 가장 자주 발생하는 복제 오류를 감지하기 위한 감시 “수단”으로 설정한다는 것을 입증합니다.
Figure
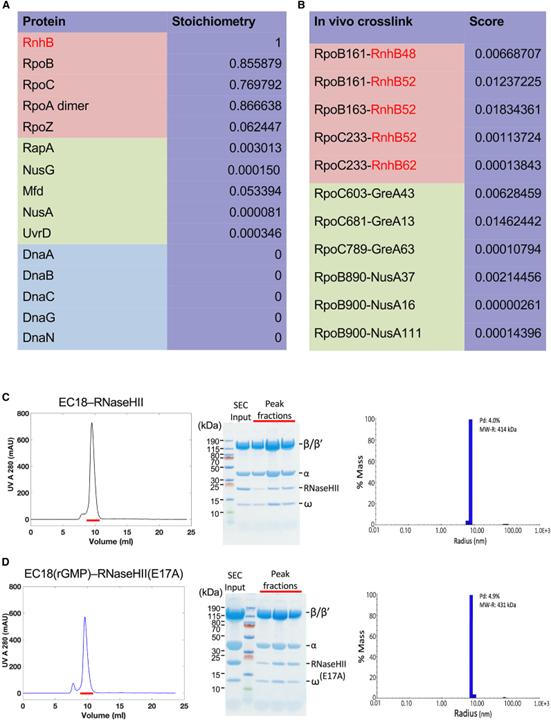
[Figure 1] TC-RER 복합체의 생체 내 발견 및 시험관 내 어셈블리
(A) 생체 내 RNaseHII 관련 단백질의 정량적 질량 분석.
(B) 염색체 10xHis 태그가 지정된 RNAP pull-down의 XLMS에서 RNAP 소단위와 RNaseHII 사이의 생체 내 비 중복 단백질 간 교차 링크의 목록.
(C) 체외에서 EC18-RNaseHII 복합체의 형성.
(D) 체외에서 EC18(rGMP)-RNaseHII(E17A) 복합체의 형성.
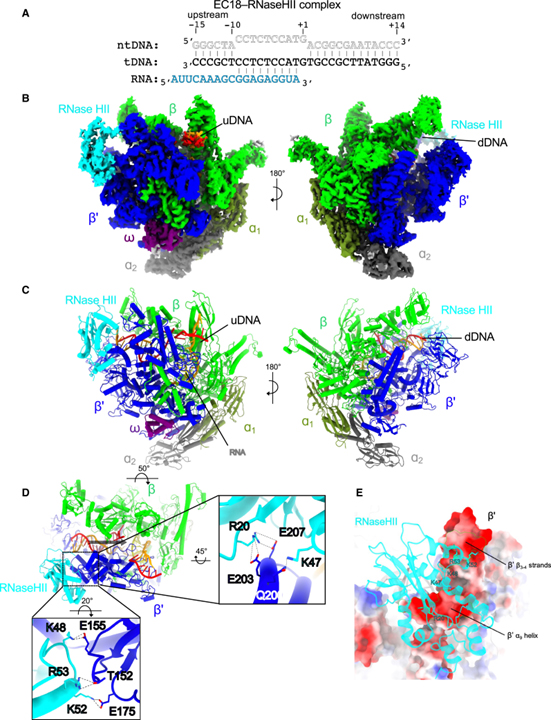
[Figure 2] cryo-EM 밀도 맵의 개요와 결합되지 않은 TC-RER 복합체의 구조 모델
(A) 3차 신장 복합체 (EC18)를 조립하는 데 사용되는 뉴클레오타이드 스캐폴드의 개략도.
(B) 2.96Å 공칭 해상도에서 EC18-RNaseHII 복합체의 cryo-EM 밀도 맵.
(C) EC18-RNaseHII의 구조 모델.
(D) RNaseHII와 EC18의 주요 단백질-단백질 상호작용.
(E) RNaseHII와 RNAP β’ 소단위 사이의 광범위한 인터페이스.
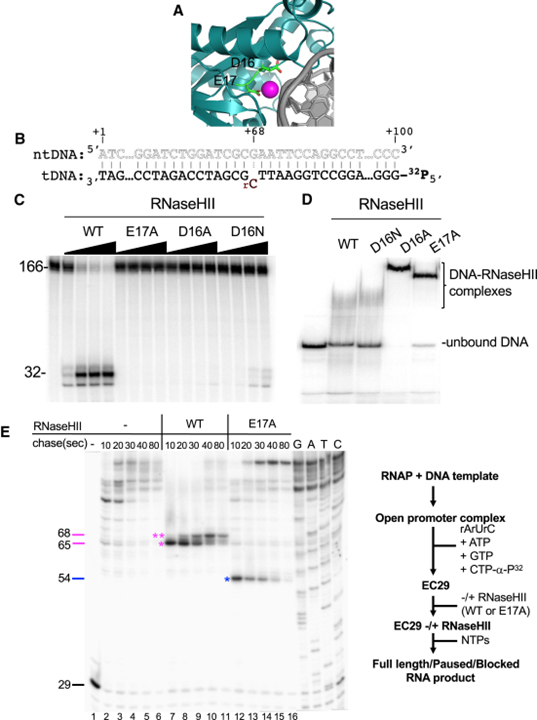
[Figure 3] RNaseHII 돌연변이 유발 및 효소 활성 테스트
(A) I-TASSER의 COFACTOR 모듈에 의해 생성된 DNA 및 촉매 마그네슘 이온에 결합된 대장균 RNaseHII의 상동성 모델.
(B) 체외 전사 분석에 사용되는 DNA template의 전사된 부분.
(C) WT 및 돌연변이 RNaseHII에 의한 기질 절단.
(D) WT 및 돌연변이 RNaseHII를 사용한 EMSA.
(E) RNAP 촉매 부위와 기질 결합 RNaseHII 사이의 거리 맵.
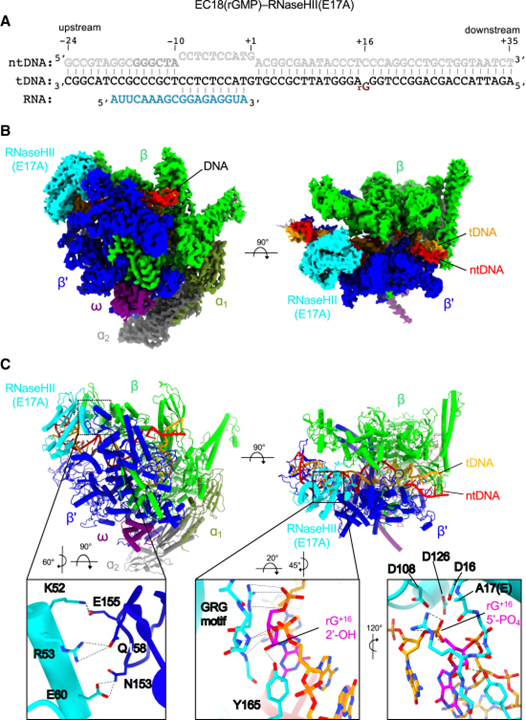
[Figure 4] cryo-EM 밀도 맵의 개요 및 결합된 TC-RER 복합체의 구조 모델
(A) 3차 신장 복합체-EC18(rGMP)을 조립하는 데 사용되는 뉴클레오타이드 스캐폴드의 개략도.
(B) 3.16Å 공칭 분해능에서 EC18(rGMP)-RNaseHII(E17A) 복합체의 cryo-EM 밀도 맵.
(C) EC18(rGMP)-RNaseHII(E17A)의 구조 모델.
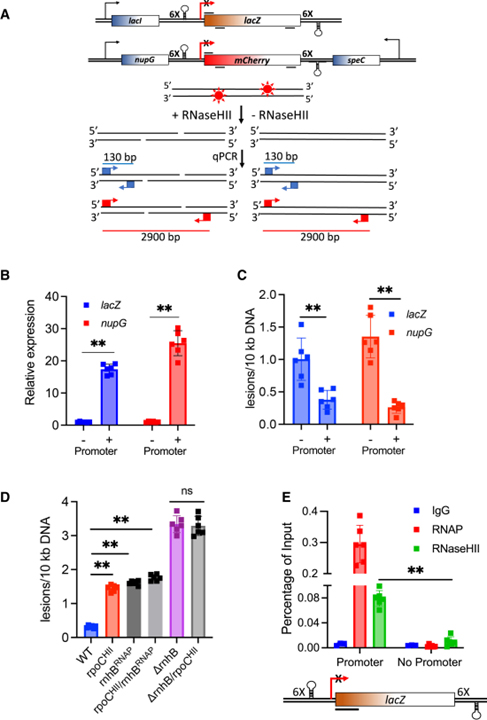
[Figure 5] 생체 내에서 RER과 전사의 결합
(A) 전사 절연체 모델.
(B) RT-qPCR에 의해 검출된 lacZ 및 nupG 절연체 내의 전사.
(C) SLR-qPCR 분석에 의해 검출된 바와 같이 절연체 내에서 rNMPs 복구.
(D) RNaseHII-RNAP 상호 작용을 약화시키는 RNAP와 RNaseHII의 rpoCHII 및 rnhBRNAP 돌연변이와 그 조합에 의한 로컬 RER의 손상.
(E) 로컬 전사에 따른 DNA에 대한 RNaseHII의 모집 차이.
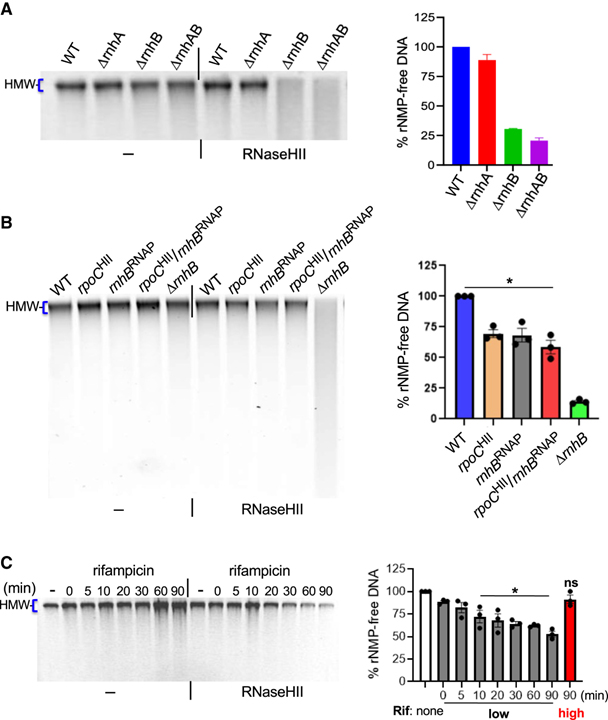
[Figure 6] 전사로부터의 부분적 분리에 의한 게놈 RER의 손상
(A) 글로벌 RER 분석.
(B) RNAP-RNaseHII 상호작용을 약화시키는 rpoCHII 및 mhBRNAP 돌연변이에 의한 게놈 RER 손상.
(C) Rifampicin을 사용한 RNAP에 의한 프로모터 탈출의 억제를 통한 전체 RER의 억제.
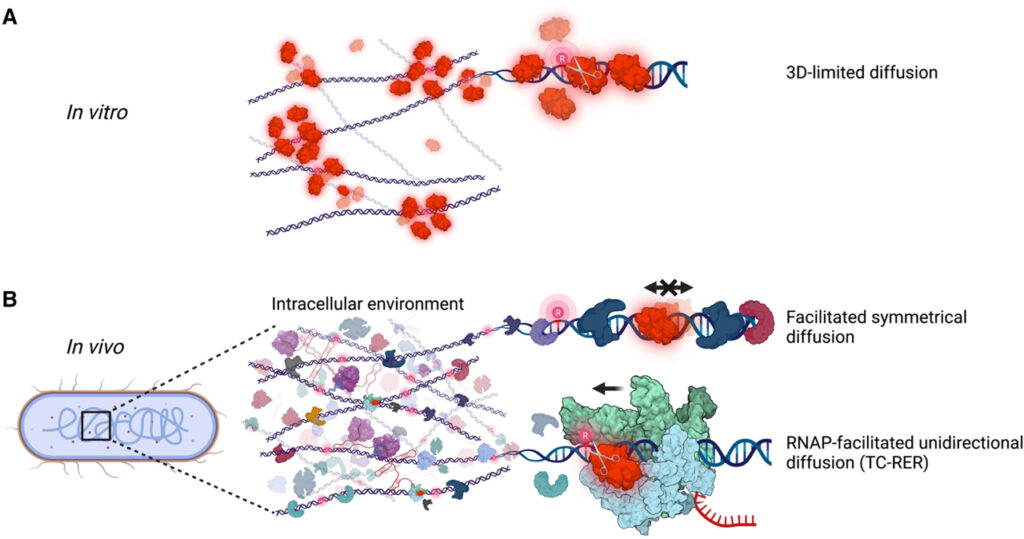
[Figure 7] RNA 중합효소의 게놈 차원의 RER 구동
(A) 시험관 내의 희석된 버퍼 상태에서 RNaseHII 표적 검색 모델.
(B) 생체 내의 거대 분자 혼합 상태에서 RNaseHII 표적 검색 모델.
Disscussion
전사 커플링 (TC)이 명확하게 확립된 유일한 DNA 복구 경로는 뉴클레오타이드 절제 복구 (NER)입니다. TC-NER에서 RNAP는 DNA 손상의 기본 센서, 병변 부위에 NER 효소를 전달하는 매개체, 기능적 NER 복합체의 적절한 조립을 위한 스캐폴드 역할을 합니다. 놀랍게도, 현재 결과는 RNAP가 RER에서 유사한 기능을 수행한다는 것을 보여줍니다. 기본 손상 센서는 RNaseHII 자체여야 하지만 RNAP는 주로 “전달 수단”으로 작동합니다. 실제로 RNAP는 DNA를 따라 RNaseHII의 1D 단방향 확산을 가능하게 하는 동시에 신속한 병변 인식 및 처리를 위해 DNA 기질에 대해 촉매 중심의 방향을 정확하게 지정합니다. 이러한 보조 확산은 RNaseHII의 낮은 풍부도, 염색체 rNMP의 높은 빈도, 거대분자 밀집, DNA 압축 및 수많은 DNA 결합 단백질을 포함하여 살아있는 세포에서 이 과정을 방해하는 고효율 RER과 여러 교란 요인 사이의 명백한 부조화를 해결하고 RNaseHII로부터 기판을 보호합니다.
NER에 의해 처리되는 것과 같은 부피가 큰 DNA 병변은 일반적으로 연장되는 RNAP에 탁월한 차단을 제공하며, 이는 NER 효소에 노출시키기 위해 UvrD 매개 RNAP 역추적을 필요로 합니다. 대조적으로 잘못 통합된 rNMP는 일시적으로 RNAP를 일시 중지하고 생체 내에서 광범위한 역추적을 유발할 가능성이 없습니다. 실제로 우리는 TC-RER이 역추적 단계를 통해 작동하지 않을 것이라고 주장하면서 RER에 대한 uvrD 결핍의 중요한 영향을 보지 않습니다. RNAP가 염색체 병변에 대한 기본 센서 역할을 하는 TC-NER와 달리 TC-RER은 RNAP를 스캐너가 아니라 “자동차”로 활용하여 잘못 통합된 rNMP에 대한 1D 검색을 용이하게 합니다. 역추적 기반 TC-NER는 rNMP 복구에도 기여할 수 있습니다. 그러나 이 과정에서 TC-NER의 역할은 2차적이어야 합니다. uvrA의 삭제는 활성 RNaseHII가 있는 경우 RER에 사실상 영향을 미치지 않기 때문입니다.
맞물린 TC-RER 복합체에서 RNaseHII의 형태는 주형 가닥에서 rNMP가 5′ O-P 결합을 끊을 수 있도록 합니다. 이러한 명백한 가닥 특이성은 박테리아 게놈의 모든 부분이 TC-RER이 두 가닥 모두에서 기능하도록 전사되어야 함을 의미합니다. 사실, 침투성 전사 현상은 박테리아에서 잘 확립되어 있습니다. 우리의 RNA-seq 분석은 기하급수적으로 성장하는 E. coli에서 일반적으로 낮은 효율성에도 불구하고 거의 모든 안티센스 및 유전자간 영역이 전사됨을 보여줌으로써 이 견해를 뒷받침합니다.
포유류 RNaseH2는 rNMP에 대한 1D 스캐닝을 가능하게 할 수 있는 복제 기계 및/또는 PCNA와 상호 작용하도록 제안되었습니다. 연구에서 사용된 생체 내 XLMS 분석은 RNaseHII와 박테리아 레플리솜 또는 PCNA paralog-DnaN 베타 클램프를 포함한 관련 요인 사이의 안정적이거나 특정한 상호 작용을 감지하지 못합니다. 박테리아의 RNaseHII는 단일 폴리펩타이드로서 활성인 반면 진핵생물에서는 3-서브유닛 효소로서 활성이라는 점에 유의해야 합니다. 따라서 이 소단위 구성에 따라 모집 및 상호 작용 파트너의 방법이 다를 수 있습니다. 박테리아 RNaseHII는 효율적인 RER을 달성하기 위해 전적으로 전사 결합에 의존하는 것으로 보입니다. 최근 인간 세포에서 RNaseH2와 RNAP II의 게놈 공동 위치화가 보고되었습니다. RNAPII 관련 RNaseH2의 역할이 공동 전사 R-루프를 감소시키는 것으로 제안되었지만 TC-RER에서도 기능할 수 있다고 추측하고 있습니다. 이러한 포유류 TC-RER은 최근 암과 관련이 있는 과정인 ribonucleotide를 포함하는 전사 관련 돌연변이 유발을 방지할 수 있습니다.