대장암에서 게놈과 후생유전자의 공진화
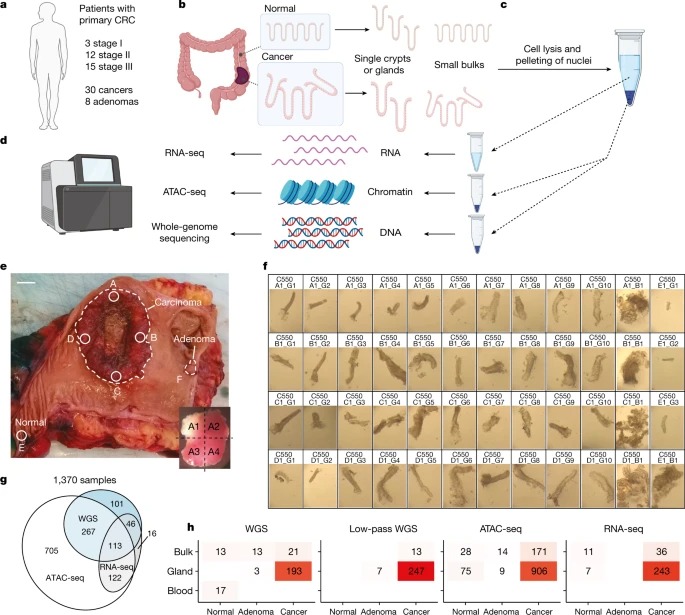
대장암은 암과 관련된 사망의 주요 원인이며 광범위한 게놈 연구를 거쳤다. 그러나 DNA 돌연변이만으로는 악성 형질전환을 완전히 설명할 수 없다. 여기서는 개별 분비선의 spatial multi-omic profiling을 사용하여 단일 클론 분해능에서 대장종양의 게놈과 후생유전자의 공진화를 조사한다. 우리는 30개의 primary 암과 8개의 부수적인 adenoma에서 1,370개의 샘플을 수집했고 1,207개의 크로마틴 접근성 프로파일, 527개의 전체 게놈 및 297개의 전체 transcriptome를 생성했다. 우리는 chromatin modifier gene에서 DNA 돌연변이에 대한 긍정적인 선택과 유전자 돌연변이가 없는 암 유발 유전자의 조절 영역을 포함하여 반복되는 체세포 염색질 접근성 변화를 발견했다. transcription factor 결합에 대한 접근성에 대한 게놈 전반의 변화는 CTCF, 인터페론의 조절 감소, SOX 및 HOX transcription factor 패밀리의 접근성 증가를 포함했으며, tumourigenesis 동안 발달 유전자의 개입을 시사했다. 체세포 염색질 접근성 변화는 유전될 수 있으며 adenoma와 암을 구별한다. Mutational signature analysis는 epigenome가 DNA 돌연변이의 축적에 영향을 미친다는 것을 보여주었다. 이 연구는 대장암 생물학을 이해하는 데 근본적인 영향을 가진 유전적, 후성적 tumour heterogeneity의 지도를 제공한다.
