ωRNA 및 target DNA와 복합체의 OMEGA nickase IsrB의 구조
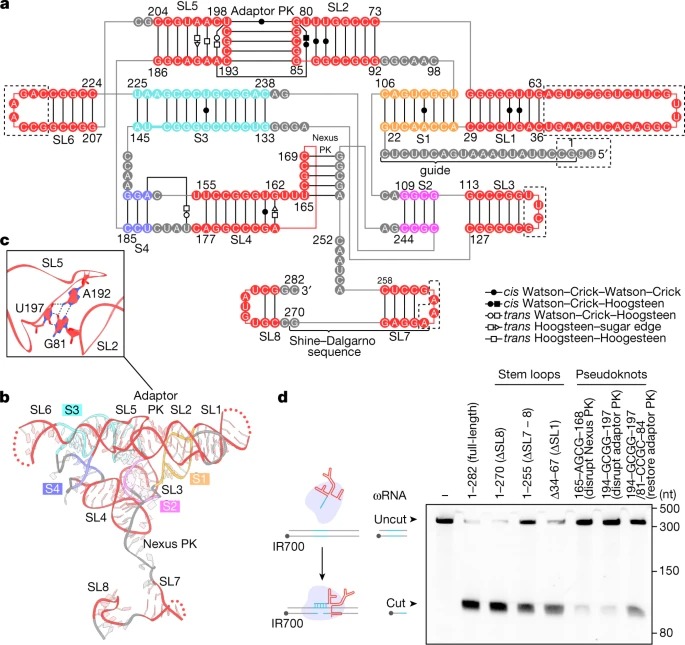
CRISPR-Cas와 같은 RNA-guided 시스템은 프로그램 가능한 substrate 인식과 enzymatic 기능을 결합하는데, 이는 강력한 molecular technology를 개발하는 데 유리하게 사용되어 왔다. 이러한 시스템에 대한 구조적 연구는 RNA와 단백질이 어떻게 그들의 substrate를 공동으로 인식하고 절단하는지 밝혀주었고, 추가적인 기술 개발을 위한 합리적인 공학을 안내했다. 최근의 연구는 Cas9의 조상인 IscB와 HNH nuclease 도메인이 없는 IscB의 homologue인 nickase IsrB를 포함하는 새로운 종류의 RNA-guided 시스템을 식별했다. IsrB는 약 350개의 아미노산으로 구성되어 있지만, 작은 크기는 비교적 큰 RNA 가이드(약 300-ntΩRNA)에 의해 균형을 이룬다. 여기서, 우리는 cognate ωRNA 및 표적 DNA와 복합적인 Desulfovirgula thermocuniculi IsrB(DtIsrB)의 극저온 전자 현미경 구조를 보고합니다. 우리는 IsrB 단백질의 전체적인 구조가 Cas9과 공통의 발판을 공유한다는 것을 발견했다. 그러나 표적 선택을 용이하게 하기 위해 인식(REC) 로브를 사용하는 Cas9와 달리, IsrB는 ωRNA에 의존하며, ωRNA의 일부는 REC와 유사한 위치에 있는 복잡한 ternary structure를 형성한다. 다른 RNA-guided 시스템과의 비교뿐만 아니라 IsrB와 그 ωRNA의 구조 분석은 단백질과 RNA 사이의 기능적 상호작용을 강조하여 이러한 다양한 시스템의 생물학과 진화에 대한 이해를 증진시킨다.