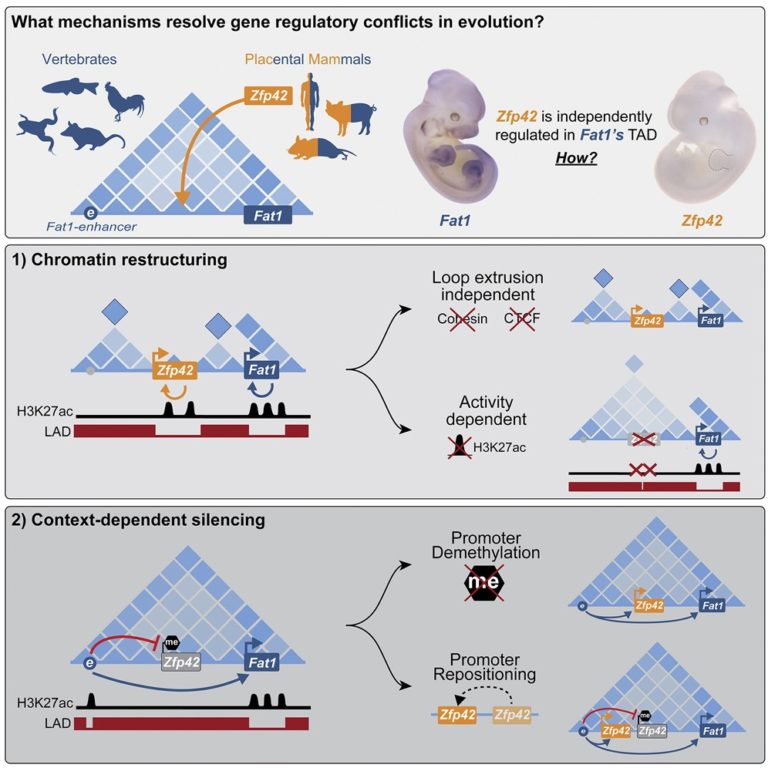
Repression과 3D-restructuring을 통해 진화적으로 재배치된 게놈의 regulatory conflict을 해결한다.
Abstract
Regulatory 환경은 복잡한 발달 유전자 발현을 유도하지만, 진화 과정에서 새로운 유전자와 기능을 통합할 때 그 integrity가 어떻게 유지되는지 불분명하다.
여기서는 태반 포유동물 특이 유전자 Zfp42가 그 유전자 Fat1의 보존된 발현을 채택하거나 방해하지 않고 고대 척추동물 위상적으로 연관된 도메인(TAD)에서 어떻게 나타났는지 조사했다. ESC에서 물리적 TAD partitioning은 Zfp42와 Fat1을 독립적인 발현을 구동하는 뚜렷한 local enhancers로 분리한다. 이러한 분리는 CTCF/cohesin이 아닌 chromatin activity에 의해 이루어진다. 반대로, embryonic limb에서는 inactive Zfp42가 active Fat1 enhancers에 반응하지 않고 Fat1의 온전한 TAD를 공유한다. 그러나 Fat1 enhancer-incompatibility 또는 nuclear envelope-attachment은 Zfp42의 무반응을 설명하지 않는다. 오히려 Zfp42의 프로모터는 context-dependent DNA methylation에 의해 enhancers에 대해 불활성화된다.
따라서 다양한 메커니즘으로 Fat1 locus에 독립적인 Zfp42 조절을 통합할 수 있었다. 결정적으로, 이러한 regulatory complexity는 게놈 전체에 걸쳐 대부분의 TAD가 독립적으로 발현되는 여러 유전자를 포함하고 있기 때문에 진화에서 공통적으로 나타난다.
Figure
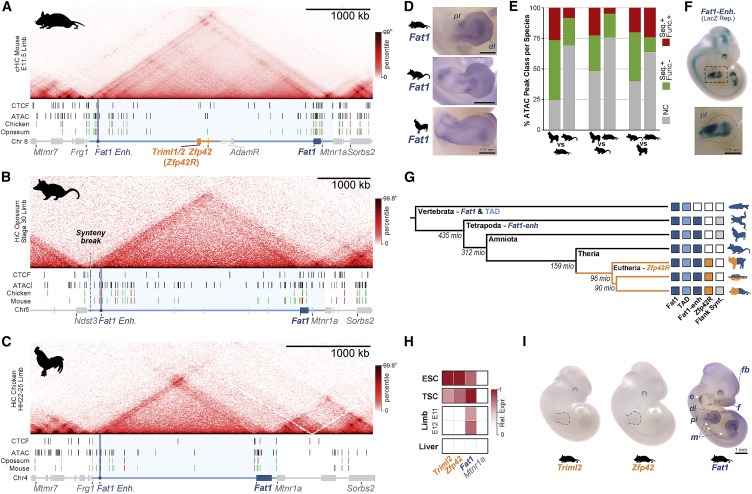
Figure 1. Zfp42R gene은 Fat1의 고대 TAD regulatory 환경에서 다양한 발현으로 나타난다.
(A–C) (A) 쥐, (B) 주머니 쥐, (C) 닭의 cHi-C or Hi-C(genomic analysis technique)와 아래 ATAC-seq and CTCF ChIP-seq 피크가 있는 embryonic limb buds.
(D) embryonic limbs에서 species-specific Fat1 WISH (whole mount in situ hybridization). n = 2–4 스케일 바, 0.5mm
(E) species ATAC-seq peaks의 pairwise conservation quantification.
(F) E11.5 embryo에서 mouse Fat1-enh의 LacZ reporter assay, n = 4 embryos.
(G) Fat1, the TAD, Fat1-enh, Zfp42R, or flanking synteny outside the TAD가 표시된 Phylogenetic tree.
(H) Fantom5 CAGE expression의 Gene activity overview.
(I) WISH의 Gene activity overview.
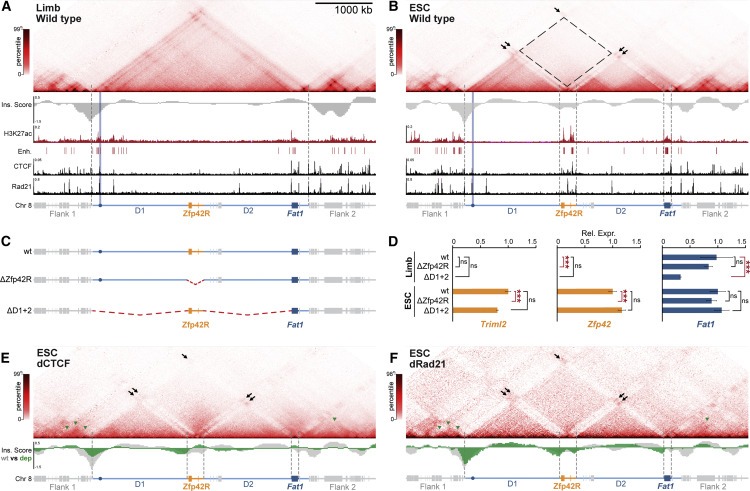
Figure 2. Fat1 및 Zfp42는 ESC에서 분리되고 재구성된 도메인에서 local enhancer를 독립적으로 활용한다.
(A and B) (A) E11.5 limb buds, (B) ESCs의 cHi-C와 아래 H3K27ac, CTCF & Rad21 ChIP-seq에 대한 와 Ins. score(insulation score). E11.5 limb cHi-C은 figure1에서 재현된다.
(C and D) RNA-seq(D)에 의해 분석된 gene expression effect를 갖는 (C) deletion mutants의 개략도.
(E and F) (E) dCTCF 또는 (F) dRad21 ESCs의 cHi-C와 wild type (gray) 또는 depletion (green) Ins. Scores.
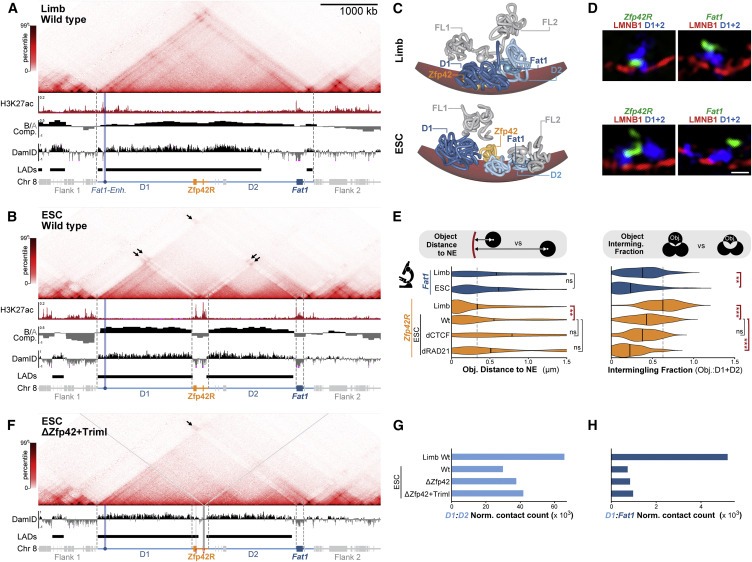
Figure 3. The Zfp42/Fat1 TAD는 limb에서 다양한 chromatin 환경을 수용하지만 ESC의 discrete compartments로 재구성됩니다.
(A and B) (A) E11.5 limb buds (B) ESCs의 cHi-C와 아래 H3K27ac-ChIP-seq, compartments, and Lamin B1 DamID tracks (LADs).
(C) E11.5 limbs (top) and ESCs (bottom) 안의 simulated NE (red)가 있는 locus의 대표적인 polymer model.
(D) Lamin B1 (red), D1+D2 (blue) and Zfp42R or Fat1 (green)이 있는 대표적인 immune FISH Z-slice. Scale bar, 500 nm.
(E) wild-type limb or wild-type, CTCF-depleted (dCTCF), and Rad21-depleted (dRad21) ESCs의 FISH measurements.
(F) deleted H3K27ac regions을 강조하는 회색 선이 있는 ΔZfp42+Triml ESCs의 cHi-C와 Lamin B1 DamID.
(G and H) (G) D1:D2 and (H) D1:Fat1 cHi-C interactions의 정량화.
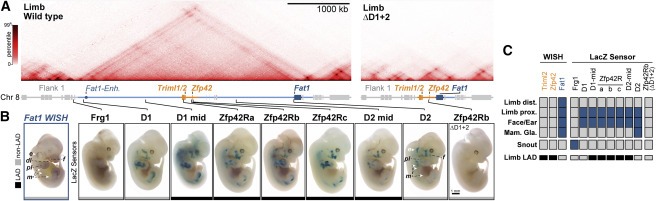
Figure 4. NE attachment는 Zfp42R gene activation이나 Fat1 enhancer와의 communication을 막지 않는다.
(A) wild-type and ΔD1+2 E11.5 limb buds의 Hi-C.
(B) E12.5 embryos에서 endogenous Fat1 (WISH, left) 또는 integrated β-globin LacZ sensors (LacZ, right)의 staining n = 4–10 embryos.
(C) LAD-status가 표시된 gene, enhancer, and sensor activity의 요약
Discussion
TAD는 종종 도메인의 경계 내에서 발견되는 enhancer activity의 전달을 보장하는 안정적이고 보존된 structural scaffolds로 설명된다(Andrey and Mundlos, 2017). 이 간단한 모델에서 유사한 기능을 가진 유전자는 공유 TAD에서 함께 제어될 수 있는 반면, divergent regulation이 필요한 유전자는 분리된 도메인에 단독으로 배치되어야 한다(Wu et al., 2021). 그러나 우리는 게놈의 대부분의 TAD가, 특정 위치(ex. the essential Hox genes)에 대한 이전 연구에서 볼 수 있듯이 독립적으로 조절된 여러 발달 유전자를 포함하고 있다는 것을 발견했다(Andrey et al., 2013; Huang et al., 2017; Noordermeer et al., 2011; Palstra et al., 2003; Soshnikova and Duboule, 2009). 결과적으로, 간단한 TAD 조절 모델만으로는 게놈 진화를 설명할 수 없다. 대신, 우리는 기존의 Fat1 landscape와 보고된 다양한 생리학적 기능을 방해하지 않고 Zfp42R 유전자 조절을 통합한 여러 메커니즘을 보여준다(그림 6E-6G; Peng et al., 2021). 이와 같이, single loci라도 진화에 충돌하는 규제 프로그램을 쉽게 통합할 수 있으며, 이 용량은 게놈이 엄청난 규제 복잡성과 유연성을 갖추게 한다. 인간 유전학에서, 이것은 또한 왜 섞인 TAD에서 새로운 enhancer-promoter 조합을 생성하는 많은 게놈 재배열이 gene misexpression이나 질병을 유발하지 않는지 설명할 가능성이 있다(Despang et al., 2019; Laugsch et al., 2019). 특히, 3D를 포함한 다른 mechanisms restructuring 및 silencing dependent context가 규제 환경에서 enhancer usage를 개선하기 때문입니다.
우리는 진화적으로 안정적인 TAD도 특정 세포 유형의 transcription를 조절하기 위해 대규모로 재구성될 수 있음을 보여준다. 그러나 이전 예와 달리, 우리는 이것이 underlying chromatin activity에 의해 예기치 않게 cohesin 및 CTCF와 독립적으로 구동된다는 것을 보여준다(Bonev et al., 2017; Isoda et al., 2017). 구체적으로, ESC에서 Zfp42 및 Fat1 영역의 activity은 active and inactive chromatin의 antagonistic 경향을 공간적으로 분리하도록 유도한다. 이와 같이, 두 유전자는 분리된 도메인에서 독립적인 enhancer와 함께 분리된다. 흥미롭게도, 다른 위치는 Dppa2/4를 포함한 더 큰 기존 TAD 내에서 유사한 활동 의존적 고립을 보이는 것으로 보고되었으며, 이는 태반 포유류의 기존 영역에서도 나타났다(그림 S6G-S6I; Sima et al., 2019). 따라서, TAD는 loop extrusion뿐만 아니라 compartmentalization에 의해 구조화되며, 이는 진화적 적응을 위해 변경될 수 있다. 그러나 이는 chromatin structure와 underlying epigenetic state가 상호 구동되고 transcription을 제어하기 위해 서로 self-reinforce 될 수 있다는 흥미로운 일반화 가능성을 더욱 제기한다.
우리는 LAD에서 NE의 chromatin positioning이 endogenous gene expression을 조절하는 결정론적 역할을 할 필요가 없다는 것을 입증한다. 몇몇 그룹은 이전에 in vitro에서 LADs로 또는 LAD로부터 ectopically transplanted promoters를 이식했으며 NE가 일반적으로 transcriptionally repressive environment임을 밝혔다(Finlan et al., 2008; Leemans et al., 2019; Reddy et al., 2008). 따라서 NE에 특정 유전자를 보유하는 것은 in vitro differentiation 동안 inactivity를 유지하는 것으로 보고되었다(Poleshko et al., 2017; Robson et al., 2016). 그러나, 우리는 Fat1 enhancer가 LADs와 그것들을 분리하는 광범위한 intervening lamina-association에도 불구하고 LacZ reporter gene를 여전히 활성화할 수 있다는 것을 발견했다. 따라서, LAD는 (1) 유전자를 silence시키기에 충분하지 않고 (2) enhancer-promoter communication의 효과적인 insulator도 아니다. 대신에, LAD는 필요할 때 유전자가 국소적으로 탈출하고 활성화될 수 있도록 쉽게 재구성될 수 있다(Brueckner et al., 2020; Therizols et al., 2014). 이와 같이, LAD는 유전자 조절이나 진화에서 새로운 gene activity의 출현을 위한 완전히 불친절한 환경은 아니다.
대신, 우리는 multi-gene TAD의 극단적인 expression 차이가 특정 enhancer와의 비호환성보다는 프로모터의 정확한 endogenous position 및 sequence-context에 의해 주도될 수 있다는 것을 발견했다. 특히, Triml 1/2 및 Zfp42 프로모터는 TAD의 embryonic Fat1 enhancer에 의해 활성화될 수 있지만, endogenous position에서 떨어져 재배치될 때만 활성화될 수 있다. 이는 enhancer- promoter 양립성이 포유류의 transcription을 조절하는 정도가 논란의 여지가 남아 있고 episomal in vitro assay에서 native contexts 밖에서 주로 조사되기 때문에 중요하다(버그만 외, 2021; 마르티네즈-아라 외, 2022; 레이-존스와 스피바코프, 2021; 반 아렌스베르겐 외, 2014). 결과적으로, 그러한 접근법은 native regulatory landscapes에서 발견되는 많은 endogenous gene expression 결과를 예측하지 못할 수 있다(Arnold et al., 2017; Martinez-Aar et al., 2022).
마지막으로, 우리는 DNA 메틸화가 context-dependent silencing을 유도할 수 있지만 promoter sequence에 의한 유전자만을 대상으로 하는 것이 아니라는 것을 발견했다. 대신에, 우리는 프로모터 DNA 메틸화가 genomic position와 아마도 인접한 flanking DNA의 sequence에 의존할 수 있다는 것을 발견했다. 따라서 우리는 specific chromatin modification의 absence/presence를 넘어 DNA 메틸화를 목표로 하는 특징을 확장한다(Oui 등, 2007; Weinberg 등, 2021; Zhang 등, 2010). 그러나, 그렇게 함으로써, 우리는 또한 DNA 메틸화가 promoter’s intrinsic transcriptional activity를 억제하는 것이 아니라 enhancer에 대한 sensitivity를 억제하는 추가적인 방법을 보여준다. 중요한 것은, 이는 early embryo에서 DNA 메틸화가 완전히 제거되었을 때 왜 minor gene expression defect만 관찰되는지를 설명할 수 있다(Grosswendt et al., 2020; Yagi et al., 2020). 이러한 관점에서, misexpression은 공유된 환경 내에서 enhancer에 노출된 비메틸화 유전자로 제한될 것이며, 심지어 그 enhancer가 활성화된 특정 세포 유형에서만 제한될 것이다. 그러나, 우리는 모든 세포 유형에서 DNA 메틸화와 survey transcription을 완전히 제거할 수 없는 우리의 무능함이 이 규정의 전체 범위를 테스트하는 것을 방해한다는 것에 주목한다. 그럼에도 불구하고, DNA 메틸화는 다중 유전자 TAD 내에서 난잡한 enhancer 사용을 정제하는 것으로 알려진 억제 메커니즘을 추가한다(Gjalta et al., 2021; Noordermeer et al., 2011; Soshnikova and Duboule, 2009).